Molclus:简便实用的团簇构型和分子构象搜索程序
最新版本:1.13 (2025-Dec-11发布)
本页目录
下载 引用方式 简介 使用说明 使用咨询 更新历史
Molclus辅助工具 使用Molclus已发表的学术文章
下载:
最新版Molclus程序包的可执行文件可在此免费下载:Windows版,Linux 64bit版。没有Mac版。源代码不公开提供。
引用方式:
如果Molclus(无论是其中哪个组件)在你的文章中被使用,都必须在正文进行引用!引用格式为:
Tian Lu, Molclus program, Version x.x, http://www.keinsci.com/research/molclus.html (accessed 月 日, 年)
x.x代表实际版本号。具体格式可根据期刊要求修改。如果没有按要求引用,一经发现将被列入黑名单,禁止在未来使用Molclus,并且会在本页面里进行公示!
另:我们未来会专门发表论文对Molclus进行介绍,届时会在本页面提供文章信息,之后请引用论文而不要再引用网址,目前发表文章必须按照上述说明引用。
如果在Molclus中调用了Shermo程序计算了热力学量,也务必同时按照Shermo程序主页(http://sobereva.com/soft/shermo)上的说明恰当引用Shermo程序原文。
简介:
Molclus是一款团簇构型搜索和分子构象搜索的程序,由卢天(北京科音自然科学研究中心)开发。优点有:
- 完全免费:对用户分文不取,以后也不会收费。有任何使用问题都可以在计算化学公社论坛上发帖求助,开发者会迅速提供义务解答
- 普适:原理上可以用于包含任意元素、任意原子数的原子/分子团簇的构型搜索和分子构象搜索
- 简便、友好:使用步骤极尽简单,无需安装,输出信息简明易懂。只要有最基本的计算化学常识和正常的理解能力,阅读相关介绍和实例文章后很快就可以上手计算、获得需要的数据
- 灵活:程序设计得非常灵活、流程十分透明,用户可以控制构型/构象搜索的全部细节,对数据修改、删减、拼接、续算、复算等都极其方便。每个环节涉及的结构都可以用免费、强大的可视化程序VMD方便地观看
- 精度随意可控:用户可以根据对精度和耗时的要求以及体系的特点选择最恰当的方式执行搜索。是愿意付出较高耗时来获得较精确的结果,还是想以较低的耗时来获得相对粗糙的结果,都完全可以由用户自行决定
由于上述优点,稍微熟悉Molclus后会觉得比用很多动辄几千甚至上万元的昂贵的商业程序做构型、构象搜索好用得多,不仅精度高得多得多,也明显更灵活和方便。而那些收费昂贵且只支持分子力场进行构象搜索的商业程序其实根本没什么实际用处,又不灵活,精度又实在太差。
Molclus已经被大量研究者们使用并在Nature、Nature Catalysis/Commun.、JACS Au、Adv. Funct. Mater.、Sci. Adv.、Chem、Carbon、Chem. Eng. J.、Chem. Eur. J.、J. Mater. Chem.等高水平的刊物上发表了优秀的文章,使用Molclus已发表的文章列表请点击此处查看。
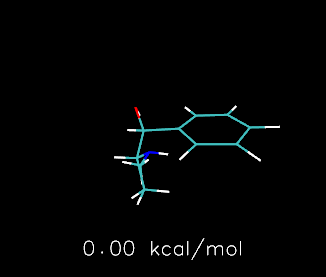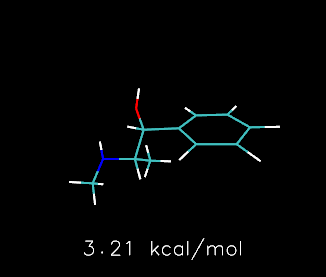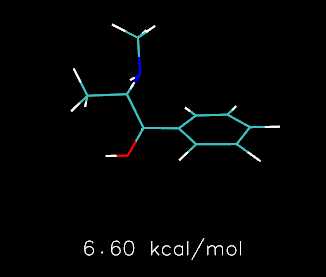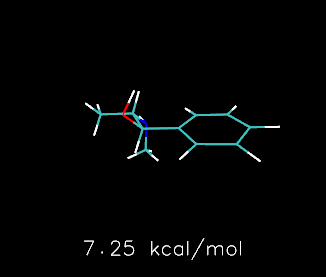
下图是利用Molclus对(H2O)4团簇和麻黄碱进行构型/构象搜索的结果的例子,由免费的VMD程序载入molclus的结果文件绘制
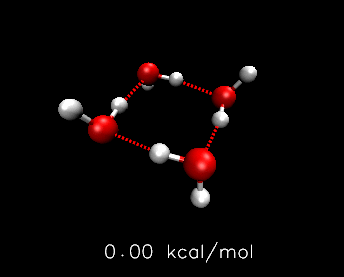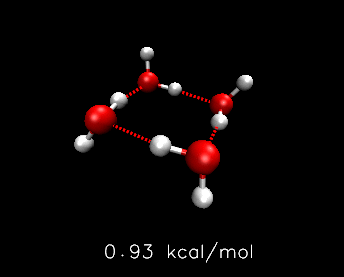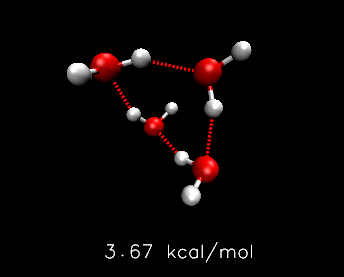
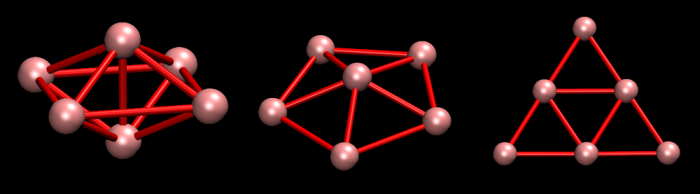
下图是Molclus搜索出的Li6团簇的三种构型
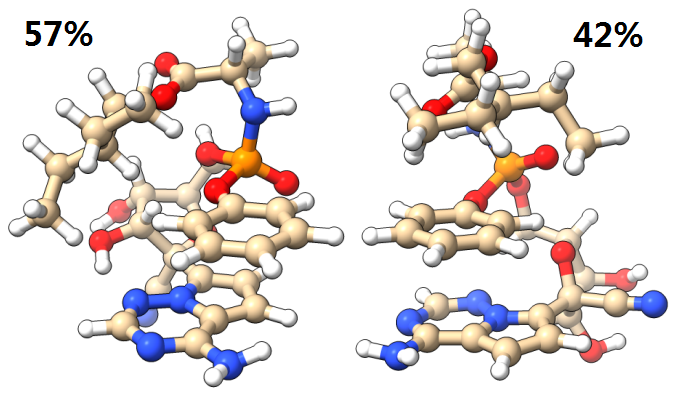
下图是Molclus搜索出的治疗2019年12月开始蔓延的COVID-19肺炎的特效药瑞德西韦(Remdesivir)在水环境下常温时自由能最低的两种构象和Boltzmann分布比例,图像由ChimeraX绘制
使用说明:
极其详细、易懂的Molclus的使用介绍与实例(这个就是现阶段的手册):
- 《使用molclus程序做团簇构型搜索和分子构象搜索》(http://bbs.keinsci.com/thread-577-1-1.html)
此文介绍了molclus的基本知识,并且给出将经典分子动力学程序与molclus结合做分子团簇构型搜索和构象搜索的例子。molclus的用户首先要看此文! - 《genmer:生成团簇初始构型的超便捷工具》(http://bbs.keinsci.com/thread-2369-1-1.html)
此文介绍了molclus中的genmer组件的用法,并且给了genmer结合molclus实现原子和分子团簇构型搜索的实例。 - 《gentor:扫描方式做分子构象搜索的便捷工具》(http://bbs.keinsci.com/thread-2388-1-1.html)
此文介绍了molclus中的gentor组件的用法,并且给了gentor结合molclus实现分子构象搜索的实例。 - 《使用Molclus结合xtb做的动力学模拟对瑞德西韦(Remdesivir)做构象搜索》(http://bbs.keinsci.com/thread-16255-1-1.html)
此文通过一个实例,介绍如何将xtb程序跑的分子动力学轨迹作为初始输入结构,通过molclus寻找水环境中能量最低构象。此文提出的构象搜索流程具有极高的普适性,对于含有10个以上可旋转键的情况比用gentor的做法更适合。 - 《将Confab或Frog2与Molclus联用对有机体系做构象搜索》(http://bbs.keinsci.com/thread-20063-1-1.html)
此文介绍了如何将专门的构象生成程序Confab或Frog2与molclus结合,方便、傻瓜化地对构象空间很大的柔性有机体系做构象搜索 - 《将多帧xyz文件转化成量子化学输入文件的工具:xyz2QC》(http://bbs.keinsci.com/thread-12468-1-1.html)
打算在超算等特殊环境使用molclus者建议阅读此文,介绍了molclus中的xyz2QC组件的用处和用法。
《Molclus FAQ》(http://sobereva.com/730) 是很重要的文章,也要注意看。
有molclus用户针对在集群上通过PBS作业提交方式使用molclus的方法做了介绍,见:http://bbs.keinsci.com/thread-15598-1-1.html。
基本使用流程:
记录初始结构的traj.xyz文件既可以通过molclus自带的产生初始团簇构型的genmer工具或产生分子初始构象的gentor工具生成,也可以通过GROMACS、xtb等程序跑分子动力学或者做模拟退火产生,也可以用专门的构象生成程序如Confab产生。然后molclus会对traj.xyz记录的所有或部分结构自动调用收费的Gaussian或免费的ORCA、MOPAC、xtb、Open Babel进行优化或单点或振动分析计算,并对结果通过自带的isostat工具进行去重和按照能量排序。
使用基本流程图如下。青色方框是molclus自身的组件,椭圆包围的文件名是运行过程中涉及的文件
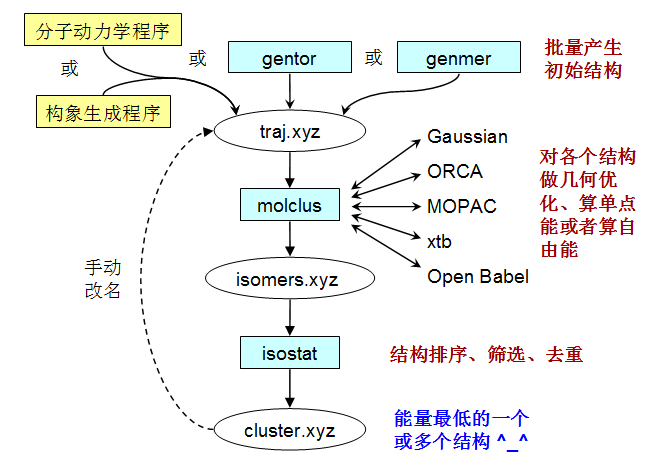
Molclus通常有以下使用方式,各有长处,可根据实际情况决定最佳组合:
- Molclus自带的genmer程序结合Molclus:用于分子团簇或原子团簇构型搜索,使用简便。对于分子团簇搜索这通常是首选。唯一局限性是对分子的构象考虑不够充分,因为genmer将分子当成刚性来堆积,因此不适合单分子柔性很强的情况(这种情况适合后面说的做动力学程序结合molclus)。相关介绍和示例看《genmer:生成团簇初始构型结合molclus做团簇结构搜索的超便捷工具》(http://bbs.keinsci.com/thread-2369-1-1.html)
- Molclus自带的gentor程序结合Molclus:用于分子构象搜索。使用简便,但不支持环状区域构象搜索(环状区域是指诸如环己烷这样有多种构象的环状区域)。对于可旋转的键不很多的分子的构象搜索这是首选,可控性最强。相关介绍和示例看《gentor:扫描方式做分子构象搜索的便捷工具》(http://bbs.keinsci.com/thread-2388-1-1.html)
- 第三方的Confab或Frog2构象生成程序结合Molclus:用于分子构象搜索。使用最为傻瓜化,但不支持环状区域构象搜索、不支持普通有机分子以外的情况。相关介绍和示例看《将Confab或Frog2与Molclus联用对有机体系做构象搜索》http://bbs.keinsci.com/thread-20063-1-1.html)
- xtb程序跑动力学轨迹结合Molclus:普适性极强,用于构象搜索、原子/分子团簇构型搜索皆可,使用容易。但由于模拟时间长度有限,有动力学采样不充分导致遗漏构型/构象的风险(风险随动力学模拟时间增长而减小)。相关介绍和示例看《使用Molclus结合xtb做的动力学模拟对瑞德西韦(Remdesivir)做构象搜索》(http://bbs.keinsci.com/thread-16255-1-1.html)
- GROMACS等经典力场分子动力学程序结合Molclus:用于分子团簇搜索和分子构象搜索。使用稍繁琐,因为得创建拓扑文件,且被动力学模拟的体系必须有恰当的力场,好处是动力学模拟耗时极低,因此动力学采样不充分导致遗漏构型/构象的风险很小。相关介绍和示例看《使用molclus程序做团簇构型搜索和分子构象搜索》(http://bbs.keinsci.com/thread-577-1-1.html)。不了解GROMACS使用的话推荐通过《北京科音分子动力学与GROMACS培训班》(http://www.keinsci.com/KGMX)学习相关使用知识。
Molclus可以调用的用于计算能量、优化的程序有以下程序。完全覆盖小体系高精度计算和巨型体系粗放计算
- Gaussian(收费):DFT、后HF、半经验、分子力场(UFF等)。安装方法
- ORCA(免费):DFT、后HF、半经验。安装方法
- MOPAC(免费):半经验。安装方法
- xtb(免费):GFN-xTB(半经验形式的DFT)、GFN-FF力场。简介
- OpenBabel(免费):分子力场(MMFF94等)。简介
使用咨询:
在计算化学公社论坛(http://bbs.keinsci.com)的量子化学版发帖咨询,发帖时请选择"Molclus"分类。如果发现程序里有bug也非常欢迎发帖报告。也可以在思想家公社QQ群里提问,群号见http://sobereva.com/QQrule.html。
更新历史
2025-Dec-11:1.13版发布
• 利用template_SP.gjf自动调用Gaussian算高精度单点的情况,现在当ibkchk不为0要求备份chk时,chk文件会被备份为gauSP[结构序号].chk。利用template_SP.inp自动调用ORCA算高精度单点的情况,现在当ibkgbw不为0要求备份gbw时,gbw文件会被备份为orcaSP[结构序号].gbw。
2023-Aug-23:1.12版发布
• ibkout=-1也同时有了ibkout=1的效果。即ibkout=-1时如果有输出文件是被新算出来的,也会被备份。
2023-Aug-17:1.11版发布
• 改进了settings.ini里ibkout=-1时的处理:对于且当前目录下有template_SP.inp或template_SP.gjf而自动调用ORCA或Gaussian自动算单点时,如果发现当前目录下有以前备份好的相应文件(如orcaSP00002.out、gauSP00005.out),则Molclus会直接读里面的数据而不会重新计算。
2023-Jul-9:1.10版发布
• Molclus的settings.ini里ibkout新增-1选项。对于调用Gaussian或ORCA的情况,ibkout设为-1时如果发现当前目录下有以前备份好的文件(如gau00002.out、orca00005.out),则Molclus会直接读里面的数据而不会重新计算。
2022-Mar-6:1.9.9.9版发布
2022-Sep-9:最后更新
• Molclus判断traj.xyz中要忽略的结构时支持了对原子间距离更精细的控制。在settings.ini里若将distmax设为0,molclus启动后就会从当前目录下的distrange.txt里读取设置,其中每一行的格式为:原子组1序号 原子组2序号 最近距离 最远距离,距离单位为埃。例如,若此文件内容为1,3-5 2,8 2.5 10.2,就代表1,3,4,5和2,8这两批原子间最小距离必须大于2.5埃,最大距离不能超过10.2埃,如果traj.xyz里某结构违背了这个条件,则这个结构就会被跳过而不计算。distrange.txt里可以设多行,同时满足所有条件的结构才被计算。
• 2022-May-28修正了一个bug:isostat用-Edis设置判断重复结构的能量阈值时单位转换有问题
• 2022-Jun-8修正了genmer的$distcons设置存在的bug
• 2022-Jun-14使molclus完全兼容了Windows版xtb
• 2022-Jun-24修正了molclus调用xtb时settings.ini里freeze设置不生效的bug
• 2022-Sep-9修正了molclus调用ORCA时如果用了Recalc_Hess则统计的虚频数是所有次输出的虚频的总和的bug
2022-Mar-3:1.9.9.8版发布
• genmer组件的genmer.ini里的ishuffle选项现在可以设负值。如果设-N,则前N类分子插入的时候还是按顺序插入,而其余的分子则随机顺序插入。这对于构建很多体系很有用,比如构建某个溶质分子被两类分子混合构成的溶剂层包围,可以在genmer.ini里把溶质设为第一类分子,而第2、3类分子设成两种溶剂分子,并把ishuffle设为-1,这样构造出的体系中溶质分子肯定出现在中心。
2021-Nov-23:1.9.9.7版发布
• Molclus的settings.ini里现可以把energyterm参数设为"TD=",此时如果Gaussian模板文件对应的是TDDFT计算,Molclus会从Gaussian输出文件里读取TDDFT算的root指定的态的激发态能量。由此可以实现用molclus做激发态构象搜索或者激基复合物构型搜索。
2021-Sep-4:1.9.9.6版发布
2021-Sep-29:最后更新
• molclus的setting.ini设置文件里现在可以设置热力学量计算程序Shermo(http://sobereva.com/soft/shermo/)的路径和运行参数,当molclus调用Gaussian或ORCA做freq或opt freq任务后会自动调用Shermo来算热力学量,比Gaussian和ORCA自身算热力学数据的代码更灵活、强大、可靠。见《使用molclus程序做团簇构型搜索和分子构象搜索》(http://bbs.keinsci.com/thread-577-1-1.html)里加入的说明。
• genmer组件的genmer.ini设置文件里现在可以在末尾加入一行$distcons,然后下面通过一行或多行,指定一对或多对元素间必须满足的距离范围(没设的话就不做限制),指定特定元素间必须满足的距离范围(没设的话就不做限制),例如写H Mg 2.0就代表氢和镁之间距离不能小于2.0埃,Pt Pt 1.8 3.2代表铂之间距离必须在1.8~3.2埃。违背约束条件的构型会被视为失败而不输出。genmer.ini末尾还可以加入一行$modrad,然后在下面通过一行或多行,对一个或多个元素半径进行修改,比如Pt 3.5就把半径改为了3.5埃。详见《genmer:生成团簇初始构型的超便捷工具》(http://bbs.keinsci.com/thread-2369-1-1.html)里面新加入的说明和例子。
• genmer组件启动时如果发现当前目录下没有genmer.ini,将会提示用户手动输入配置文件路径,因此不是非得叫genmer.ini且放在当前目录不可了。molclus启动时如果发现当前目录下没有traj.xyz和settings.ini,也会直接让用户手动输入它们的路径。
• 修正了一个bug:molclus组件调用OpenBabel时产生的临时xyz文件里元素名都是大写,导致双字符的原子如Cl在OpenBabel用力场计算的时候没法正常指认原子类型。
2021-Jul-4:1.9.9.5版发布
2021-Aug-28:最后更新
• genmer组件的genmer.ini设置文件里现在可以用xmin、xmax、ymin、ymax用于控制分子出现的X、Y范围,和之前版本里的zmin、zmax用处类似。因此目前可以严格控制指定的分子出现的三维空间范围。
• xyz2QC组件的Gaussian模板文件中现在可以使用[FILENAME]占位,产生输入文件时它将被替换为"前缀 帧号",因此可以在模板文件里写比如%chk=/sob/[FILENAME].chk从而使得计算完毕后chk文件以恰当的名字留存在/sob目录下。
2021-Apr-15:1.9.9.4版发布
• isostat组件可以通过命令行运行并结合各种参数,更为强大也更便于嵌入到脚本里。比如执行isostat Rize.xyz -Nout 8 -Eout 2.4 -Edis 0.5 -Gdis 0.2 -T 350 -nt 32命令,代表将Rize.xyz 作为输入文件,往cluster.xyz里导出的簇是能量最低的8个且相对能量还必须在2.4 kcal/mol以内。使用0.5 kcal/mol和0.2埃分别作为归簇的能量和几何偏差判据。计算Boltzmann分布使用350K。使用32个线程并行计算。凡是通过命令行指定的参数就不需要在交互式界面里再输入。
2021-Feb-11:1.9.9.3版发布
• genmer的genmer.ini文件中现在允许写noxrot、noyrot、nozrot分别要求单体不能绕X、Y、Z轴随机旋转。也可以同时写两个,比如noxrot noyrot要求单体只能绕Z轴随机旋转。
2020-Dec-28:1.9.9.2版发布
• 解决了某些情况下Gaussian几何优化没收敛时Molclus会卡住的问题。
2020-Dec-10:1.9.9.1版发布
• 当molclus的settings.ini的igaucontinue=1时,如果Gaussian做优化失败后基于template2.gjf继续优化还没成功,这个新版本程序会试图基于当前目录下的template3.gjf继续进行优化,因此用户对于难收敛的情况可以有更多备选方案。
• settings.ini里对ORCA用户新增了ibkhess选项。若=1且任务涉及到振动分析,ORCA产生的.hess文件会被自动备份为 orca[序号].hess文件,便于用户之后重新利用Hessian。
2020-Oct-28:1.9.9版发布
2020-Nov-16:最后更新
• 加入了mol2_xyz组件,主要用于将Frog2构象生成程序产生的多帧mol2文件转换成traj.xyz文件,这种做法的意义见molclus新的教程《将Confab或Frog2与Molclus联用对有机体系做构象搜索》。
• 新特性:当当前目录下出现名为STOP的文件时,molclus对traj.xyz里各个结构的循环将被强行终止(每开始算一个新的结构时探测一次)。因此在当前目录下创建名为STOP的文件是一种优雅的强制结束molclus运行的做法。
• 新特性:如果isomers.xyz是molclus做优化+振动分析或振动分析任务产生的,isostat在处理时会报告哪些构型存在虚频。
• 改进:支持了目前最新版的crest程序输出的crest_ensemble.xyz
2020-Oct-17:1.9.8版发布
2020-Oct-18:最后更新
• 显著加快了isostat排序时候的速度,对于帧数非常多的情况甚至比老版本快一个数量级。
• 当molclus的settings.ini中iappend=1(续写模式)时,程序启动时不会自动将当前目录下.out、.chk、orca开头的文件删掉(因为这些可能是之前用ibkout、ibkchk等选项保留下来的有意义的文件)。
• 让ibkout、ibkchk等选项保留出来的文件的文件名中的序号与traj.xyz里帧号保持了一致(以前版本总是从1开始计)。这使得利用iappend=1时可以跑多次molclus将traj.xyz里的所有帧分别算完,确保每次备份出来的文件序号不会彼此覆盖
2020-Oct-14:1.9.7版发布
• molclus的settings.ini里新增加了iappend选项。如果设为1,新产生的结构会累加到当前目录下之前的isomers.xyz文件(如果有的话)末尾。如果为0,则原先的isomers.xyz会被新的数据覆盖。
• gentor的gentor.ini中若在旋转设置后面空一行写上reportfail=1,则会将未通过检查的构象的失败原因显示出来,判断是由于哪些原子间距离太近导致未通过检测。默认的critskip参数的阈值从0.8减小为了0.75,避免含有氰基的时候容易出现难以通过检测的情况。
• 如果运行gentor时当前目录下没发现mol.xyz或gentor.ini,会提示用户直接输入这两种文件的路径(文件名随意)
2020-Jul-12:1.9.6版发布
• 解决了itask=3且没有基于单点模板文件自动做高精度单点计算的时候,在isomers.xyz里记录的能量为0的bug(本应记录的是freq任务给出的自由能)。
2020-Jun-1:1.9.5版发布
• 将isostat组件做了并行化。当要处理的帧数很多的时候(好几千),在核数较多的机子上耗时可以降低一个数量级。
2020-Apr-21:1.9.4版发布
• 修正了itask=3且没有提供单点任务模板文件时给出的能量有误的bug。
2020-Feb-11:1.9.3版发布
• 兼容了crest程序的-mdopt任务产生的crest_ensemble.xyz文件(即可以当做isomers.xyz文件来被isostat处理。只要发现文件名里有crest字样就按照这种格式来读取)。
2020-Feb-5:1.9.2版发布
• 已支持在振动分析(itask=2)或优化+振动分析(itask=3)之后自动用高精度级别算单点能,从而直接给出较好精度的自由能。具体做法是在当前目录下提供template_SP.gjf(template_SP.inp)文件,设置Gaussian(ORCA)高精度单点计算的关键词,格式同template.gjf(template_SP.inp)。之后,当优化或优化+振动分析任务结束后,程序发现当前目录下有template_SP.gjf(template_SP.inp)时就会用其中的信息构成单点任务文件并调用Gaussian(ORCA)计算,并自动将新的单点能与自由能热校正量相加作为最终能量输出。几个细节:
(a)如果template_SP.gjf和template_SP.inp同时存在,优先考虑template_SP.inp
(b)opt、opt freq任务用的程序与自动算高精度单点的可以不同,比如iprog=1调用Gaussian时也可以通过提供template_SP.inp来令程序自动调用ORCA算高精度单点
(c)利用template_SP.gjf基于Gaussian自动算高精度单点时,Gaussian用户还需在settings.ini里对energyterm参数恰当设置,用于指定如何读取这个级别的单点任务输出文件里的能量
• 为提升灵活性,molclus支持了直接指定设置文件和初始结构文件的路径。例如molclus /RAS/Layer.ini代表将/RAS/Layer.ini作为设置文件。再比如molclus lock.ini /RAS/chu2.xyz代表用当前目录下的lock.ini的设定对/RAS/chu2.xyz里的结构进行计算。
2020-Jan-19:1.9.1版发布
• 兼容了2019年12月18日发布的xtb 6.3版预览版,对老版本xtb不再兼容。
2019-Nov-5:1.9版发布
• molclus的settings.ini支持了itask=2(振动分析)和itask=3(几何优化+振动分析),可结合Gaussian、ORCA和xtb使用。做这些任务时molclus会提取自由能、自由能热校正量、虚频数目,在屏幕上输出的同时也写入到isomers.xyz。之前版本里针对xtb的itask=-2被撤掉(已被itask=3代替)
• 去掉了molclus的settings.ini中意义不大的xtb_path(因为xtb运行命令是固定的)
• 在执行计算任务之前会对模板文件内容进行检查
2019-Sep-28:1.8.9版发布
• 更新了isostat。改进在于将某isomer纳入已有的簇的时候,如果这个isomer的能量比那个簇的能量更低,则这个簇的结构和能量将会被这个isomer的所代替。另外,从本版本开始的Windows版中,isostat.exe对应64bit版,而isostat_32bit.exe对应32bit版。
• 当ibkout>0而MOPAC运行没有正常结束(没生成arc)的时候,原先版本无法备份文件,而当前版本会将out进行备份。
• gentor里的critskip参数的默认值从原来的0.5改大为了0.8,避免得到的初始结构里有一些原子因为离得太近在优化后变成诡异结构。
• 修正了itask=0的时候与最新版xtb不兼容的问题
2019-Aug-12:1.8.8版发布。完美支持了近期发布的ORCA 4.2(之前版本的备份优化轨迹文件的功能不兼容ORCA 4.2)。从此molclus不再完美支持ORCA 4.2以前的版本
2019-Aug-7:1.8.7版发布。调用xtb之前会自动删除xtbrestart文件,减小了对某些体系调用最新版xtb程序优化失败的几率。
2019-Jul-28:1.8.6版发布。molclus支持了冻结原子,通过settings.ini里的freeze设置,如设成2,5-8,12-14代表在涉及优化的任务中将2,5,6,7,8,12,13,14号原子的XYZ笛卡尔坐标冻结。此选项对调用Gaussian、xtb、ORCA、MOPAC的情况皆生效。借助此选项可以实现诸如过渡态结构的构象搜索(即反应的区域冻结而只对其余部分进行构象搜索)。
2019-Jul-1:1.8.5版发布。molclus支持了对traj.xyz里完全自定义帧号范围进行计算,通过settings.ini里的ngeom设置,如设成2,5-8,12-14代表只处理第2,5,6,7,8,12,13,14帧
2019-Jun-12:1.8.4版发布。gentor程序的gentor.ini里增加了critskip选项,如果gentor产生的结构中有任意两个原子间距离小于两个原子共价半径乘以critskip值的话,则这个构象将不被输出到traj.xyz中(critskip默认值为0.5,这也是之前版本用的数值)。此外,molclus里ieneonly选项的名字改为了itask,其中增加了选项-2,此时若调用xtb将会做优化和振动分析(即--ohess任务),并且molclus读取的是自由能而非电子能量。
2019-May-26:1.8.3版发布。genmer程序的genmer.ini里增加了ngenmust选项,用于要求genmer运行结束前必须成功生成多少个构型,而不是像ngen选项那样指定的是期望生成多少个构型(其中有的可能生成失败)。
2019-May-15:1.8.2版发布(最后更新于2019-May-21)。改进了xyz2QC组件,产生单独的Gaussian或ORCA输入文件时现已可以指定文件名前缀,而且产生单独的Gaussian输入文件时会自动保留chk文件。给molclus的settings.ini增加了ibkgbw选项,可以让molclus调用ORCA计算每个体系后将gbw自动备份。
2019-Mar-30:1.8.1版发布。molclus的settings.ini里的ieneonly选项增加了-1,表示任务是调用Gaussian做热力学组合方法的计算。此时如果将template.gjf里的关键词设为比如G4,将energyterm=设为"G4 Free Energy=",则molclus提取的将是G4热力学组合方法算的高精度自由能。同时,此版本对settings.ini的格式做了轻微修改,并去掉了没用的isys选项。
此版本也更新了isostat,当归簇后簇的数目非常大(>500)时,会提示是否只保留用户指定数目的能量最低的簇,由此可避免屏幕上输出的簇的信息量过大、产生的cluster.xyz文件过大。同时给《gentor:扫描方式做分子构象搜索的便捷工具》文章增加了第4节,给出了通过molclus结合xtb和ORCA程序实现高度柔性分子精确构象搜索的实例。
2019-Mar-20:1.8版发布。扩展了genmer的功能,增加了rmin、zmax和zmin选项,从而能轻松地构造笼状团簇、平面团簇、环状团簇、桶状团簇的初猜结构,并进而结合molclus优化这些团簇的实际结构,这对于原子团簇研究很重要。请查看已更新的genmer的帖子了解详情。
2019-Mar-17:1.7版发布。新添加了xyz2QC工具,用于将molclus/genmer/gentor产生的多帧.xyz文件转化成Gaussian或ORCA的输入文件(含多步任务的单一文件或者一批单独的文件),便于用户往超算上直接提交。点此链接查看详情。
2019-Feb-12:1.6版发布。大幅扩展了genmer和gentor的功能,并令molclus兼容2019-Feb-8发布的xtb。点此链接查看详情。
2019-Jan-1:为造福广大计算化学工作者,molclus从此由收费变为免费!
2018-Aug-28:1.5版发布。支持了Open Babel(http://openbabel.org),这是免费的格式转换程序,同时附带基于力场的计算功能。通过令molclus调用此程序可以实现基于MMFF94、GAFF、UFF等力场的几何优化和能量计算任务。
2018-Jun-25:1.4.3版发布。对xtb程序作了更好的支持,可以指定运行选项。另外对gentor进行了升级,现在可以用比如e5 120 140来让片段依次旋转120,125,130,135,140度。同时修正了一些bug。
2017-Dec-1:1.4版发布。支持了Grimme开发的做GFN-xtb (见J. Chem. Theory Comput., 13, 1989)计算的xtb程序(可由此免费获得https://www.chemie.uni-bonn.de/pctc/mulliken-center/software/xtb/xtb)。settings.ini里增加了mpioption选项,用于设定Linux下ORCA并行运行时给mpirun的额外参数,例如root用户下运行需要的--allow-run-as-root。
2016-Sep-16:1.3.5版发布。修正了一个某些情况下无法正确读取优化的结构,有时还会导致死循环的bug。
2016-Aug-16:1.3.4版发布。gentor可以现在可以直接设定原子间是否成键,以及设定成键阈值。
2016-Jul-10:1.3.3版发布。修正了gentor无法剔除含有原子过近接触的结构的bug。修正了对计算时间统计的bug。
2016-Jun-18:1.3.2版发布。通过settings.ini里的ibkchk可以将Gaussian对每个结构计算产生的.chk文件进行保留。
2016-Apr-28:1.3.1版发布。此版本修正了读取ORCA优化任务能量错误问题。并且可以靠ngeom参数选择只考虑traj.xyz中某一范围的结构。
2015-Dec-18:1.3版发布。此版本加入了gentor子程序用于通过扫描方式产生分子构象搜索用的初始结构。
2015-Dec-15:1.2版发布。此版本加入了genmer子程序用于方便地批量产生团簇构型的初始结构。isostat增加了输出波尔兹曼分布的功能。
2015-Feb-8:1.1版发布。此版本对所含isostat进行了升级,在对结构归簇时,不仅对比能量,还对比几何结构偏差,都小于设定的阈值时才被归到一个簇里。由此可以区分开能量接近简并的不同构型/构象。
2015-Jan-5:1.0版发布
Molclus辅助工具
以下是Molclus用户们开发的Molclus辅助工具
《consearch:一键提交slurm的molclus构象搜索脚本》(http://bbs.keinsci.com/thread-43932-1-1.html)
《chatGPT辅助生成molclus懒人脚本:一键完成对复杂天然产物NMR和ECD计算》(http://bbs.keinsci.com/thread-41199-1-1.html)
《分享molclus配套PBS作业提交脚本》(http://bbs.keinsci.com/thread-15598-1-1.html)
《分解合并Molclus任务的小脚本splitMC》(http://bbs.keinsci.com/thread-14361-1-1.html)
用户们使用Molclus已发表的文章
Molclus由于其强大、灵活、易用,已经被巨量论文所使用,如下所示,由新到旧排序。这里顺带对不按照求正确引用Molclus的文章表示谴责!这些文章都已做了标记。
- Liyuan Chen, Zhisong Chen, Jiafen Qian, et al., Deep eutectic solvents-based extraction of organic acids from Angelica sinensis:process optimization and mechanistic insights, China Pharmacy, 36, 2783 (2025) https://journal.china-pharmacy.com/rc-pub/front/front-article/download/136772261/lowqualitypdf/%E5%BD%93%E5%BD%92%E6%9C%89%E6%9C%BA%E9%85%B8%E7%9A%84%E4%BD%8E%E5%85%B1%E7%86%94%E6%BA%B6%E5%89%82%E6%8F%90%E5%8F%96%E5%B7%A5%E8%89%BA%E4%BC%98%E5%8C%96%E5%8F%8A%E6%9C%BA%E5%88%B6%E7%A0%94%E7%A9%B6.pdf
- Wei Wang, Yujie Chen, Mengyao Shi, et al., Thermal decomposition mechanism of tert-butyl peroxybenzoate: insights from calorimetry, GC/MS, and quantum chemical calculation, J. Therm. Anal. Calorim. (2025) https://link.springer.com/article/10.1007/s10973-025-15000-2
- Changhang Zhang, Xinyu Wu, Xiaoyu Li, Hailan Lian, Photoactivated coordination polymerization, ChemRxiv (2025) https://chemrxiv.org/engage/chemrxiv/article-details/69319e17ef936fb4a213477f
- Tengfei Zhao, Guojun Zhu, Changgui Wu, et al., Variation of aroma compounds in soy sauce aroma type baijiu during aging: Focusing on the regulatory effect of ethyl lactate on aroma release, Food Chem. X (2025) https://www.sciencedirect.com/science/article/pii/S259015752501243X
- Weibin Zhang, Xiaohong Wang, Ying Zhao, et al., Mechanistic Investigation and Process Simulation of Liquid–Liquid Extraction of Pyridine-Based Pollutants from Aqueous Solutions Using Hydrophobic Deep Eutectic Solvents, Ind. Eng. Chem. Res. (2025) https://pubs.acs.org/doi/abs/10.1021/acs.iecr.5c03725
- Houzhi Wang, Xiao Tian, Jiayue Yun, et al., Mechanism of regeneration effect of aromatic hydrocarbons on aged asphaltene: a combined study of DFT calculations and experiments, Int. J. Pave. Eng., 26, 2592687 (2025) https://www.tandfonline.com/doi/abs/10.1080/10298436.2025.2592687
- Chengde Ding, Baolei Tang, Yuxing Zhou, et al., Cryogenically self-healing organic crystals, Nat. Mater. (2025) https://www.nature.com/articles/s41563-025-02411-7
- Hongping Liu, Hao Su, Ning Chen, et al., Confirmation-dependent organic phosphor reveals amino acid nanoaggregates in ice with insight for prebiotic chemistry, Nat. Commun., 16, 10873 (2025) https://www.nature.com/articles/s41467-025-65885-8
- Yingnan Sun, Cuicui Sun, Zhengkai Wu, et al., Mechanism of 5-hydroxymethylfurfural preparation from glucose by a nonmetallic Lewis/Brønsted bifunctional solid acid catalyst CNs-PF6 under photo-induction, Fuel Proc. Technol., 280, 108370 (2025) https://www.sciencedirect.com/science/article/pii/S0378382025001948
- Xuefeng Liu, Shengpeng Hao, Xin Gao, et al., High-Accuracy Theoretical Studies on the Gas-Phase Reaction Mechanisms of Sulfur Mustard with Reactive Oxygen Species (OH/O 2 /HO 2 /O), Phys. Chem. Chem. Phys. (2025) https://pubs.rsc.org/en/content/articlehtml/2025/cp/d5cp03593g
- Qingfeng Wu, Jun Wang, Yuting Xiao, et al., Unveiling the Clustering-Triggered Emission Mechanism of Raffinose Aqueous Solution and Ion Detection, ChemistrySelect (2025) https://chemistry-europe.onlinelibrary.wiley.com/doi/abs/10.1002/slct.202505092
- Yan Lei, Wang Ping-Ping, Fu Xiong, 113516The improvement in stability and coloration effect of gallic acid acylation of anthocyanins and their quantum chemical mechanism, Food Wellness (2025) https://www.sciencedirect.com/science/article/pii/S3050711125000349
- Xiaojun Bao, Zhiyuan Ma, Dongshun Deng, Efficient extraction of phenol from oil using halogen-free deep eutectic solvents: experimental investigation and computational analysis, Fuel, 408, 137743 (2026) https://www.sciencedirect.com/science/article/abs/pii/S0016236125034696
- Yingda Zhang, Shunyu Wang, Tianhe Xu, et al., Sustainable ether electrolyte engineering for all-climate and durable viologen electrochromic devices, Sust. Mater. Technol., 47, e01792 (2026) https://www.sciencedirect.com/science/article/abs/pii/S2214993725005603
- Xionghui Ma, Wenwen Cao, Rui Chen, et al., Metal-bridged self-assembly of recognition-functionalized MCOFs for ultrasensitive photoelectrochemical sensing of perfluorooctanoic acid, Chem.Eng. J., 526, 171291 (2025) https://www.sciencedirect.com/science/article/abs/pii/S1385894725121384
- Xin-Cheng Xu, Xiao-Xia You, Jian-Sen Wang, e al., Theoretical study on the conformational diversity and dynamic effect in Pd-catalyzed atroposelective Suzuki–Miyaura cross coupling, J. Chem. Phys., 163, 204305 (2025) https://pubs.aip.org/aip/jcp/article-abstract/163/20/204305/3373199/Theoretical-study-on-the-conformational-diversity
- Chao Sun, Feng Xue, Peizhe Cui, et al., Molecular mechanism and extraction process design for the separation of ethanol and N-propyl acetate by deep eutectic solvents and ionic liquids, Proc. Saf. Environ. Prot., 205, 108206 (2026) https://www.sciencedirect.com/science/article/pii/S0957582025014739
- Xiuwu Liu, Dexin Kong, Guoyun Tong, et al., Microsize and environment regulation of Mn-based MOF for inverse ethane/ethylene separation, Sep. Purif. Technol., 382, 136172 (2026) https://www.sciencedirect.com/science/article/abs/pii/S1383586625047690
- Yuqian Peng, Shanyu Han, Xiaobin Cao, et al., Theoretical evaluation of kinetic triple oxygen isotope fractionation during sulfite oxidation by atmospheric oxygen, Geochim. Cosmoch. Acta (2025) https://www.sciencedirect.com/science/article/abs/pii/S001670372500643X
- Xueer Hu, Xinyu He, Chao Guo, et al., Trade-off between thermodynamic ability and recovery economics of ionic liquid for the sustainable separation of propyl propionate/n-propanol/water azeotropes, Proc. Saf. Environ., 205, 108184 (2026) https://www.sciencedirect.com/science/article/pii/S095758202501451X
- Qiuyu Chen, Siqi Zhu, Yimin Wang, et al., Water-induced modulation of natural deep eutectic solvents properties: A case study of the L-lysine-glycerol system, Fuel, 408, 137610 (2026) https://www.sciencedirect.com/science/article/abs/pii/S0016236125033368
- Mingmei Wang, Junyang Liu, Wenwen Wang, et al., Multi-scale design and optimization of composite absorbents for selective absorption of maleic anhydride from n-butane oxidation gas streams, Chem. Eng. Sci. (2025) https://www.sciencedirect.com/science/article/abs/pii/S0009250925018317
- Xueying Zheng, Haotian Zhu, Zhongqiang Wang, et al., Nanoengineered aqueous-hydrotrope hybrid liquid electrolyte solutions for efficient zinc batteries across a wide temperature range, Nat. Nanotechnol. (2025) https://www.nature.com/articles/s41565-025-02060-6
- Shuhua Liang, Zhaohui Liang, Minting Liang, et al., Lignin stabilizes lauric-stearic acid eutectics and suppresses supercooling for efficient thermal energy storage, Chem. Eng. Sci., 321, 122996 (2026) https://www.sciencedirect.com/science/article/abs/pii/S0009250925018172
- Bingqing Sun, Hui Wang, Lei Liu, Photophysics of a coumarin-based fluorescence probe for Al3+: Relations between multiple ESIPT processes and TICT states, Spectrochim. Acta A (2025) https://www.sciencedirect.com/science/article/abs/pii/S1386142525015045
- Siming Chen, Jingchao Liu, Xinzhu Zhang, Shijian Lu, Low-energy N-(2-hydroxyethyl)ethylenediamine based biphasic solvent for CO2 capture, part I: Design with N,N-Diethylacetamide as a phase separation agent, Sep. Purif. Technol. (2025) https://www.sciencedirect.com/science/article/abs/pii/S1383586625046751
- Hongyu Wen, Zhisheng Lai, Jinhui Wang, et al., Enabling the Identification of Dopamine Receptor Ligands from Complex Matrices Using a Cyclic Tetrapeptide, Anal. Chem. (2025) https://pubs.acs.org/doi/abs/10.1021/acs.analchem.5c04692
- Yue Li, Qiyue Wei, Jinhui Xie, et al., Unraveling the Deactivation Precipitation Mechanism of Iridium Catalysts in Methanol Carbonylation to Acetic Acid via Dissipative Particle Dynamics, Langmuir (2025) https://pubs.acs.org/doi/abs/10.1021/acs.langmuir.5c03230
- Yingjie Huang, Yushun Zhou, Yulu Wang, et al., Environment-Responsive Self-Assembly and Coupled-Network Effect in Dynamic Replicating Nucleopeptide Networks, Angew. Chem. (2025) https://onlinelibrary.wiley.com/doi/abs/10.1002/anie.202518297
- Xiaoyu Zhang, Tai Wang, Yi Qin Gao, Yunlong Xiao, Noncollinear Spin-Flip TDDFT for Potential Energy Surface Crossings: Conical Intersections and Spin Crossings, J. Chem. Theory Comput. (2025) https://pubs.acs.org/doi/abs/10.1021/acs.jctc.5c01272
- Yongkang Cui, Xinyue Chen, Yilun Zhou, et al., Tuning carbon chain length and methyl group of triamine for high-efficiency CO2 biphasic solvents with enhanced oxidative stability, Sep. Purif. Technol. (2025) https://www.sciencedirect.com/science/article/abs/pii/S1383586625046283
- Ning Li, Dingkai Hu, Chenqi Peng, et al., Chitosan composite aerogels doped with oilfield drill cuttings for arsenic removal: A combined experimental and computational approach, Int. J. Biol. Macromol., 333, 148865 (2025) https://www.sciencedirect.com/science/article/abs/pii/S014181302509422X
- Ke Zhang, Huina Liu, Yan Miao, et al., Enhancing polymorphic purity by inducing specific pre-assembled dimer and reducing template particle size, Chem. Eng. J. (2025) https://www.sciencedirect.com/science/article/abs/pii/S1385894725113843
- Zhenyu Zhang, Renting Li, Yixian Xu, et al., Separation of Methyl Ethyl Ketone/Methanol Azeotrope via Extractive Distillation with Ionic Liquids: Mechanism Analysis and Process Intensification, ACS Sustainable Chem. Eng. (2025) https://pubs.acs.org/doi/abs/10.1021/acssuschemeng.5c08737
- Yongqing Zhang, Xiaolong Shang, Jiaqi Guo, et al., A computationally driven screening-construction-mechanism strategy of magnetic molecularly imprinted polymers for aliphatic aldehydes detection, Food Chem. (2025) https://www.sciencedirect.com/science/article/abs/pii/S0308814625042268
- Yudi Wang, Wenrui Li, Xin Wu, et al., Graphene oxide doping of the hole injection layer enables 23.6% efficiency in perovskite solar cells with carbon electrodes, Nature Energy (2025) https://www.nature.com/articles/s41560-025-01893-8
- Naixin Zhao, Yonglin Wang, Xin Jin, Yuning Li, An Intrinsically Conductive Cross-Conjugated Polymer with a Quinhydrone-Like Donor–Acceptor Charge-Transfer Network, Angew. Chem. Int. Ed., 2025, e18109 (2025) https://onlinelibrary.wiley.com/doi/pdf/10.1002/ange.202518109
- Zhanpeng Qin, Shirong Wang, Yanliu Zhu, et al., Suppression of Excimer Formation via Conjugation-Driven Interactions in Crosslinked Hole Transport Layer for Efficient Blue Quantum Dot Light-Emitting Diodes, Angew. Chem. Int. Ed. (2025) https://onlinelibrary.wiley.com/doi/abs/10.1002/anie.202519610
- Bin Jiang, Yan Cui, Shuai Li, et al., Salicylate-based ionic liquids for extraction of critical metals from spent lithium-ion batteries via coordination, Chem. Eng. Sci., 321, 122899 (2026) https://www.sciencedirect.com/science/article/abs/pii/S0009250925017208
- Peng Cui, Shideng Yuan, Xuxue Zhang, et al., Aging-induced changes in the molecular structure and interactions of pavement asphalt, Comput. Theor. Chem., 1255, 115579 (2026) https://www.sciencedirect.com/science/article/pii/S2210271X25005158
- Xiaolong Yi, Yongtao Wang, Haoran Li, Unveiling the Influence of Hydrogen-Bonding Dynamics on Water-Affected Methyl Internal Rotation by Microwave Spectroscopy, J. Phys. Chem. A (2025) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.5c04492
- Ming Lin, Yihong Liu, Jinzhao Guan, et al., Exploring Species in Low-Molar-Ratio NaF-AlF3 Molten Salts Using Raman Spectroscopy and Quantum Chemical Calculations, Inorg. Chem. (2025) https://pubs.acs.org/doi/abs/10.1021/acs.inorgchem.5c04372
- 董志, 舒启江, 田爽, et al., 黄芩苷与苦参碱分子间相互作用的量子化学计算与验证, Journal of Atomic & Molecular Physics, 42, 14 (2026) https://doi.org/10.19855/j.1000-0364.2025.041002
- Yuantong Liu, Mengqin Yao, Fei Liu, et al., Hydrophilic Engineering of MOF-Derived Pd Nanocatalysts Enables Efficient Formic Acid Dehydrogenation: Intermolecular Protons/Hydrides Shuttling Mediated by FA–H2O Hydrogen-Bonding Network, ACS Appl. Mater. Interfaces (2025) https://pubs.acs.org/doi/abs/10.1021/acsami.5c13772
- Fang Zhao, Wukun Zhong, Chenyu Ding, et al., Carrier-free self-assembled nanodrug with responsive release of carbon monoxide and antisense oligonucleotide for synergistic tumor therapy, Colloid Surface B, 258, 115220 (2026) https://www.sciencedirect.com/science/article/abs/pii/S0927776525007271
- Qian Xu, Fangfang Ma, Chang Liu, et al., Chlorine Radical-Initiated Atmospheric Oxidation of Imines: Implications for Structural Influence on the Nitrosamine Formation, EGUsphere (2025) https://doi.org/10.5194/egusphere-2025-4896
- Yuling Huang, Bingjia Xu, Huiwen Zeng, et al., Simultaneously fluorescence-phosphorescence dual-emission based on phenoxathiin and polycyclic aromatic hydrocarbons towards temperature sensing, Mater. Chem. Front. (2025) https://pubs.rsc.org/en/content/articlelanding/2025/qm/d5qm00618j/
- Hui Xu, Zhengrun Chen, Sinan Cheng, et al., Experimental and computational insights into efficiently synthesizing trimethyl orthoformate from calcium methoxide and chloroform, J. Ind. Eng. Chem. (2025) https://www.sciencedirect.com/science/article/abs/pii/S1226086X25006951
- Long Chen, Yu Huang, Yonggang Xue, et al., Mechanistic investigations of the formation of highly oxidized1 products from the multi-generation OH oxidation of styrene, EGUsphere (2025) https://egusphere.copernicus.org/preprints/2025/egusphere-2025-4646/egusphere-2025-4646.pdf
- Wei Dai, Yiqin Zheng, Nanchen Lai, et al., Ultrasound-Assisted green extraction and resin purification of Hypaphorine from Nanhaia speciosa using deep eutectic solvents, Ultrason. Sonochem., 122, 107644 (2025) https://www.sciencedirect.com/science/article/pii/S1350417725004237
- Jiping Liao, Xinnan Li, Cheng Fan, et al., Discovery of a Natural Small-Molecule Inhibitor to Novel Target STAT5A for Ischemic Stroke Therapy, J. Med. Chem. (2025) https://pubs.acs.org/doi/abs/10.1021/acs.jmedchem.5c01752
- Xuting Si, Shuai Wei, Yilin Yang, et al., Novel bifunctional aminophosphonic acid extractants for efficient lithium recovery from spent battery leachates with high sodium interference, Hydrometallurgy (2025) https://www.sciencedirect.com/science/article/abs/pii/S0304386X25001653
- Jianwei Luo, Yingsi Huang, Xinghai Huang, et al., Closed-Loop Utilization of Sophora tonkinensis Gagnep.: Nanocellulose from Non-Medicinal Parts into Magnetic Imprinted Polymers for Targeted Quercetin Separation of Medicinal Parts, ACS Sustainable Chem. Eng. (2025) https://pubs.acs.org/doi/abs/10.1021/acssuschemeng.5c05620
- Jian-Sen Wang, Xiao-Xia You, Lu Li, et al., Stereogenic and Conformational Diversity in Multicomponent Ir-Catalyzed Borylation of Arenes: Unveiling the Origin of Enantioselectivity by Conformational Sampling, JACS Au (2025) https://pubs.acs.org/doi/full/10.1021/jacsau.5c00830
- Yongfeng Liu, Yuanchao Shao, Chenyang Yin, et al., Effect of NH3 and product NO on the oxidation of C7H16 under CO2/O2 atmosphere: quantum chemistry and SVUV-PIMS experiment, Fuel, 406, 137122 (2026) https://www.sciencedirect.com/science/article/abs/pii/S0016236125028479
- Wenyu Ye, Haonan Chu, Zihan Wang, et al., High-temperature Decomposition Characteristics and Mechanism of Environmentally Friendly Insulating Gas PMVE at the Molecular Scale, IEEE Transactions on Dielectrics and Electrical Insulation (2025) https://ieeexplore.ieee.org/abstract/document/11203241
- Kai Sun, Tsukasa Irie, Kohki Sasaki, et al., Architecting Redox-Active Framework Interfaces for Synergistic Sulfur Adsorption and Catalysis in Lithium–Sulfur Batteries, ChemRxiv (2025) https://chemrxiv.org/engage/api-gateway/chemrxiv/assets/orp/resource/item/68ebac9fbc2ac3a0e09666fd/original/architecting-redox-active-framework-interfaces-for-synergistic-sulfur-adsorption-and-catalysis-in-lithium-sulfur-batteries.pdf
- Jipeng Xu, Xiaojuan Tang, Jin Cheng, et al., Multitarget Design of Fluorinated Carboxylate Electrolytes for Lithium–Metal Batteries under Low Temperature, J. Phys. Chem. B (2025) https://pubs.acs.org/doi/abs/10.1021/acs.jpcb.5c04003
- Ating Li, Hao Dong, Bo Wang, et al., Enhanced H2S capture using sterically hindered amine-based non-aqueous solvents: experimental and computational simulation, Sep. Purif. Technol. (2025) https://www.sciencedirect.com/science/article/abs/pii/S1383586625042467
- Baiyun Jin, Hongmei Du, Mingzhao Zhu, et al., Enhanced demulsification of W/O emulsions via a tailored amphiphile with tri-hydrophobic architecture and multifunctional hydrogen bonding arrays, J. Water. Proc. Eng., 79, 108889 (2025) https://www.sciencedirect.com/science/article/abs/pii/S2214714425019622
- Biao Liu, Dingkai Hu, Jiajie Liu, et al., Application of imidazole ionic liquids in extractive distillation of benzene-cyclohexane system: Experimental verification and mechanism analysis, Chem. Eng. Res. Des., 223, 280 (2025) https://www.sciencedirect.com/science/article/abs/pii/S026387622500543X
- Junhong Xie, Yi Qin, Quanyu Zhao, One-step green approach for lutein extraction from Chlorella sp., Algal Res., 92, 104345 (2025) https://www.sciencedirect.com/science/article/abs/pii/S2211926425004564
- Qian Lin, Guangju Ma, Xinmin Liu, et al., Efficient Extraction of Phenolics from Coal Gasification Wastewater by Hydrophobic Deep Eutectic Solvents: Experimental and Mechanistic Study, Proc. Safety Environ. Protect. (2025) https://www.sciencedirect.com/science/article/abs/pii/S095758202501273X
- Jun Zhang, Jize Sun, Yujie Zhao, et al., Dissociative photoionization of 3-methyl-3-buten-1-ol: experiments and calculations, J. Mol. Struct. (2025) https://www.sciencedirect.com/science/article/abs/pii/S0022286025029205
- Jian Yang, Cheng Bian, Shengjie Guan, et al., Influence of intermolecular interaction about polyhedral oligomeric silsesquioxane and silicone rubber on mechanical and thermal properties, Coll. Surf. A (2025) https://www.sciencedirect.com/science/article/abs/pii/S0927775725024926
- Dezhi Cao, Dingkai Hu, Wenna Wang, et al., Coal tar phenolic DES back-extraction study based on Hansen parameters: A combined experimental and computational approach, Fuel, 406, 137030 (2026) https://www.sciencedirect.com/science/article/abs/pii/S0016236125027553
- Jianfei Xu, Zhiwei Han, Yaning Li, et al., Quantum chemical study on the weak intermolecular interaction between 3,4-bis(3-nitrofurazan-4-yl) furoxan (DNTF) and microcrystalline wax, J. Mol. Model., 31, 288 (2025) https://link.springer.com/article/10.1007/s00894-025-06512-7
- Lu Hu, Yuanbin Li, Shanshan Li, et al., Green extraction of proteins and anti-skin aging peptides from Tricholoma Matsutake using natural deep eutectic solvents, J. Food Meas. Charact. (2025) https://link.springer.com/article/10.1007/s11694-025-03573-3
- Mengna Xu, Weikang Gao, Chuanlei Liu, et al., Leveraging π-Conjugation in Benzimidazolium Ionic Liquids for Selective Extraction of Bicyclic Aromatic Hydrocarbons: A Combined Experimental and Computational Study, Energy Fuels (2025) https://pubs.acs.org/doi/abs/10.1021/acs.energyfuels.5c04215
- Han-Chuan Wang, Yu-Qi Shi, Bing-Hang Ji, et al., Sesquiterpenoids from Eupatorium chinense L. induce G0/G1 cell cycle arrest and apoptosis in AGS cell via DNA-PK/AKT/p53 pathway, Bioorg. Chem. (2025) https://www.sciencedirect.com/science/article/abs/pii/S0045206825009393
- Hailin Ran, Mengdie Ren, Hui Li, et al., Extraction of Macranthoidin B and Dipsacoside B from Lonicera macranthoides Utilizing Ultrasound-Assisted Deep Eutectic Solvent: Optimization of Conditions and Extraction Mechanism, ACS Omega (2025) https://pubs.acs.org/doi/full/10.1021/acsomega.5c03885
- Liwen Zhuang, Yu Chen, Zhigang Lei, et al., Enhanced solubility of carbamazepine using cholinium-based ionic liquid: From COSMO-RS screening to molecular dynamics simulation, Chem. Eng. Sci. (2025) https://www.sciencedirect.com/science/article/abs/pii/S0009250925015301
- Mingmei Wang, Junyang Liu, Bin He, et al., Theoretical Development of the Composite Absorbent for Maleic Anhydride Production: Insights from COSMO-RS, VLE Experiments, and Process Simulation, Ind. Eng. Chem. Res. (2025) https://pubs.acs.org/doi/abs/10.1021/acs.iecr.5c01925
- Ying Chen, Yan Qi, Deqi Peng, et al., The Role of Hydrogen Bonds in Thermodynamic and Structural Properties in Binary Mixtures of Amino Acid Ionic Liquid–H2O: A Combined Experimental and Computational Study, J. Phys. Chem. B (2025) https://pubs.acs.org/doi/abs/10.1021/acs.jpcb.5c02525
- Weihao Chen, Xiangliu Chen, Mengjing Cong, Jianglian She, et al., Novel Cyclic Tetrapeptides as Neuraminidase Inhibitors from a Sponge-Associated Penicillium sp. SCSIO41035, Mar. Drugs, 23, 377 (2025) https://doi.org/10.3390/md23100377
- Yujian Li, Xuefei Wu, Qian-Rui Huang, et al., Structural insights into nitrile–methanol hydrogen-bonded complexes based on mass-selected infrared spectroscopy and theoretical calculations Available to Purchase, J. Chem. Phys., 163, 124304 (2025) https://pubs.aip.org/aip/jcp/article-abstract/163/12/124304/3364288/Structural-insights-into-nitrile-methanol-hydrogen
- Di Wu, Chen Chen, Wei Chen, et al., Synergistic enhancement of anti-migraine efficacy through supramolecular self-assembly of tizanidine with meloxicam, J. Controlled Release, 387, 114248 (2025) https://www.sciencedirect.com/science/article/abs/pii/S0168365925008600
- Naisheng Wu, Fangling Wu, Sisi He, et al., Molecular-Level Insights into TIMS-MS Separation of α/β-Estradiol Isomers: Synergistic Effects of Cyclodextrins and Metal Ions, Anal. Chem. (2025) https://pubs.acs.org/doi/abs/10.1021/acs.analchem.5c03830
- Xincheng Zhang, Changqing He, Lin He, et al., Breaking oil–water emulsions co-stabilized by CaCO3 solid and natural stabilizers: efficient strategy and mechanisms, Chem. Eng. Sci. (2025) https://www.sciencedirect.com/science/article/abs/pii/S0009250925014484
- Meifang Chen, Shengrui Tong, Shanshan Yu, et al., Elucidation of the myrcene ozonolysis mechanism from a Criegee Chemistry perspective, EGUsphere (2025) https://doi.org/10.5194/egusphere-2025-4310
- Chao Guo, Kai Luo, Wei Hou, et al., A novel [MMIM]-based ionic liquid extractive distillation process for achieving liquid-phase propylene at 1.8 MPa with enhanced energy-economic-environmental benefits, J. Cleaner Prod., 525, 146579 (2025) https://www.sciencedirect.com/science/article/abs/pii/S0959652625019298
- Dongxia Wu, Wenxuan Hao, Qiyan Liu, et al., Theoretical insights into “D-A-D” type Aza-BODIPY dyes for enhanced photothermal performance, Chem. Phys. Lett., 880, 142421 (2025) https://www.sciencedirect.com/science/article/abs/pii/S0009261425005639
- Xinru Tan, Jihui Lu, Yihang Zhao, et al., Analogical prediction of chiral-regulated self-assembling hydrogel guided by computational chemistry and molecular similarity principles to enhance wound healing, Chem. Eng. J. (2025) https://www.sciencedirect.com/science/article/abs/pii/S1385894725091776
- Chenzhou Wang, Lanling Liu, Yutong Liu, et al., Deep eutectic solvents-assisted alkali lignin modification for pyrolytic monophenols production, Int. J. Biol. Macromol., 328, 147584 (2025) https://www.sciencedirect.com/science/article/abs/pii/S0141813025081413
- Zhihui Wang, Xiaobin Ding, Cairong Zhang, Chenzhong Dong, Quantum Chemistry Study of Hydrophilic Sulfonated Preorganized Ligand: Complexation Behavior and Selective Separation of Th(IV)/U(VI) from Ln(III), J. Mol. Liq. (2025) https://www.sciencedirect.com/science/article/abs/pii/S0167732225016356
- Houzhi Wang, Xiao Tian, Jiayue Yun, et al., Comprehensive shedding light on the strength and nature of interactions among SARA fractions via configurational search, Road Mater. Pavement Design (2025) https://www.tandfonline.com/doi/full/10.1080/14680629.2025.2549576
- Liyan Jia, Kaixiang Zhou, Xuechun Zhang, et al., High Stability and Low Irritation of Enrofloxacin– Colistin Combination Injection Through a Tripartite Strategy, Drug Design Dev. Therapy (2025) https://doi.org/10.2147/DDDT.S536132
- Hanyu Wu, Molecular Insights into Carbon Spacer Length Effects on Sulfobetaine–Phosphoric Acid Interactions: Optimizing Acid Retention for Polymer Electrolytes, J. Phys. Chem. B (2025) https://pubs.acs.org/doi/abs/10.1021/acs.jpcb.5c03775
- Xizi Du, Ziyang Huang, Qi Shao, et al., Acrylamide-Linked Water-Soluble Nitroxide Biradicals with Enhanced Electron–Electron Couplings for Dynamic Nuclear Polarization at 14.1 T, J. Phys. Chem. Lett., 16, 9217 (2025) https://pubs.acs.org/doi/full/10.1021/acs.jpclett.5c01749
- Yan-li Zhang, Peng Jiang, Wu-yan Chen, et al., Three undescribed Schinortriterpenoids from Schisandra chinensis and their neuroprotective effects, Fitoterapia, 186, 106853 (2025) https://www.sciencedirect.com/science/article/abs/pii/S0367326X25004794
- Mengtian Chen, Mengdi Yuan, Tong Bu, et al., Effect of food matrices on AFB1 recovery by lateral flow immunoassay with deep eutectic solvents as a green alternative to conventional extraction, Microchim. Acta, 192, 627 (2025) https://link.springer.com/article/10.1007/s00604-025-07488-0 (此文没有按要求引用Molclus!)
- Wen-Shuai Dai, Xi-Long Li, Hong-Guang Xu, et al., Geometric Structures and Electronic Properties of Co2Ge10-/0 : Anion Photoelectron Spectroscopy and Quantum Chemistry Calculations, Chin. J. Chem. Phys. (2025) https://cjcp.ustc.edu.cn/hxwlxb/en/article/id/0371551e-b8fe-4308-86ec-4f02bb0de8d6?viewType=HTML (此文没有按要求引用Molclus!)
- Yuanbin Li, Siyuan Chen, shanshan Li, et al., Enhanced White Tea Extraction and Multifunctional Dermatoprotection via L-Proline/Xylitol NaDES: Antioxidant, Anti-photoaging, and Anti-glycation Effects, New J. Chem. (2025) https://pubs.rsc.org/en/content/articlehtml/2025/nj/d5nj02454d (此文没有按要求引用Molclus!)
- Xiao-Fei Gao, Wenjin Cao, Shihu Deng, Xue-Bin Wang, Structure and stabilization mechanism of [ATP4–∙Mg2+]2–: a joint negative ion photoelectron spectroscopic and computational study, Chin. J. Chem. Phys. (2025) https://cjcp.ustc.edu.cn/hxwlxb/article/id/a68baae7-539e-4e45-98b0-42e3259163d0
- Yuting Wan, Hang Luo, Zhongna Yan, et al., Ultrahigh Energy Density in Dielectric Polymers Near Glass Transition Temperature by Molecular Twisting Conformation Locking, Adv. Mater. (2025) https://advanced.onlinelibrary.wiley.com/doi/abs/10.1002/adma.202510122
- Chunxiao Gong, Zhaoyang Ju, Qixuan Lin, et al., One-step valorization of cellulose acetate plastic waste into 5-hydroxymethylfurfural, Appl. Catal. B, 381, 125880 (2026) https://www.sciencedirect.com/science/article/abs/pii/S092633732500863X (此文没有按要求引用Molclus!)
- Zhengrun Chen, Haojie Chu, Hui Xu, et al., Molecular insights and experimental verification on reactivity regulation of CaC2 by ionic liquid solvents, AIChE J. (2025) https://aiche.onlinelibrary.wiley.com/doi/abs/10.1002/aic.70057
- Demiao Ma, Qiuhao Yi, Wenqiang Ren, et al., Gas-Phase Synthesis and Soft-Landing Deposition of Silver-Coumarin Complexes [Ag(C9H6O2)2]+ within Weakly Bonded Interactions, Inorg. Chem. (2025) https://pubs.acs.org/doi/abs/10.1021/acs.inorgchem.5c02999
- Zichun Zhang, Lin Cheng, Xia Shao, Lisha Ma, DFT Mechanism Study of Ligand-Controlled Ru-Catalyzed Selective Aerobic Oxidative Generation of Homocoupled Imines and Nitriles, J. Phys. Chem. A (2025) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.5c02676
- Jingya Hou, Yutong Wang, Yu Sheng, et al., Experimental Investigation and Mechanism Analysis for the Separation of sec-Butanol and sec-Butyl Acetate System via Liquid–Liquid Extraction with Ionic Liquids, J. Chem. Eng. Data (2025) https://pubs.acs.org/doi/abs/10.1021/acs.jced.5c00404
- Hongguang Kang, Xianwei Hu, Liang Tian, et al., Evaluation of NaCl-KCl-Al2S3 Molten Salts by Raman Spectroscopy and Quantum Chemical Calculations, Inorg. Chem. (2025) https://pubs.acs.org/doi/abs/10.1021/acs.inorgchem.5c01671
- Abhishek Sharma, Saurabh Shah, Suraj Wagh, et al., Ever-Increasing Role of Computational Tools in Solid-State Pharmaceutics: Advancing Drug Development with Enhanced Molecular Understanding and Risk Assessment, Mol. Pharmaceutics (2025) https://pubs.acs.org/doi/abs/10.1021/acs.molpharmaceut.5c00296
- Runjia Wang, Xin Wen, Sifan Du, et al., Unraveling Solvent-Dependent Chirality Inversion in the Self-Assembly of Glutamide Amphiphiles, Chem. Eur. J. (2025) https://chemistry-europe.onlinelibrary.wiley.com/doi/abs/10.1002/chem.202501991
- Jianfei Xu, Zhiwei Han, Yaning Li, et al., Quantum chemical study on the weak intermolecular interaction between DNTF and MW, J. Mol. Model. (2025) https://www.researchsquare.com/article/rs-7250076/v1
- 张晗阳, 胡宪伟, 康红光, et al., 粉煤灰酸浸液中AlCl3-FeCl3-FeCl2-HCl-H2O体系的离子结构研究, 湿法冶金, 44, 440 (2025) https://doi.org/10.13355/j.cnki.sfyj.2025.04.002 (此文没有按要求引用Molclus!)
- Wencong Fan, Xiangce Meng, Guobin Song, et al., Adsorption behaviors of typical phthalic acid esters on graphene oxide and the molecular mechanisms: Experiments and computational simulations, J. Mol. Struct. (2025) https://www.sciencedirect.com/science/article/abs/pii/S002228602502277X
- Yixin Sun, Baoyuan Zhang, Jiayao Wang, Robust antitumor treatment driven by lock-and-key biorecognition of dynamic cyclic diselenide-guided chiral prodrug self-assembly, Cell Chem. Biol. (2025) https://www.cell.com/cell-chemical-biology/abstract/S2451-9456(25)00226-0
- Mengtian Chen, Mengdi Yuan, Tong Bu, et al., The mechanism of weakening fat-induced matrix interference by deep eutectic solvents promoting fat phase transfer: A case of aflatoxin B1 detection by lateral flow immunoassay in 26 food matrices, Food Chem., 493, 145886 (2025) https://www.sciencedirect.com/science/article/abs/pii/S0308814625031371
- Ying Chen, Jianying Qu, Xinyi Li, et al., Hydrogen bond kinetics and their temperature dependence of biocompatibility-based choline amino acid ionic liquids, J. Mol. Graph. Model. (2025) https://www.sciencedirect.com/science/article/abs/pii/S1093326325001998
- Yuanzhu Mi, Yuqi Tang, Xuebing Jiang, et al., Synthesis of a demulsifier based on noncovalent interaction reconstruction and positive potential, Sep. Purif. Technol. (2025) https://www.sciencedirect.com/science/article/abs/pii/S1383586625032794
- Baocong Zhao, Luxin Ren, Sihao Lin, et al., Larger than expected organic acid yields from the multi-generation oxidation of petrochemical alkenes, EGUsphere (2025) https://doi.org/10.5194/egusphere-2025-3182
- Jian-Sen Wang, Xiao-Xia You, Zhong-Min Su, et al., Diastereomeric Iridium Catalysts for Enantioselective C(sp3)–H Borylation: Roles of Two Chiralities at the Metal Center and Ligand, J. Am. Chem. Soc. (2025) https://pubs.acs.org/doi/abs/10.1021/jacs.5c10820
- Liujun Pei, Jun Zhu, Danni Lu, et al., Influences of Commercial Dispersants on the Dyeing Performance and Dye–Fiber Interaction of Disperse Dyes in D5 Nonaqueous Medium Dyeing Systems, Langmuir (2025) https://pubs.acs.org/doi/abs/10.1021/acs.langmuir.5c02373 (此文没有按要求引用Molclus!)
- Jian Sun, Junmeng Luo, Ming Gao, et al., Quantum Chemistry Calculation-Assisted Large-Scale Collision Cross Section Prediction Empowers Derivatization-Enhanced Multidimensional Metabolomics, Angew. Chem. (2025) https://onlinelibrary.wiley.com/doi/abs/10.1002/ange.202507483 (此文没有按要求引用Molclus!)
- Fei Zhao, Shuqing Liu, Qinghua Liu, et al., Computational molecular engineering of ionic liquids for CO2 drying: Design strategies and molecular mechanisms, AIChE J. (2025) https://aiche.onlinelibrary.wiley.com/doi/abs/10.1002/aic.70014
- Kaining Mao, Chenfei Liu, Yi Wang, et al., Dynamic kinetic resolution of phosphines with chiral supporting electrolytes, Nature (2025) https://www.nature.com/articles/s41586-025-09238-x
- Qingzhi Lv, Yan Cui, Shuai Li, Phase behavior and computational thermodynamics of salt solutions in organic solvents, Chem. Eng. Sci., 319, 122250 (2025) https://www.sciencedirect.com/science/article/pii/S0009250925010711
- Wei Hou, Xinyu He, Chao Guo, et al., Molecular Insights into Ionic Liquids-Based Ethylene/Ethane Separation and Pressure Effects on Extractive Distillation: Cooling Medium and Economics, ACS Sustainable Chem. Eng. (2025) https://pubs.acs.org/doi/abs/10.1021/acssuschemeng.5c05218
- Ying yang, Yiming Ma, Guobin Ren, et al., Additive-mediated Crystallization of Metastable Fluxapyroxad Polymorph: Characterization, Structure, and Theoretical Insights, Cryst. Growth Des. (2025) https://pubs.acs.org/doi/abs/10.1021/acs.cgd.5c00507
- Hongguang Kang, Xianwei Hu, Ruidong Guo, et al., Cathodic process in NaCl-KCl-Al2S3 molten salts, J. Elect. Chem., 995, 119333 (2025) https://www.sciencedirect.com/science/article/abs/pii/S1572665725004072
- Yanrong Jiang, Wenjin Cao, Xiao-Fei Gao, et al., From Electronic Structure to Ion Transport: Photoelectron Spectroscopy and Molecular Dynamics Simulations Reveal the Role of Anions in Lithium Battery Electrolytes, J. Phys. Chem. A (2025) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.5c03415
- Xiaorui Fan, Theoretical Studies on the Interaction between 2-Aminobenzothiazole and Water, Russ. J. Phys. Chem., 99, 1566 (2025) https://link.springer.com/article/10.1134/S0036024425700943
- Hanwen Yan, Xiaoyan Zheng, Yuan Tian, et al., Machine learning-guided identification of critical descriptors of hydration-driven structural transitions in ionic liquids, Chem. Eng. Sci., 318, 122183 (2025) https://www.sciencedirect.com/science/article/abs/pii/S0009250925010048
- Yuting Wan, Hang Luo, Zhongna Yan, et al., Decoupling thermal stability and insulation in dielectric polymers via donor-acceptor rearrangement, Nat. Commun., 16, 6242 (2025) https://www.nature.com/articles/s41467-025-61539-x
- Kang Hongguang, Hu Xianwei, Meng Qingling, Evaluation of Na3AlF6-LiF-Al2S3 Molten Salts by Raman Spectroscopy and Quantum Chemical Calculations, J. Sust. Metal. (2025) https://link.springer.com/article/10.1007/s40831-025-01176-2
- Zhe Zheng, Hanyu Lv, Ruina Liu, et al., Internal Cavity versus External Cavity: A Theoretical Study of Molecular Recognition of Biphenarene, J. Phys. Chem. A, 129, 5799 (2025) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.5c02267
- Han Xie, Wei-Qi Wang, Yan Liu, et al., Chiral Cryptographic Key-Based Fluorescence Encryption Enabled by Stimuli-Responsive Metal-Organic Frameworks, Angew. Chem. Int. Ed. (2025) https://onlinelibrary.wiley.com/doi/abs/10.1002/anie.202510219
- Wenwen Zhang, Tai-Ping Zhou, Wenjing Zou, et al., Formation of β-cyclodextrin inclusion complexes with a series of structurally related parabens: Preparation, physicochemical characterization and antifungal properties, Carbohyd. Polym. Technol. Appl., 11, 100924 (2025) https://www.sciencedirect.com/science/article/pii/S2666893925002658
- Xiaoyi Gao, Bin Xu, Wufeng Jiang, et al., Phase separation prediction modeling and structure-activity relationship of CO2 capture by biphasic amine absorbents based on phase separation load, Chem. Eng. J., 519, 165311 (2025) https://www.sciencedirect.com/science/article/abs/pii/S1385894725061479
- Bingqing Sun, Ran Ding, Chuan Liu, et al., Sensing Mechanism of a Pyrene-Based Fluorescence Probe for TNP: Invalidity of Hydrogen Bond-Assisted Charge Transfer, J. Phys. Chem. A (2025) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.5c02704
- Rong Gu, Da Zhang, Shengtao Xu, et al., Thermoresponsive ether-based electrolyte for wide temperature operating lithium metal batteries, Nat. Commun., 16, 5474 (2025) https://www.nature.com/articles/s41467-025-60524-8 (此文没有按要求引用Molclus!)
- Yan Zhang, Xiaowei Chen, Wei Li, et al., Construction of Stretchable, Tough and Damped Eutectogel Resistant to −100 °C using 2-Methyl-1,3-Propanediol Based Deep Eutectic Solvent, Adv. Funct. Mater. (2025) https://advanced.onlinelibrary.wiley.com/doi/abs/10.1002/adfm.202511554
- Chao-Tun Cao, Naichen Song, Lanyu Zhang, Chenzhong Cao, The concept of instantaneous supramolecules and its application to simulating the thermal conductivity of water-ethylene glycol mixtures, J. Mol. Liq. (2025) https://www.sciencedirect.com/science/article/abs/pii/S0167732225011912
- Jiaming Li, Youjian Yang, Jiaxin Yang, et al., Study on the dissolution behavior and mechanism of LFP in the cryolite molten salt system of NaF-AlF3, Sep. Purif. Technol. (2025) https://www.sciencedirect.com/science/article/abs/pii/S1383586625026577
- Zhongfei Shang, Meimei Wang, Jiguang Li, et al., Exactly Matching the FRET Energy of the Functionalized Upconversion Nanoprobe to Boost the Sensing Performance toward Perchlorate, Anal. Chem. (2025) https://pubs.acs.org/doi/abs/10.1021/acs.analchem.5c01167
- Chuqiu Zhang, Haobo Chen, Yuanjing Zou, et al., Computationally-guided multifunctional nanoplatform for enhanced oral delivery and targeted therapy of Helicobacter pylori using herbal bioactives, Biomater. (2025) https://www.sciencedirect.com/science/article/abs/pii/S0142961225004363 (此文没有按要求引用Molclus!)
- Qian Teng, Qinghua Tan, Mengyue Hou, Fanglong Yuan, Thermally Activated Sensitized Fluorescence Enables Efficient Carbon Quantum Dot-Based Electroluminescent LEDs with Current Efficiency of 31 cd A−1, Angew. Chem. Int. Ed. (2025) https://onlinelibrary.wiley.com/doi/abs/10.1002/anie.202508650
- Jiahui Wei, Zexu Yan, Lingxiao Zhu, et al., Novel ternary deep eutectic solvent for simultaneous extraction of active compounds and cellulose nanofibers from rose petals, Int. J. Biol. Macromol., 318, 145264 (2025) https://www.sciencedirect.com/science/article/abs/pii/S0141813025058192
- Yi Zheng, Rongrong Shen, Yunuo Dai, et al., Hydrogen Bonds Mediating the Structural and Dynamic Behaviors in Choline-Phenolic Acid Ionic Liquids: A Computational Investigation, J. Phys. Chem. B (2025) https://pubs.acs.org/doi/abs/10.1021/acs.jpcb.5c01232
- Junyi Wo, Weijie Yang, Fullerene nano-confinement enables ultra-low dehydrogenation temperature of MgH2: A DFT-Based theoretical prediction, Int. J. Hyd. Energy, 146, 149987 (2025) https://www.sciencedirect.com/science/article/abs/pii/S0360319925029581
- Guo-Rui Zhu, Qin Zhang, Ying-Ying Zhang, et al., Soft-Hard Synergistic Solid All-Polymer Electrolyte Inspired by Musculoskeletal Structure for High-Temperature Lithium Metal Batteries, Adv. Funct. Mater. (2025) https://advanced.onlinelibrary.wiley.com/doi/abs/10.1002/adfm.202503481 (此文没有按要求引用Molclus!)
- Shirong Yan, Lu Qiao, Lei Chen, A recyclable type-I photosensitizer to enable red-light-driven gram-scale aerobic photocatalysis, Smart Mol., e20250003 (2025) https://doi.org/10.1002/smo.20250003
- Yunxiu Jia, Li Zhang, Weijiang Guan, et al., Ion-π Interactions Unveil a Universal Mechanism for Boosting Electrochemiluminescence, J. Phys. Chem. Lett., 16, 6023 (2025) https://pubs.acs.org/doi/abs/10.1021/acs.jpclett.5c01134
- Xin Liu, Yongbo Zhao, Huimin Guo, Changgong Meng, A Machine-Learning-Based Investigation on the Formation and Evolution of Silicic Acid Oligomers Structurally Analogous to Zeolite Building Units, J. Phys. Chem. Lett., 16, 5918 (2025) https://pubs.acs.org/doi/abs/10.1021/acs.jpclett.5c01376
- Zhoucheng Li, Zhutao Luo, Wentao Ma, et al., Synthesis of a demulsifier with concentrated hydrogen bonding sites and its demulsification mechanism, Sep. Purif. Technol., 376, 133888 (2025) https://www.sciencedirect.com/science/article/abs/pii/S1383586625024852 (此文没有按要求引用Molclus!)
- Waseem Akram, Xincheng Zhang, Zhenyu Wang, et al., Understanding the demulsification of highly waxy shale oil emulsions by oxygen-rich demulsifiers, Fuel, 401, 135766 (2025) https://www.sciencedirect.com/science/article/abs/pii/S0016236125014917
- Shaoqing Wang, Yan Huang, Qing Ma, Fu-Xue Chen, Theoretical research on tricyclic-based as high-energy performance energetic materials, J. Mol. Model., 31, 183 (2025) https://link.springer.com/article/10.1007/s00894-025-06401-z (此文没有按要求引用Molclus!)
- Abbas Ahmad Kassem, Chengmin Gui, Zhigang Lei, et al., Evaluation of nine candidate absorbents for VOC removal in biogas upgrading: Experimental and theoretical study, Sep. Purif. Technol., 375, 133865 (2025) https://www.sciencedirect.com/science/article/pii/S1383586625024621
- Duo Xu, Bowen Du, Xuan Jiang, et al., Sorption behavior of diclofenac sodium and venlafaxine on microplastics: a mechanistic study based on spectroscopy and density functional theory, J. Clean. Prod., 518, 145930 (2025) https://www.sciencedirect.com/science/article/abs/pii/S0959652625012806 (Multiwfn was not properly cited!)
- Abbas Ahmad Kassem, Chengmin Gui, Zhigang Lei, et al., Evaluation of nine candidate absorbents for VOC removal in biogas upgrading: Experimental and theoretical study, Sep. Purif. Technol., 375, 133865 (2025) https://www.sciencedirect.com/science/article/pii/S1383586625024621
- Rongzhen Zhang, Zhentao Jiang, Jingxin Rao, et al., Mechanistic Multi-Enzyme Engineering for High-Yield Bilirubin Biosynthesis, ResearchSquare (2025) https://www.researchsquare.com/article/rs-6554107/v1
- Zhonghua Hao, Wenna Ai, Neil Qiang Su, Unveiling the Delicate Balance of Polarity and Strain Effect in Determining the Site-Selective Intramolecular Hydrogen Atom Transfers, J. Phys. Chem. A (2025)https://pubs.acs.org/doi/10.1021/acs.jpca.5c00340
- Hao Zhang, Xiaoyan Zhuang, Yutong Wang, et al., Viscosity-thickening mechanism of polyethylene isobutyl ether in supercritical carbon dioxide, Coll. Surf. A, 723, 137346 (2025) https://www.sciencedirect.com/science/article/abs/pii/S092777572501249X
- Shuang Long Jiang, Qi Liang Lu, Qi Quan Luo, Structures and properties of M2Cn (M = Be, Mg; n = 8–20) clusters, Struct. Chem. (2025) https://link.springer.com/article/10.1007/s11224-025-02519-1
- Ya-ping Tao, Qi-feng Jiang, Xin-Hui Zhang, Ri-qing Liu, Effect of Fluorinated Substitution on the Properties of Phenylethylamine Derivatives: A Density Functional Theory Study, Russ. J. Gen. Chem., 95, 1104 (2025) https://link.springer.com/article/10.1134/S1070363225600882
- Wenlong Li, Yuwan Du, Yuan Liu, et al., Differentiation and identification of CA series synthetic cannabinoids based on SERS technology coupled with support vector machine, Sens. Actuat. B, 441, 138024 (2025) https://www.sciencedirect.com/science/article/abs/pii/S0925400525008007
- Ming Lin, Yihong Liu Jinzhao Guan, et al., Investigation of Structural Dynamics in KF-ZrF4 Molten Salt Systems via High-Temperature Raman Spectroscopy and Quantum Electrochemical Methods, Inorg. Chem. (2025) https://pubs.acs.org/doi/full/10.1021/acs.inorgchem.5c00861
- Hui Xu, Zhi-Yao Yang, Chao Tang, Wen-Yong La, Europium Coordinate and Covalent Derivatives of Phenanthroline for Intramolecular Carrier Dynamics, J. Phys. Chem. C (2025) https://pubs.acs.org/doi/abs/10.1021/acs.jpcc.5c01626 (此文没有按要求引用Molclus!)
- Dong Su, Xinyu Ma, Junkui Miao, et al., DNA enrichment using deep eutectic solvents for improved PCR detection in Antarctic krill oil, Sep. Purif. Technol. (2025) https://www.sciencedirect.com/science/article/abs/pii/S1383586625022488
- Quanjie Zhong, A length descriptor to measure the linear and third-order nonlinear optical responses of atomic-cluster isomers, Phys. Chem. Chem. Phys. (2025) https://pubs.rsc.org/en/content/articlehtml/2025/cp/d5cp00810g
- Yiming Lu, Weihong Shen, Zhong Li, et al., Structural Optimization of Aphid Repellents Based on the Low-Energy Conformation of (E)-β-Farnesene, J. Agric. Food Chem. (2025) https://pubs.acs.org/doi/abs/10.1021/acs.jafc.5c01913
- Zeyuan Chen, Junjun Zhao, Shuangfei Zhu, et al., Quantum chemistry and molecular dynamics study on the intermolecular interactions and stability of NL24 and MTNP complexes, Comput. Theor. Chem., 1250, 115282 (2025) https://www.sciencedirect.com/science/article/abs/pii/S2210271X2500218X
- Yongkang Cui, Dongfang Guo, Xinyue Chen, et al., The impact of directly introducing aromatic nitrogen heterocycles on the performance of CO2 capture by diethylenetriamine/diethylene glycol dimethyl ether biphasic solvents: experimental and theoretical analysis, Chem. Eng. J., 515, 163625 (2025) https://www.sciencedirect.com/science/article/abs/pii/S1385894725044596
- Xiao-Fei Gao, Wenjin Cao, Xue-Bin Wang, Probing Host–Guest Interactions of the Dual Anion Receptor Harmane with Halide and HSO4– Anions, J. Phys. Chem. A (2025) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.5c01523
- Qijiang Shu, Linjing Yang, Li Li, et al., Unveiling the mechanism of dodecylphosphorylcholine as an extremely promising drug delivery system: From self-assembly clusters to drug encapsulation pathways, PLOS One (2025) https://journals.plos.org/plosone/article?id=10.1371/journal.pone.0320737
- Peng Xiaogai, Hu Zhubin, Sun Haitao, et al., Theoretical Study on Dihydrogen Bond Interaction Dominated Dodecaborate-Solvent Molecular Clusters, Acta Chim. Sinica (2025) https://doi.org/10.6023/A25020041
- Zhao-Ou Gao, Zhen-Chao Long, Hong-Guang Xu, et al., Vibrationally Resolved Photoelectron Spectroscopy and Quantum Chemical Calculations of InCn– (n = 3–10) Clusters, J. Phys. Chem. A (2025) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.5c00496
- Jiarui Ma, Wenhai Zhang, Dan Li, et al., Highly separation of phenolics from coal tar by quaternary ammonium salt-based deep eutectic solvents, Sep. Purif. Technol. (2025) https://www.sciencedirect.com/science/article/abs/pii/S138358662501891X (此文没有按要求引用Molclus!)
- Chengmin Gui, Qinghua Liu, Zhigang Lei, et al., Supramolecular Deep Eutectic Solvents and Their Aqueous Solutions: Green Carriers for Essential Oils, ACS Sustainable Chem. Eng. (2025) https://pubs.acs.org/doi/abs/10.1021/acssuschemeng.5c00881
- Xuehui Zhang, Qing Sun, Yuan Chen, et al., Efficient extraction of essential oil compounds from agarwood using low eutectic solvents assisted by molecular simulation, J. Mol. Liq. (2025) https://www.sciencedirect.com/science/article/abs/pii/S0167732225008554 (此文没有按要求引用Molclus!)
- Yankai Wang, Biao Hong, Baozang Yin, et al., Engineering Antibacterial, Biocompatible, Anti-Oxidant Titanium-Based Implants Using Polyphenols-Chlorhexidine Networks for Bone Regeneration, Adv. Healthcare Mater. (2025) https://advanced.onlinelibrary.wiley.com/doi/abs/10.1002/adhm.202404958 (此文没有按要求引用Molclus!)
- Zhichao Gong, Guangbo Kang, Yu Cao, et al., Flexible Regulation of Optical Properties Based on Structure Size-Driven Intermolecular Interactions for Phototherapy, Adv. Sci., 2025, 2501468 (2025) https://doi.org/10.1002/advs.202501468
- Qiaojing Zhao, Fangfang Ma, Hui Zhao, et al., Acid-catalyzed hydrolysis kinetics of organic hydroperoxides: Computational strategy and structure-activity relationship, EGUsphere Preprint (2025) https://doi.org/10.5194/egusphere-2025-1662
- Ao You, Yuhui Li, Xinlang Yang, et al., Conformational equilibrium of aminoethanol in the gas phase and in solution driven by intra- and intermolecular interactions and solvent polarity, J. Chem. Phys., 162, 164309 (2025) https://pubs.aip.org/aip/jcp/article/162/16/164309/3345102
- Manzhen Li, Likang Lu, Yaoyao Guo, Self-assembly of paclitaxel derivative and fructose as a potent inducer of immunogenic cell death to enhance cancer immunotherapy, Mater. Today Bio, 32, 101793 (2025) https://doi.org/10.1016/j.mtbio.2025.101793
- Hao Qi, Kai Qi, Yan Wang, et al., Steric Complementarity Drives Strong Co-Assembly of Short Peptide Stereoisomers, J. Am. Chem. Soc. (2025) https://pubs.acs.org/doi/abs/10.1021/jacs.4c17497
- Yan Wang, Yurong Zhao, Henghao Yu, et al., Chirality Inversion upon Coassembly of Stereoisomeric Short Peptides with Like-Handedness, ACS Nano (2025) https://pubs.acs.org/doi/abs/10.1021/acsnano.5c02425
- Haonan Cheng, Wenbo Wang, Yun Zeng, et al., Thermal Cα–C6 Cyclization of Enediynes, J. Org. Chem. (2025) https://pubs.acs.org/doi/abs/10.1021/acs.joc.4c03124
- Hsiao-Yang Hsi, George Hsiao, Shih-Wei Wang, et al., Polyketides from a marine medicinal brown alga-derived fungal strain Hypomontagnella monticulosa SC1047, Phytochem., 236, 114508 (2025) https://doi.org/10.1016/j.phytochem.2025.114508
- Zhongxiao Sun, Zhiyong Xu, Jinyu Wang, et al., Temperature optimal control and entropy–enthalpy compensation phenomenon in the CO2 capture with DEA/PEG200 as absorbent, AIChE J. (2025) https://aiche.onlinelibrary.wiley.com/doi/abs/10.1002/aic.18857
- Chengmin Gui, Minghao Song, Qinghua Liu, et al., Rational screening of deep eutectic solvents for the removal of halogenated volatile organic compounds, AIChE J. (2025) https://aiche.onlinelibrary.wiley.com/doi/abs/10.1002/aic.18858
- Xiaohong Yang, Lin Chen, Guangyu He, et al., Chitosan-loaded coconut fiber for highly-enhanced heavy metal adsorption from wastewater, Sep. Purif. Technol. (2025) https://doi.org/10.1016/j.seppur.2025.133105 (此文没有按要求引用Molclus!)
- Luis A. Rios, Melanny J. Barraza, Pedro A. Robles, Gonzalo R. Quezada, Chalcopyrite Flotation, Molecular Design and Smart Industry: A Review, Int. J. Mol. Sci., 26, 3613 (2025) https://doi.org/10.3390/ijms26083613
- Da Zhu, Yu Ou, Yingchun Xia, et al., Design Rules for Selecting Suitable Weakly Solvating Electrolytes for Lithium Metal Batteries, J. Phys. Chem. Lett., 16, 3879 (2025) https://pubs.acs.org/doi/full/10.1021/acs.jpclett.5c00627
- Yeqiu Zhou, Feiwu Chen, Yu Zhou, Studies on the Deviation of a Solution from the Hypothetical Ideal Solution with the Total Activity Coefficient, Molecules, 30, 1681 (2025) https://doi.org/10.3390/molecules30081681
- Xinying Li, Yangyang Jiang, Yueyang Cheng, et al., A systematic entrainer screening and mechanism analysis method based on DFT for methanol/methyl propionate azeotrope separation, Sep. Purif. Technol. (2025) https://www.sciencedirect.com/science/article/pii/S1383586625015254
- Junfeng Qi, Chanjuan Hu, Jiayi Li, et al., Vancomycin-bacterial imprinted polymer hybrid for viable Staphylococcus aureus highly efficient capture, photothermal inactivation, and sensitive detection, Food Chem. (2025) https://www.sciencedirect.com/science/article/abs/pii/S030881462501475X
- Jiahao Dong, Chuanfang Zhao, Jinliang Ning, et al., Noncovalent Interaction-Based Probe Design for PET-Facilitated Fluorescence Sensing of Synthetic Cannabinoids, ACS Omega (2025) https://pubs.acs.org/doi/full/10.1021/acsomega.5c00540
- Yue Yuan, Zuopeng Li, Haji Akber Aisa, Targeted characterisation of bioactive prenylated flavonoids from Ficus carica L. fruits, Food Chem. (2025) https://www.sciencedirect.com/science/article/pii/S0308814625014529
- Gui-Xin Yan, Er-Xia Chen, Jin-Xia Yang, et al., Organotin-Oxo Clusters with Enhanced π-Conjugation for Iodine Capture, Inorg. Chem. (2025) https://pubs.acs.org/doi/abs/10.1021/acs.inorgchem.4c05502
- Xi-Long Li, Zhen-Chao Long, Kai-Wen Liu, et al., Size-Selected Anion Photoelectron Spectroscopy and Density Functional Theory Calculations of Ferromagnetic Fe2Cn−/0 (n = 2–6) Clusters, Phys. Chem. Chem. Phys. (2025) https://pubs.rsc.org/en/content/articlelanding/2025/cp/d5cp00662g/
- Wenjin Cao, Xue-Bin Wang, Electronic Instability and Solvation Stabilization of Oxocarbon Dianions (CnOn)2– (n = 4–6), J. Phys. Chem. A (2025) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.5c01355
- Chenzhou Wang, Yangyue Wei, Mingjin Wang, et al., Deep eutectic solvent pretreatment for improving lignin properties and subsequent 4-vinylphenol production: an integrated experimental and modeling investigation, Green Chem. (2025) https://pubs.rsc.org/en/content/articlelanding/2025/gc/d4gc06194b/
- Xing Zhao, Guangfeng Zhou, YiHao Geng, et al., A highly selective copper-tin alloy nanosheet/carbon-paper sensitive electrode for detecting nitrite at low electrochemical reduction potential based on a binary cooperative strategy, Chem. Eng. J. (2025) https://www.sciencedirect.com/science/article/pii/S1385894725030256 (此文没有按要求引用Molclus!)
- Dingkai Hu, Nuerbiya Yalikun, Dezhi Cao, et al., The hydrogen bond-driven mechanism for carbazole extraction: Experimental and theoretical analysis of alcohol-based deep eutectic solvents, J. Environ. Chem. Eng., 13, 116451 (2025) https://www.sciencedirect.com/science/article/pii/S2213343725011479
- Yongqiang Cheng, Yangfan Zhou, Hongwei Kang, et al., Vapor-liquid phase equilibrium study and computational thermodynamics of mixed inorganic salt aqueous solution, Chem. Eng. Sci. (2025) https://doi.org/10.1016/j.ces.2025.121608
- Xinlong Li, Jianglian She, Meiqun Cai, et al., New Polyketides from a Marine Sponge-Derived Fungus, Neopestalotiopsis sp., with Anti-Renal Fibrosis Activity, Mar. Drugs, 23 , 148 (2025) https://doi.org/10.3390/md23040148 (此文没有按要求引用Molclus!)
- Qingchao Fan, Xin Jiang, Yingchao Zhang, et al., 3D Printable Supramolecular Viologen-Cationic Polyurethane Ionotronics for Multimodal Sensing and Displays, Small (2025) https://onlinelibrary.wiley.com/doi/abs/10.1002/smll.202412798 (此文没有按要求引用Molclus!)
- Qian Liu, Kunchi Xie, Jie Cheng, et al., Rational screening-validation and mechanism analysis of ionic liquids for peeling cathode materials off aluminum foil from end-of-life lithium-ion batteries, Chem. Eng. J., 511, 161975 (2025) https://www.sciencedirect.com/science/article/abs/pii/S1385894725028013
- Gang Hu, Qiang Lu, Xiaolu Mao, et al., Enantioselective extraction of ibuprofen enantiomers using quinine-terminated tetra-arm PEGs as a recyclable chiral selector, Sust. Chem. Pharm., 45, 102009 (2025) https://www.sciencedirect.com/science/article/abs/pii/S235255412500107X
- Qiuhao Yi, Siyang Ye, Haiming Wu, et al., Gas-phase synthesis and soft-landing deposition of imidazole clusters with narrowband emission, Sci. China Chem. (2025) https://link.springer.com/article/10.1007/s11426-024-2544-y
- Qingyun Peng, Yuxiu Ye, Qinlin Cao, et al., Prenylated xanthones from the coral-derived fungus Aspergillus stellatus SCSIO41406 and their antibacterial activities, Nat. Prod. Res. (2025) https://www.tandfonline.com/doi/abs/10.1080/14786419.2025.2480663
- Yameng Xie, Zhi Wang, Qi Liu, et al., Antioxidants as coenzymes modulating the peroxidase-like activity of Ag6 clusters for the specific recognition of themselves, Chem. Eng. J. (2025) https://www.sciencedirect.com/science/article/abs/pii/S1385894725025835
- Zhao-Ou Gao, Xi-Ling Xu, Umar Farooq, et al., The structural resemblance between InSin − and Sin+1 (n = 3– 11): Anion photoelectron spectroscopy and density functional calculations, J. Chem. Phys. 162, 114302 (2025) https://doi.org/10.1063/5.0253378
- Yaping Liu, Chao Pan, Xianjie Li, et al., Efficient conversion of HMF to FDMC utilizing gold-based poly(ionic liquid)s with bis(trifluoromethylsulfonyl)imide anion, Chem. Eng. Sci. (2025) https://www.sciencedirect.com/science/article/abs/pii/S0009250925003641 (此文没有按要求引用Molclus!)
- Xu-Xin Zhu, Yao-Yue Fan, Cheng-Yu Zheng, et al., Alstolarsines A–E, Indoline Alkaloids with Two Different Carbon Skeletons from Alstonia scholaris, J. Nat. Prod. (2025) https://pubs.acs.org/doi/abs/10.1021/acs.jnatprod.5c00027 (此文没有按要求引用Molclus!)
- Ming Li, Yunying Lian, Yuying Gao, et al., Surface and texture-modified fillers and stable crosslinking agents synergistically achieve high-performing polyester-based composite polymer electrolytes for lithium−oxygen batteries, J. Power Sources, 640, 236773 (2025) https://www.sciencedirect.com/science/article/abs/pii/S0378775325006093
- Jiajia He, Xingjie Luo, Si-Qiang Fang, et al., Mechanistic study on enantiodivergent kinetic resolution of axial chiral binaphthol via peptide-phosphonium salt-catalyzed Atherton-Todd reaction, Org. Chem. Front. (2025) https://pubs.rsc.org/en/content/articlehtml/2025/qo/d5qo00119f
- Huahua Fan, Runjia Wang, Li Zhang, Minghua Liu, Amplified circularly polarized luminescence in assembly of terpyridine-ZnII and GTP induced by silver(I) ion, Sci. China Chem. (2025) https://link.springer.com/article/10.1007/s11426-024-2571-3
- Xiaoyu Li, Simiao Huang, Xinyu Wu, et al., Reactive amino acid-derived deep eutectic solvents for tailored lignin modification, Int. J. Biol. Macromol., 307, 142003 (2025) https://www.sciencedirect.com/science/article/abs/pii/S0141813025025541 (此文没有按要求引用Molclus!)
- Xuekun Xue, Lingling Hu, Yue Liu, Quantum chemical calculation of maximum absorption wavelength based on dispersed dye structure, J. Eng. Fibers Fabrics, 20, 1 (2025) https://journals.sagepub.com/doi/pdf/10.1177/15589250251324370
- Zefang Chen, Lin Du, Victor Fung, et al., B-Modified Pd Cathodes for the Efficient Detoxification of Halogenated Antibiotics: Enhancing C–F Bond Breakage beyond Hydrodefluorination, Environ. Sci. Technol. (2025) https://pubs.acs.org/doi/full/10.1021/acs.est.4c12635
- Xiao Li, Ziyang Liu, Minqiang Mai, et al., High-Efficiency and Stable Tandem Organic Light-Emitting Diodes Based on In Situ Coordination-Activated N-Doping, Adv. Funct. Mater. (2025) https://advanced.onlinelibrary.wiley.com/doi/abs/10.1002/adfm.202500409
- Liyao Ying, Zhaoyang Ju, Chunjin Lin, et al., Theoretical study on the alkyl chain length impact of azobenzene-based photoresponsive ionic liquids, BMC Chem., 19, 66 (2025) https://bmcchem.biomedcentral.com/articles/10.1186/s13065-025-01433-1
- Chao Sun, Feng Xue, Peizhe Cui, et al., Comparison of deep eutectic solvents and ionic liquids effects on the separation of Isopropyl alcohol–Isopropyl acetate based on experiments and molecular simulations, J. Environ. Chem. Eng., 13, 116095 (2025) https://www.sciencedirect.com/science/article/pii/S2213343725007912
- Ashenafi Emiru Teka, Bingzhi Liu, Yushen Yu, et al., Elucidating reaction pathways and kinetic modeling in ozone-assisted low-temperature oxidation of n-pentanol, Combust. Flame, 275, 114098 (2025) https://www.sciencedirect.com/science/article/abs/pii/S0010218025001361
- Jun-Wei Zha, Yuting Wan, Hang Luo, et al., Breaking the adverse correlation between thermal stability and electrical insulation in polymer dielectrics by rearranging the donor and acceptor units, ResearchSquare (2025) https://www.researchsquare.com/article/rs-6051609/v1
- Mingxia Hu, Yanhui Liu, Guanjun Wang, Yanying Zhao, Mass-Selected Infrared Photodissociation Spectroscopic and Theoretical Insights into Nitrobenzene Dimer Anion Tagged by Argon, J. Phys. Chem. A (2025) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.4c06875
- Yufan Yuan, Danping Li, Huajun Huang, et al., Direct photodegradation of aromatic carbamate pesticides: Kinetics and mechanisms in aqueous vs. non-aqueous media, J. Hazard. Mater., 489, 137648 (2025) https://www.sciencedirect.com/science/article/pii/S030438942500562X
- Shinan Liu, Houzhi Wang, Jun Yang, Investigation of equilibrium and convergence in MD simulations of asphalt system, Const. Build. Mater., 468, 140317 (2025) https://www.sciencedirect.com/science/article/abs/pii/S0950061825004659
- Haijun Fu, Zhendong Li, Yanying Zhao, Jingbai Li, Interplays between Functional Groups and Substitution Sites Modulate the Photophysics of the Bithiophenes, J. Phys. Chem. A (2025) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.4c08513
- Huanhuan Wang, Xiaobin Shan, Fuyi Liu, et al., Mechanistic Insights Into the Dissociative Photoionization Pathways of Ethyl Propionate: A Combined Experimental and Theoretical Approach, J. Mass Spectromet. (2025) https://analyticalsciencejournals.onlinelibrary.wiley.com/doi/abs/10.1002/jms.5119
- Shujun Tan, Hairong Wang, Zijun Mao, et al., Supramolecular room-temperature flowable lignin (RTFL)/MXene for sustainable coatings, Int. J. Biol. Macromol., 305, 141120 (2025) https://www.sciencedirect.com/science/article/abs/pii/S0141813025016691 (此文没有按要求引用Molclus!)
- Peng Jiang, Yu-meng Luo, Xin-yuan Li, et al., Discovery of schinortriterpenoids schisaterpenes O–R from the fruits of Schisandra chinensis via molecular networking, J. Mol. Struct., 1333, 141765 (2025) https://www.sciencedirect.com/science/article/abs/pii/S002228602500451X
- Jin Chen, Xixi Zeng, Ling Chen, Dissociation and aggregation behaviors of starch in choline amino acid ionic liquid solvents: the anion structure effect, Phys. Chem. Chem. Phys. (2024) https://pubs.rsc.org/en/content/articlehtml/2024/cp/d4cp03626c
- Pengfei Wang, Mingdong Shang, Kai Chen, et al., Ionic liquid-assisted extraction for separation of n-hexane and ethyl acetate: Experimental and process performance evaluation, Sep. Purif. Technol. (2025) https://www.sciencedirect.com/science/article/abs/pii/S1383586625006112
- Rong-Rong Du, Wen-Jie Qin, Xiu-Mei Duan, et al., Megastigmane glycosides from Heterosmilax yunnanensis and their neuroprotective activity, Phytochem. (2025) https://www.sciencedirect.com/science/article/abs/pii/S003194222500055X (此文没有按要求引用Molclus!)
- Wanting Wang, Yuke Zuo, Ziqing Xi, et al., Efficient molecular recognition of polycyclic aromatic hydrocarbons by X-diimide-pillar[4,6]arenes, Comput. Theor. Chem., 1246, 115127 (2025) https://www.sciencedirect.com/science/article/pii/S2210271X25000635
- Chenzhou Wang, Yiye Lu, Yangyue Wei, et al., Mechanistic insights into the role of deep eutectic solvent toward enhanced production of monophenols from bagasse lignin, Int. J. Biol. Macromol. (2025) https://www.sciencedirect.com/science/article/pii/S0141813025013170
- Shini Yang, Peiqing Yang, Wenxin Wang, et al., A normalization strategy of cyanidin-3-diglucoside-5-glucoside recovery from red cabbage byproduct, J. Cleaner Prod. (2025) https://www.sciencedirect.com/science/article/pii/S0959652625003154 (此文没有按要求引用Molclus!)
- Qingyun Peng, Wenjia Huang, Xiao Zhang, et al., Structurally Diverse New Metabolites from Three Hadal Trench-Derived Microorganisms, ACS Omega (2025) https://pubs.acs.org/doi/full/10.1021/acsomega.4c11083
- Junhao Long, Jiayue Lu, Liheng Chen, et al., Lignin Ultrafiltration Fractionation and Self-Assembly to Monodisperse Nanoparticles for Photonic Materials, ACS Omega (2025) https://pubs.acs.org/doi/full/10.1021/acsomega.4c11260
- Meifang Chen, Shengrui Tong, Shanshan Yu, et al., Impact of the Criegee Intermediate on the Formation of Secondary Organic Aerosols during E-4-Hexen-1-ol Ozonolysis, J. Phys. Chem. A (2025) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.4c08028
- Yihao Li, Xiaoxuan Zhang, Jiali Xie, et al., Analyzing the Linker Structure of PROTACs throughout the Induction Process: Computational Insights, J. Med. Chem. (2025) https://pubs.acs.org/doi/abs/10.1021/acs.jmedchem.4c02637
- Shifan Xu, Hui Wang, Hongtu Zhao, et al., Revealing and visualizing the microscopic solution chemical molecular mechanism of β-HMX solubility in binary mixed solvents, Chem. Eng. Sci. (2025) https://www.sciencedirect.com/science/article/abs/pii/S0009250925001113
- Yifan Shang, Siqi Li, Xiaopeng Zhang, et al., A Novel Chrysene-Anthracene Host Material for Enhanced Efficiency and Lifetime in Blue Fluorescent OLEDs, Adv. Opt. Mater. (2025) https://onlinelibrary.wiley.com/doi/abs/10.1002/adom.202402893
- Jie Ma, Ping Zhou, Yong Liu, et al., Humic Acid with Vertical Adsorption Conformation Enhanced the Transport of Petroleum Hydrocarbon-Contaminated Colloids, Environ. Sci. Technol. (2025) https://pubs.acs.org/doi/abs/10.1021/acs.est.4c11233
- Qian Liu, Wei Meng, Yuxin Qiu, et al., Multilevel screening and mechanism analysis of ionic liquids for separating pyridine from coal pyrolysis model oil, Sep. Purif. Technol., 362, 131856 (2025) https://www.sciencedirect.com/science/article/abs/pii/S1383586625004538
- Tao Xiong, Yingchao Chen, Qiang Peng, et al., Heterodimeric Photosensitizer as Radical Generators to Promoting Type I Photodynamic Conversion for Hypoxic Tumor Therapy, Adv. Mater. (2025) https://advanced.onlinelibrary.wiley.com/doi/abs/10.1002/adma.202410992
- Jiamiao Wang, Jurong Lv, Zheyu Shen, et al., Development of rapid and data-driven detection methods for organotin compounds from microplastics: Integrating theoretical and experimental Raman and SERS database, Microchem. J. (2025) https://www.sciencedirect.com/science/article/pii/S0026265X25001900
- Fei Zhao, Yongqiang Cheng, Ruisong Zhu, et al., Computer-aided ionic liquids design and molecular insight for chloromethane dehydration, AIChE (2025) https://aiche.onlinelibrary.wiley.com/doi/abs/10.1002/aic.18737
- Zhenghuai Feng, Miao Xie, Jialiang Lai, et al., Stereochemistry-Tuned Hydrogen-Bonding Synergistic Covalent Adaptable Networks: Towards Recycled Elastomers with Excellent Creep-Resistant Performance, Angew. Chem. (2025) https://onlinelibrary.wiley.com/doi/abs/10.1002/ange.202423712
- Shuang Zhang, Dongling Wu, Qian Liu, et al., Deep eutectic solvent induced silver-gel as a flexible SERS substrate for sensitive detection of antibiotics under low temperature conditions, J. Hazard. Mater. (2025) https://www.sciencedirect.com/science/article/abs/pii/S0304389425001918 (此文没有按要求引用Molclus!)
- Zhiyu Chen, Guan-Wen Yang, Tianhao Wu, et al., Dual Kinetic Control of Polycarbonate Sequences via Breaking Catalysis Symmetry Using Dual Biomimetic Organoboron Catalysts, Macromolecules (2025) https://pubs.acs.org/doi/abs/10.1021/acs.macromol.4c02912
- Hang Yang, Yutie Bi, Yan Gao, et al., Theoretical and Experimental Investigation of Blue-emitting Plastic Scintillator with Fast-timing Property, New J. Chem. (2025) https://pubs.rsc.org/en/content/articlelanding/2025/nj/d4nj04728a/
- Huan An, Asiya, Gulmire Yaermaimaiti, et al., Theoretical study on the conformation-dependent charge transfer of the excited state of dopamine, Heliyon, 11, e42058 (2025) https://www.sciencedirect.com/science/article/pii/S2405844025004384
- Qinghua Liu, Minghao Song, Fei Zhao, et al., Experimental and molecular thermodynamics insights into separating bicyclic aromatics from diesel oil with ionic liquids, AIChE (2025) https://aiche.onlinelibrary.wiley.com/doi/abs/10.1002/aic.18729
- Shenglin Wang, Jiahui Wei, Hanwen Ge, et al., Molecular simulation and machine learning assisted in exploring betaine-based deep eutectic solvent extraction of active compounds from peony petals, Sep. Purif. Technol. (2025) https://www.sciencedirect.com/science/article/abs/pii/S1383586625001479
- Yueyue Wang, Minming Yan, Haoyu Huang, et al., Nonpolar P-type Conjugated Small Molecules Enable High-Performance Organic Photodetectors for Potential Application in Optical Wireless Communication, Nano Lett. (2025) https://pubs.acs.org/doi/abs/10.1021/acs.nanolett.4c05826
- Xiang-Ming Zeng, Minjian Wu, Liao-Yuan Yao, Guo-Yu Yang, Dynamic Phosphorescence Behavior of Carbene-Metal-Amide Complexes from the Perspective of Excited State Modulation, Angew. Int. Ed. Chem. (2025) https://onlinelibrary.wiley.com/doi/abs/10.1002/anie.202419614
- Qi Zhang, Qiangqiang Zhao, Han Wang, et al., Tuning Isomerism Effect in Organic Bulk Additives Enables Efficient and Stable Perovskite Solar Cells, Nano-Micro Lett. (2025) https://link.springer.com/article/10.1007/s40820-024-01613-z
- Ruinan Zhang, Yandong Guo, Bobo Cao, et al., Structure design of photosensitive ionic liquid for CO2 capture, Chem. Eng. Sci. (2025) https://www.sciencedirect.com/science/article/abs/pii/S0009250925000156
- Miaomiao Chen, Jiabin Yin, Hanyu Wu, Unveiling the Preference for a Carbon Spacer Length of Three in Zwitterionic Sulfobetaines: Insights from DFT Calculations, Langmuir (2024) https://pubs.acs.org/doi/abs/10.1021/acs.langmuir.4c04179
- Jinping Ke, Jiawei Yuan, Zunting Pang, et al., Piperine as a molecular bridge mediates a ternary coamorphous system of polyphenols with enhanced pharmaceutical properties, Int. J. Pharm. (2024) https://www.sciencedirect.com/science/article/pii/S0378517324013735 (此文没有按要求引用Molclus!)
- Xiaoxiao Liang, Shiyuan Liu, Long Deng, et al., Study on the formation mechanism and effective manipulation of polymorphs and solvates in Osimertinib-Caffeic acid multi-component crystal with distinct properties, Int. J. Pharm. (2024) https://www.sciencedirect.com/science/article/abs/pii/S0378517324013796
- Shaofen Yu, Yamin Song, Zhuxiao Li, et al., Fluxional Doping-Si Manipulates Spin Coupling Characteristics in Silicon-Vacancy Defect Nanodiamond, J. Phys. Chem. C (2024) https://pubs.acs.org/doi/abs/10.1021/acs.jpcc.4c05614
- Wanxiang Zhang, Yangchangqing Zhao, Bingru Wang, et al., Efficient separation of oil–phenol mixtures and removal of neutral oil entrainment via an in situ deep eutectic method, Green Chem. (2025) https://pubs.rsc.org/en/content/articlelanding/2025/gc/d4gc05756b/
- Dongyang Li, Deshan Li, Jiabin Huang, et al., Conformational effects in the identification and quantification of ketohydroperoxides in the oxidation of n-pentane, Phys. Chem. Chem. Phys. (2024) https://pubs.rsc.org/en/content/articlehtml/2024/cp/d4cp04184d
- RunChuan Zhou, Ang Zhao, Jian Liu, et al., Effect of Internal Electron Donors on Butadiene Polymerization Catalyzed by the TiCl4/MgCl2 Ziegler–Natta Catalyst, J. Phys. Chem. C (2024) https://pubs.acs.org/doi/abs/10.1021/acs.jpcc.4c05978
- Wenjin Niu, Wen Nie, Qiu Bao, et al., Molecular mechanisms of coal dust wettability: Hydrophilic group modulation for enhanced surface interactions, Appl. Surf. Sci. (2024) https://www.sciencedirect.com/science/article/pii/S0169433224028666
- Chengmin Gui, Dong Xiang, Mengjing Feng, et al., Effective VOCs abatement using supramolecular deep eutectic solvents, Sep. Purif. Technol., 360, 131121 (2025) https://www.sciencedirect.com/science/article/pii/S1383586624048603
- Rui Wu, Shuai Yang, Naonao Zhang, Jixiong Xiao, Prediction on electrical insulation synergistic effect of gas mixture based on dimer microscopic descriptors, AIP Adv., 14, 125019 (2024) https://pubs.aip.org/aip/adv/article/14/12/125019/3325971
- Wang Haiou, Xu Yang, Sang Weihe, et al., Extractive rectification using DES for benzene-cyclohexane-cyclohexene ternary azeotropic system, J. Chem. Technol. Biotechnol. (2024) https://scijournals.onlinelibrary.wiley.com/doi/abs/10.1002/jctb.7774
- Camila M.A.C. Alves, Júlia F. Alves, Lucas L. Bezerra, et al., Adsorption of linear and cyclic pentasiloxanes onto MCM-41: Experimental and computational studies, Results Surf. Interf. (2024) https://www.sciencedirect.com/science/article/pii/S2666845924002022
- Abylay Katbashev, Marcel Stahn, Thomas Rose, et al., Overview on Building Blocks and Applications of Efficient and Robust Extended Tight Binding, ChemRxiv (2024) https://chemrxiv.org/engage/chemrxiv/article-details/6752dec4f9980725cf3ca59f
- Lihao Qu, Takuro Tsutsumi, Yuriko Ono, Tetsuya Taketsugu, Acceleration of Reaction Space Projector Analysis Using Combinatorial Optimization: Application to Organic Chemical Reactions, J. Chem. Theory Comput. (2024) https://pubs.acs.org/doi/abs/10.1021/acs.jctc.4c01072
- Shiwei Deng, Yiyang Wu, Zhuyifan Ye, Defang Ouyang, In silico prediction of metabolic stability for ester-containing molecules: Machine learning and quantum mechanical methods, Chemometr. Intell. Lab., 257, 105292 (2025) https://www.sciencedirect.com/science/article/pii/S0169743924002326
- Lin-Yan Bao, Jian-Sen Wang, Lu Li, et al., Theoretical Insight into the Multiple Roles of the Silyl-Phenanthroline Ligand in Ir-Catalyzed C(sp3)–H Borylation, J. Org. Chem. (2024) https://pubs.acs.org/doi/abs/10.1021/acs.joc.4c01779
- Leon de Villiers Engelbrecht, Narcis Cibotariu, Xiaoyan Ji, et al., Deep Eutectic Solvents Meet Non-Aqueous Cosolvents: A Modeling and Simulation Perspective─A Tutorial Review, J. Chem. Eng. Data (2024) https://pubs.acs.org/doi/abs/10.1021/acs.jced.4c00505
- Jin Li, Lei Wang, Huanhuan Gao, et al., Enhancing optoelectronic performance of organic solar cells: Alkoxy substitution of central cores and π-bridge units in fully non-fused ring electron acceptors, J. Mol. Liq. (2024) https://www.sciencedirect.com/science/article/pii/S0167732224027429
- Sourav Mandal, Arsene Kossov, Pierre Carcabal, Aloke Das, Effect of a single water molecule on the conformational preferences of a capped Pro–Gly dipeptide in the gas phase, J. Chem. Phys., 161, 214304 (2024) https://pubs.aip.org/aip/jcp/article-abstract/161/21/214304/3323343/Effect-of-a-single-water-molecule-on-the
- Qinghua Liu, Xin Guo, Yufeng Hu, et al., Functional ionic liquid anchored TS-1 molecular sieve with multiple adsorption sites for efficient capture and separation of CO2 in flue gas systems, Sep. Purif. Technol. (2024) https://www.sciencedirect.com/science/article/pii/S1383586624046598
- Danting Wang, Yuanyuan Wang, Rui Ma, et al., Efficient and green separation of phenolics by halogen free deep eutectic solvents, J. Mol. Liq. (2024) https://www.sciencedirect.com/science/article/pii/S0167732224026631 (此文没有按要求引用Molclus!)
- Shan LiuLei Wei, Tengwu Zeng, Wentao Jiang, et al., Single-Crystal Dynamic Covalent Organic Frameworks for Adaptive Guest Alignments, J. Am. Chem. Soc. (2024) https://pubs.acs.org/doi/abs/10.1021/jacs.4c13377
- Lijun Fang, ChenKai Liu, Yonghong Feng, et al., Size-dependent nanoconfinement effects in magnesium hydride, Int. J. Hyd. Energy, 96, 783 (2024) https://www.sciencedirect.com/science/article/pii/S0360319924049152
- Zhuqin Zhang, Yu Sun, Zhi Dang, Lijuan Zhang, Unraveling the mechanistic effects of oxidation and ionization on polydopamine-Pb(Ⅱ) interaction: MD and DFT study, J. Environ. Chem. Eng., 12, 114924 (2024) https://www.sciencedirect.com/science/article/pii/S2213343724030562
- Yangxia Han, Manchang Kou, Haixia Zhang, Yan-Ping Shi, Fabrication of Yellow-Emitting Chiral Silicon Nanoparticles and Fluorescence/Colorimetric Dual-Mode Recognition of Lysine Enantiomers together with Nanobioimaging, Anal. Chem. (2024) https://pubs.acs.org/doi/abs/10.1021/acs.analchem.4c04172
- Zhenxu Yang, Dingkai Hu, Qiang Wang, et al., Separation of Phenolic Compounds in Coal Tar by Using Deep Eutectic Solvent Extraction with Choline-Urea: Based on Phase Equilibrium Experiments and Molecular Mechanism Investigation, J. Chem. Eng. Data (2024) https://pubs.acs.org/doi/abs/10.1021/acs.jced.4c00382
- Ming Zhao, Tao Cheng, Tianyu Li, et al., Molecular polarity regulation of polybromide complexes for high-performance low-temperature zinc–bromine flow batteries, Energy Environ. Sci. (2024) https://pubs.rsc.org/en/content/articlelanding/2024/ee/d4ee04046e/
- Yingshuang Zhang, Yuwei Zhao, Zhibo Qian, et al., A hydrophilic/hydrophobic switch on polymer surface triggered by calcite towards separation of hazardous PVC from plastic mixtures, J. Hazard Mater. (2024) https://www.sciencedirect.com/science/article/pii/S0304389424032485
- Minhua Zhang, Tong Wu, Xinyue Guan, et al., The advancement of EVA polymerization kinetics and development of high-performance EVOH polymerization process, Chem. Eng. Sci., 303, 120988 (2025) https://www.sciencedirect.com/science/article/pii/S0009250924012880 (此文没有按要求引用Molclus!)
- Yuanbin Li, Wu Yi, Haowei Chen, et al., Enhancement of silymarin solubility and bioactivities using betaine/ascorbyl glucoside DES, New J. Chem. (2024) https://pubs.rsc.org/en/content/articlelanding/2024/nj/d4nj03748k/ (此文没有按要求引用Molclus!)
- Guangnan Wang, Feifei Liu, Fei Pan, et al., Study on the Interaction between Polyol Glycerol and Flavor Compounds of Baijiu: A New Perspective of Influencing Factors of Baijiu Flavor, J. Agric. Food Chem. (2024) https://pubs.acs.org/doi/abs/10.1021/acs.jafc.4c05935 (此文没有按要求引用Molclus!)
- Bo Han, Can Chen, Shuai Li, et al., Structure-performance relationship and molecular structure optimization design of acid phosphate ester extractants, Chem. Eng. J. (2024) https://www.sciencedirect.com/science/article/pii/S1385894724093513
- Xiujun Lin, Fangfang Liu, Zihan Ma, et al., Investigating the impact of beeswax addition and diacylglycerol profiles on bigel properties and application in bread: insights on intermolecular interaction mechanisms, Food Hydrocoll. (2024) https://www.sciencedirect.com/science/article/pii/S0268005X24011123 (此文没有按要求引用Molclus!)
- Hongpu Guo, Tongyan Liang, Shuhan Liu, et al., Phase equilibrium and separation process of byproduct vinyl acrylate in the ethylene vapor phase method producing vinyl acetate, Chem. Eng. J., 501, 157598 (2024) https://www.sciencedirect.com/science/article/pii/S1385894724090892
- Huajian Xu, Zhongtao Hu, Zongwei Hao, et al., Experimental and computational insights into starch pasting as influenced by amino acids with different R-groups, Food Chem. (2024) https://www.sciencedirect.com/science/article/pii/S0308814624036197
- Yuanhao Liao, Dongwei Sun, Xiaobo Tang, et al., Effect of structure and interaction on physicochemical properties of new [Emim][BF3X] complex anion ionic liquids studied by quantum chemistry, ResearchSquare (2024) https://doi.org/10.21203/rs.3.rs-5091979/v1 (此文没有按要求引用Molclus!)
- Shenglin Wang, Jiahui Wei, Hanwen Ge, et al., Mechanism of deep eutectic solvents based extraction of phenolic and flavonoid compounds from peony petals, Ind. Crops Prod., 222, 119947 (2024) https://www.sciencedirect.com/science/article/pii/S0926669024019241
- Qianwen Lu, Hengfeng Li, Zhijian Tan, Natural cellulose reinforced multifunctional eutectogels for wearable sensors and epidermal electrodes, Carbohyd. Polym. (2024) https://www.sciencedirect.com/science/article/pii/S0144861724011652
- Baobing Fan, Huanhuan Gao, Liyang Yu, et al., Local Structure-Induced Selective Interactions Enables High-Performance and Burn-in-Free Organic Photovoltaics, Angew. Chem. Int. Ed. (2024) https://onlinelibrary.wiley.com/doi/abs/10.1002/anie.202418439
- Lutong Yuan, Lixin Cui, Yuheng Liu, et al., Synthesis of Chiral Axially Diaryl Aldehydes by Chiral Phosphoric Acid Catalyzed Desymmetrization Reaction, Eur. J. Org. Chem. (2024) https://chemistry-europe.onlinelibrary.wiley.com/doi/abs/10.1002/ejoc.202401038
- Run Li, Xiandong Hao, Bo Wang, et al., Highly efficient CO2 capture by a non-aqueous amine-based absorbent of 3 aminopropanol/polyethylene glycol 200: Experimental and computational simulation, J. Mol. Liq. (2024) https://www.sciencedirect.com/science/article/pii/S016773222402436X
- Camila Botin Francisco, Fernanda Franco Dourado, Cleverton de Souza Fernandes, et al., The Role of Intra and Intermolecular Interactions on the Conformational Dynamics of 2-Halo-1-phenylpropanols: Structure and Solvent Effects, J. Mol. Struct. (2024) https://www.sciencedirect.com/science/article/pii/S0022286024030011
- Kai Luo, Chao Guo, Jiangchuan Liang, et al., Energy and cost-efficient ionic liquids extractive distillation for producing electronic and polymer-grade propylene with emphasis on feedstock variability, Sep. Purif. Technol., 358, 130258 (2025) pii/S1383586624039972
- Wen-Yue Lin, Hui-Ping Wen, Jia-Yao Li, et al., Compact Molecular Conformation of Prodrugs Enhances Photocleaving Performance for Tumor Vascular Growth Inhibition, Adv. Healthcare Mater. (2024) https://onlinelibrary.wiley.com/doi/abs/10.1002/adhm.202402690 (此文没有按要求引用Molclus!)
- Zhaohui Bai, Liang Xu, Yupei Zhao, et al., Effect of Solvents on the Performance and Structure of Polymethylacrylimide Aerogels, Langmuir (2024) https://pubs.acs.org/doi/abs/10.1021/acs.langmuir.4c03187
- Di Wu, Lin Fu, Hongxun Hao, et al., Effect of conformational landscape on the polymorphism and monomorphism of tizanidine cocrystallization outcomes, Int. J. Pharm. (2024) https://www.sciencedirect.com/science/article/pii/S0378517324010937
- Wanxiang Zhang, Lixia Ji, Yucui Hou, et al., Highly-efficient separation of pyromellitic acid and trimellitic acid mixtures via forming deep eutectic solvents: Experiment and calculation, Chin. J. Chem. Eng. (2024) https://www.sciencedirect.com/science/article/pii/S1004954124003306
- Yinzheng Li, Hao Ma, Caihua Wang, et al., Pretreatment of moso bamboo using urea/MEA solvent at mild conditions for improving enzymatic hydrolysis, Ind. Crops Prod., 222,119845 (2024) https://www.sciencedirect.com/science/article/pii/S0926669024018223
- Wei Chen, Han Xiao, Minyi Zhang, et al., Deciphering Surface Ligand Density of Colloidal Semiconductor Nanocrystals: Shape Matters, J. Am. Chem. Soc. (2024) https://pubs.acs.org/doi/abs/10.1021/jacs.4c09592
- Xiankai Jiang, Zishuo Wang, Changying Wang, Junjian Miao, The aromatic amino acid phenylalanine: a versatile tool for binding transition metal ions, J. Mol. Model. (2024) https://link.springer.com/article/10.1007/s00894-024-06175-w
- Meng Wang, Wangzhe Sun, Dongshuai Hou, et al., Stepwise modeling approach to explore the interfacial behavior of Ca(OH)2/Sulfate, J. Building Eng. (2024) https://www.sciencedirect.com/science/article/pii/S2352710224025737
- Yijian Zheng, Rongtong Wang, Wenjie Luo, et al., Gel-modified metal-phenolic networks membrane for emulsion separation with highly stable permeability and optimized mass transfer mode, Chem. Eng. Sci., 302, 120808 (2025) https://www.sciencedirect.com/science/article/pii/S0009250924011084
- Yihuan Zhou, Qiang Xie, Hongyang Zhou, et al., Effect of nitrogen- and oxygen-containing functional groups on adsorption of styrene by activated carbon: A theoretical study by density functional theory, Comput. Theor. Chem., 1241, 114896 (2024) https://www.sciencedirect.com/science/article/pii/S2210271X24004353
- Bumaliya Abulimiti, Huan An, Gulimire Yaermaimaiti, et al., Observation of reversible conformational interconversion accompanied by 3p internal conversions in Rydberg-excited N,N-dimethylethylamine, Spectrochim. Acta A (2024) https://www.sciencedirect.com/science/article/pii/S1386142524014458
- Yulin Min, Rong Gu, Da Zhang, et al., Novel thermoresponsive ether-based electrolyte for wide-temperature operating lithium metal batteries, ResearchSquare (2024) https://www.researchsquare.com/article/rs-5101221/v1 (此文没有按要求引用Molclus!)
- Pei Huang, Jialin Lin, Zian Wang, et al., Competitive Solvation Structures via Hetero-Salt Additive Strategy Enables Temperature-Adaptive Sodium-Ion Batteries, Adv. Funct. Mater. (2024) https://onlinelibrary.wiley.com/doi/abs/10.1002/adfm.202413302 (此文没有按要求引用Molclus!)
- Xiaoya Li, Qian Zhang, Weidong Wu, et al., Lignin-Based Visible Light-Triggered Nitric Oxide Nanogenerator for Antibacterial Applications, Biomacromolecules (2024) https://pubs.acs.org/doi/abs/10.1021/acs.biomac.4c00775
- Boris Borrisov, Giovanni M. Beneventi, Yubin Fu, et al., Deep-Saddle-Shaped Nanographene Induced by Four Heptagons: Efficient Synthesis and Properties, J. Am. Chem. Soc. (2024) https://pubs.acs.org/doi/abs/10.1021/jacs.4c09224
- Hua Yang, Yuerui Yang, Jiao Wang, et al., PVA-Stabilized and Coassembled Nano/Microparticles with High Payload of Dual Phytochemicals for Enhanced Antibacterial and Targeting Effect, ACS Omega (2024) https://pubs.acs.org/doi/full/10.1021/acsomega.4c06925
- Qing-Wei Meng, Jianguo Li, Zhuozhi Lai, et al., Optimizing selectivity via membrane molecular packing manipulation for simultaneous cation and anion screening, Sci. Adv. (2024) https://www.science.org/doi/full/10.1126/sciadv.ado8658
- Baoping Zhu , Hanwen Ge, Jiahui Wei, et al., Comprehensive analysis of lignin dimer dissolution microscopic mechanism in different aromatic-based deep eutectic solvent, Ind. Crops Prod., 222, 119736 (2024) https://www.sciencedirect.com/science/article/pii/S0926669024017138
- Yuchen Zhou, Ingso Limbu, Mary J. Garson, Elizabeth H. Krenske, Conformational Sampling in Computational Studies of Natural Products: Why Is It Important? J. Nat. Prod. (2024) https://pubs.acs.org/doi/abs/10.1021/acs.jnatprod.4c00852
- Rongzhen Ma, Lin Chang, Long D. Nghiem, et al., Ligand-to-Metal Charge Transfer Quenching of Carbon Dots for Highly Selective Hg2+ Detection in Microfluidic Devices, ACS Appl. Nano Mater. (2024) https://pubs.acs.org/doi/abs/10.1021/acsanm.4c04700
- Hao Hu, Peng Li, Yen Wah Tong, et al., Reutilization recovered nutrients from wastewater by synthesizing eco-friendly slow-release hydrogel fertilizer for sustainable vegetable production, Chem. Eng. J. (2024) https://www.sciencedirect.com/science/article/pii/S1385894724074345 (此文没有按要求引用Molclus!)
- Long Chen, Yu Huang, Yonggang Xue, Zhihui Jia, Molecular insights into the formation of Criegee intermediates from β-hydroxyperoxy radicals, Atmosph. Environ., 338, 120828 (2024) https://www.sciencedirect.com/science/article/pii/S135223102400503X
- Shijie Yin, Jianguo Li, Zhuozhi Lai, et al., Giant gateable thermoelectric conversion by tuning the ion linkage interactions in covalent organic framework membranes, Nat. Commun., 15, 8137 (2024) https://www.nature.com/articles/s41467-024-52487-z
- Xiao-Qin Lu, Jindong Li, Bin Wang, Shu Qin, Computational Insights into the Radical Scavenging Activity and Xanthine Oxidase Inhibition of the Five Anthocyanins Derived from Grape Skin, Antioxidants, 13, 1117 (2024) https://doi.org/10.3390/antiox13091117
- Yong Liu, Jie Ma, Bingcong Feng, et al., Effect of isomeric polysaccharides on heteroaggregation of nanoplastics in high ionic strength conditions: Synergies of morphology and molecular conformation, J. Hazard. Mater. (2024) https://www.sciencedirect.com/science/article/pii/S0304389424023975
- Hanwen Ge, Jiahui Wei, Baoping Zhu, et al., Microscopic mechanism investigation of extraction of lignin by benzyltriethylammonium chloride-based deep eutectic solvents, J. Mol. Liq., 413, 125996 (2024) https://www.sciencedirect.com/science/article/pii/S0167732224020555
- Yantao Liu, Diming Li, Junxian Hu, et al., New insight into synergistic interaction between the backbone or side chain of xanthan gum and the backbone of Gleditsia sinensis polysaccharide, Int. J. Biol. Macromol., 279, 135396 (2024) https://www.sciencedirect.com/science/article/pii/S0141813024062044
- Quyan Su, Jia Han, Haili Yu, et al., Computational insights into the underlying mechanism of zinc ion-specificity of the fluorescent probe, BDA-1, Spectrochim. Acta A, 322, 124854 (2024) https://www.sciencedirect.com/science/article/abs/pii/S1386142524010205
- Yanqiong Wang, Yaqi Hou, Fengwei Huo, Xu Hou, Fe3+ ion quantification with reusable bioinspired nanopores, Chin. Chem. Lett. (2024) https://www.sciencedirect.com/science/article/pii/S1001841724009471
- Lemin Chen, Jihuan Xie, Yichun Bi, et al., Facile synthesis of Fe3O4 photothermal nanoparticles coated with poly-3,4-dihydroxybenzaldehyde for improved photothermal therapy of cancer cells, Coll. Surf. A (2024) https://www.sciencedirect.com/science/article/pii/S0927775724021678 (此文没有按要求引用Molclus!)
- Yuan-Jun Gao, Wen-Kai Chen, Wei-Hai Fang, Ganglong Cui, A Green Host–Guest Protocol to Improve Water Solubility of Fluorescent Dyes, J. Phys. Chem. Lett., 15, 9380 (2024) https://pubs.acs.org/doi/abs/10.1021/acs.jpclett.4c02107
- Shujun Wang, Yue Xu, Yini Wang, et al., Solubilities measurement and molecular simulation of (R,S)- and (S)-sulpiride in seven pure solvents and four mixtures (chiral ionic liquids + methanol) from 283.15 K to 323.15 K, J. Mol. Liq. (2024) https://www.sciencedirect.com/science/article/pii/S0167732224019871
- Zihao Fu, Song Guo, Hong-Bin Xie, et al., A Near-Explicit Reaction Mechanism of Chlorine-Initiated Limonene: Implications for Health Risks Associated with the Concurrent Use of Cleaning Agents and Disinfectants, Environ. Sci. Technol. (2024) https://pubs.acs.org/doi/abs/10.1021/acs.est.4c04388
- Zhen Zhang, Xiaoguang Zheng, Jiayi Li, et al., Mechanism of reinforced interfacial adhesion between steel slag and highly devulcanized waste rubber modified asphalt and its influence on the volume stability in steel slag asphalt mixture, Const. Build. Mater., 447, 138129 (2024) https://www.sciencedirect.com/science/article/pii/S0950061824032719
- Ende Huang, Siyue Liu, Xiaohu Zhou, et al., Vibrational Spectroscopy and Structural Characterization of Neutral and Cationic Hydroxyacetone, J. Mol. Struct. (2024) https://www.sciencedirect.com/science/article/pii/S0022286024023809
- Hao Dong, Bo Wang, Xiandong Hao, et al., Kinetic and Thermodynamic Analysis of H2S Absorption by a Non-aqueous Absorbent of Monoethanolamine /Ethylene Glycol, J. Environ. Chem. Eng. (2024) https://www.sciencedirect.com/science/article/pii/S2213343724021523
- Wanxiang Zhang, Pan Xu, Zhengrun Chen, et al., Carboxylic acid-based deep eutectic solvent for efficient desulfurization: Experimental and computational thermodynamics, Fuel, 378, 132975 (2024) https://www.sciencedirect.com/science/article/pii/S0016236124021240
- Yuhang He, Hua Ke, Yangcheng Lu, Solvent Effect in the Copolymerization of Ethylene and Vinyl Acetate, Ind. Eng. Chem. Res. (2024) https://pubs.acs.org/doi/abs/10.1021/acs.iecr.4c01465 (此文没有按要求引用Molclus!)
- Wanxiang Zhang, Yasen Dai, Jianjun Sun, et al., Efficient removal and molecular insight of neutral oil in the separation of phenolic compounds by deep eutectic solvents, Sep. Purif. Technol. (2024) https://www.sciencedirect.com/science/article/pii/S1383586624030995
- Yihan Li, Lin Zhao, Daying Chen, et al., Specific adsorption of antiviral drugs on ionic liquid-modified metal–organic framework: Adsorption behavior, mechanism, and DFT analysis, Chem. Eng. J. (2024) https://www.sciencedirect.com/science/article/pii/S1385894724065616
- Yan Zhang, Yafei Luo, Changwei Hu, et al., Redox-neutral Depolymerization of Lignin-Derived Aryl Ethers Catalyzed by Rh(III)-complexes: A Mechanistic Insight, Phys. Chem. Chem. Phys. (2024) https://pubs.rsc.org/en/content/articlehtml/2024/cp/d4cp02660h
- Pengcheng Liu, Jia Han, Haili Yu, et al., Structural Study of [Sc3O4(CO2)n]+ (n = 2, 3) Complexes by Infrared Photodissociation Spectroscopy and Density Functional Calculations, J. Phys. Chem. A (2024) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.4c04163
- Shan Li, Yuanyuan Xie, Baoli Zhang, et al., A Host–Guest Approach to Engineering Oxidase-Mimetic Assembly with Substrate Selectivity and Dynamic Catalysis, ACS Appl. Mater. Interfaces (2024) https://pubs.acs.org/doi/abs/10.1021/acsami.4c08030 (此文没有按要求引用Molclus!)
- Yuhui Ci, Dong Lv, Xiangjian Yang, et al., High-performance cellulose/thermoplastic polyurethane composites enabled by interaction-modulated cellulose regeneration, Carbohyd. Polym. (2024) https://www.sciencedirect.com/science/article/pii/S0144861724008373
- Yu-Cheng Zheng, Wen-Jian Gu, Ren-Geng Shu, Pu-Zhao Zhang, Four new homoisoflavonoids from Caesalpinia pulcherrima, J. Asian Nat. Prod. Res. (2024) https://www.tandfonline.com/doi/abs/10.1080/10286020.2024.2387307
- Jing Wang, Lei Zhuang, Enze Gao, et al., Dissociation of HBr in Water Clusters Based on a Hybrid Density Functional Approach, J. Phys. Chem. A (2024) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.4c02966
- Wenjin Cao, Zhubin Hu, Haitao Sun, Xue-Bin Wang, Photoelectron Spectroscopy and Computational Study on Microsolvated [B10H10]2– Clusters and Comparisons to Their [B12H12]2– Analogues, J. Phys. Chem. A (2024) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.4c04772
- Yanrong Jiang, Wenjin Cao, Zhubin Hu, et al., A comprehensive study on three typical photoacid generators using photoelectron spectroscopy and ab initio calculations, J. Chem. Phys. 161, 054311 (2024) https://pubs.aip.org/aip/jcp/article-abstract/161/5/054311/3306673/A-comprehensive-study-on-three-typical-photoacid
- Xin Jiang, Zhaopeng Hu, Shichao Xu, et al., PEDOT:PSS/XNBR film for low-temperature stretchable electromagnetic interference shielding, Compos. Commun. (2024) https://www.sciencedirect.com/science/article/pii/S2452213924002201
- Yuehua Deng, Ge Huang, Shiyuan Liu, et al., Loading co-amorphous on Metal-Organic Frameworks for gelation elimination and anti-cancer drug delivery enhancement, Chem. Eng. J., 497, 154452 (2024) https://www.sciencedirect.com/science/article/pii/S1385894724059412
- Ling-Hao Zhao, Xin-Yi Guo, Hai-Wei Yan, et al., A class of geranylquinol-derived polycyclic meroterpenoids from Arnebia euchroma against heart failure by reducing excessive autophagy and apoptosis in cardiomyocytes, Bioorg. Chem., 151, 107691 (2024) https://www.sciencedirect.com/science/article/pii/S0045206824005960
- Pengfei Xiao, Siyue Liu, Xiaohu Zhou, et al., Structure and vibrational spectroscopy of 2-methylallyl alcohol, Chin. J. Chem. Phys., 37, 481 (2024) https://doi.org/10.1063/1674-0068/cjcp2309078
- Quanjie Zhong, Simple and effective descriptor for measuring the nonlinear optical responses of atomic clusters, ChemRxiv (2024) https://chemrxiv.org/engage/chemrxiv/article-details/66a22ec501103d79c5a360dd
- Bo Yang, Yifan Zhang, Lijuan Wang, et al., Selective extraction performance and mechanism of lithium using Lewis base ligands containing phosphoryl/carbonyl groups, Sep. Purif. Technol. (2024) https://www.sciencedirect.com/science/article/pii/S1383586624026868
- Dongbo Wang, Jingting Sang, Ying Wang, et al., Crystallization resolution of racemic compounds via chiral surfactant, Chem. Eng. Sci., 299, 120540 (2024) https://www.sciencedirect.com/science/article/pii/S0009250924008406
- Wei Wang, Shiwei Deng, Jinzhong Lin, Defang Ouyang, Modeling on in vivo disposition and cellular transportation of RNA lipid nanoparticles via quantum mechanics/physiologically-based pharmacokinetic approaches, Acta Parm. Sin. B (2024) https://www.sciencedirect.com/science/article/pii/S2211383524002429 (此文没有按要求引用Molclus!)
- Di Wu, Shuyu Li, Yaoguang Feng, et al., Unveiling the conformational landscape of tizanidine and its effects on selective nucleation: Enabling controlled manipulation of polymorphs and solvates, Sep. Purif. Technol., 354, 128827 (2025) https://www.sciencedirect.com/science/article/pii/S1383586624025668
- Baochen Ma, Haikuo Zhang, Ruhong Li, et al., Molecular-docking electrolytes enable high-voltage lithium battery chemistries, Nat. Chem. (2024) https://www.nature.com/articles/s41557-024-01585-y
- Wanfang Feng, Yuyan Hu, Maoqing Wang, Li-Yan Liu, Highly Defective Zirconium-Based Metal–Organic Frameworks for the Efficient Adsorption and Detection of Sugar Phosphates in the Biological Sample, ACS Appl. Mater. Interfaces, 16, 37641 (2024) https://pubs.acs.org/doi/abs/10.1021/acsami.4c06870
- Ya Lu, Xueyan Huang, Donovon A. Adpressa, et al., Vitifolignans, 3,4-dibenzyltetrahydrofuran lignans from Anemone vitifolia Buch.-Ham., Phytochem. (2024) https://www.sciencedirect.com/science/article/pii/S0031942224002541 (此文没有按要求引用Molclus!)
- Weiyi Xu, Jipeng Xu, Zhiyuan Yi, et al., Zwitterionic channels within covalent organic frameworks facilitate proton-selective transport for flow battery membrane, Chem. Eng. Sci. (2024) https://www.sciencedirect.com/science/article/pii/S0009250924007681
- Chunyu Liu, Wanxiang Zhang, Wuji Zhao, et al., Simultaneous absorption of CO2 and H2S by [Emim][Ac]: The competition mechanism, Sep. Purif. Technol., 354, 128643 (2025) https://www.sciencedirect.com/science/article/pii/S1383586624023827
- Xukang Jin, Qi Duan, Xiuting Li, et al., Dissolution of Curcumin by Deep Eutectic Solvent of Fatty Acids, Journal of East China University of Science and Technology, 50, 214 (2024) https://journal.ecust.edu.cn/article/doi/10.14135/j.cnki.1006-3080.20230228001
- Zhengrun Chen, Hui Xu, Xiaoteng Zang, et al., Experimental and molecular insights on the regulatory effects of solvent on CaC2 reaction activity, AIChE J. (2024) https://aiche.onlinelibrary.wiley.com/doi/abs/10.1002/aic.18511
- Qijiang Shu, Jun Bi, Jingjing Lu, Molecular dynamics study on self-assembled dodecylphosphocholine nanomicelles used as drug delivery system for carrying curcumin, J. Phys.: Conf., 2783, 012064 (2024) https://doi.org/10.1088/1742-6596/2783/1/012064
- Yongji Guan, Chuyu Li, Xin Han, et al., Effect of the external electric fields on the polarization and vibrational spectrum of 1-ethyl-3-methylimidazolium bis(trifluormethylsulfonyl)imide on graphene surface, J. Mol. Liq., 408, 125313 (2024) https://www.sciencedirect.com/science/article/pii/S0167732224013709
- Nan Li, Shideng Yuan, Ruidian Su, et al., Selective and antibacterial zinc phosphonate framework system for superior marine uranium harvesting, Desalination, 586, 117860 (2024) https://www.sciencedirect.com/science/article/pii/S001191642400571X (此文没有按要求引用Molclus!)
- Jiahui Wei, Hanwen Ge, Baoping Zhu, et al., Probing the interaction mechanism of choline chloride-based deep eutectic solvents with flavonoid and polyphenol systems, J. Mol. Liq., 408, 125310 (2024) https://www.sciencedirect.com/science/article/pii/S0167732224013679
- Nnamdi M. Ofodum, Qingkai Qi, Richard Chandradat, et al., Advancing Dynamic Polymer Mechanochemistry through Synergetic Conformational Gearing, J. Am. Chem. Soc. (2024) https://pubs.acs.org/doi/abs/10.1021/jacs.4c02066
- Yu Zhao, Menghuan Jiang, Xiaoming Xiu, et al., ESIPT-associated fluorescent property and AIE characteristic of multifunctional nanoprobe DPNAP, J. Mol. Liq., 407, 125291 (2024) https://www.sciencedirect.com/science/article/pii/S0167732224013485
- Jun Ma, Zhi Shen, Guoxuan Li, et al., Amide solvents for extraction and separation of 1-hexene from n-heptane: Effect of the amount of methyl groups, Fuel, 372, 132134 (2024) https://www.sciencedirect.com/science/article/pii/S0016236124012821
- Zuping Xiong, Jianyu Zhang, Lei Wang, et al., Virtual screening for ultra-small NIR emitter with only two isolated hexatomic rings, ChemRxiv (2024) https://chemrxiv.org/engage/chemrxiv/article-details/66697cbf12188379d8ebcf20
- Weidong Cao, Xingwen Li, Chaojie Luo, et al., A theoretical model considering the photochemical and photothermal behavior of arc radiation-induced gassing materials ablation, J. Phys. D (2024) https://iopscience.iop.org/article/10.1088/1361-6463/ad5601/meta
- Surajit Metya, Supravat Roy, Sourav Mandal, et al., Modulation of the strength of weak S–H⋯O hydrogen-bond: Spectroscopic study of the complexes of 2-flurothiophenol with methanol and ethanol, J. Chem. Phys., 160, 224302 (2024) https://pubs.aip.org/aip/jcp/article-abstract/160/22/224302/3297305/Modulation-of-the-strength-of-weak-S-H-O-hydrogen
- Binyi Qin, Jie Qiu, Ruizhao Yang, et al., Structural Investigation of Co-crystal Formed Between Theophylline And Nicotinamide Based On Terahertz and Raman Spectroscopy, IEEE Transactions on Terahertz Science and Technology (2024) https://ieeexplore.ieee.org/abstract/document/10551484
- Bumaliya Abulimiti, Jiajun Ma, Jie Wei, et al., Ultrafast conformation-dependent charge transfer in N, N, N′, N′-tetramethyl-1,3-propanediamine: Effect of flexible carbon skeleton on electron lone pair interactions, Spectrochim. Acta A (2024) https://www.sciencedirect.com/science/article/pii/S1386142524007947
- Jurong Lv, Xiaoqing Du, Yajing Hua, et al., Controllable product selectivity and reaction paths in CO2 electroreduction: A study of density functional theories of small CuxM clusters, Comput. Mater. Sci., 244, 113148 (2024) https://www.sciencedirect.com/science/article/pii/S0927025624003690 (此文没有按要求引用Molclus!)
- Guoqing Cheng, Naoki Komatsu, Diameter-selective extraction of single-walled carbon nanotubes by interlocking with Cu-tethered square nanobrackets, Beilstein J. Org. Chem. (2024) https://www.beilstein-journals.org/bjoc/articles/20/113
- Aamir Hassan Shah, Molecular-level Understanding of Electrode-Electrolyte Interface in Hydrogen Evolution Reaction, PhD thesis of UNIVERSITY OF CALIFORNIA (2024) https://escholarship.org/content/qt7863931b/qt7863931b.pdf
- Lei Zhuang, Jing Wang, Jianguo Wan, Chuanfu Huang, Why Dipole Moments of HCl-Water Cluster Fail to Determine Acid Dissociation? Phys. Chem. Chem. Phys. (2024) https://pubs.rsc.org/en/content/articlelanding/2024/cp/d4cp01316f/
- Shuai-Ting Yan, Xi-Ling Xu, Hong-Guang Xu, Wei-Jun Zheng, Anion photoelectron spectroscopy and quantum chemical calculations of LaCu2On-/0 (n=4,5): Interaction between oxygen unit and LaCu2O3 six-membered ring, Chin. J. Chem. Phys. (2024) https://pubs.aip.org/cps/cjcp/article/37/3/313/3295573
- Piao Xu, Jianduo Lu, Dongdong Wang, et al., Ultraviolet photoemission spectra of aqueous thymidine by liquid-microjet photoelectron spectroscopy: A combined experimental error assessment, Chin. J. Chem. Phys. (2024) https://pubs.aip.org/cps/cjcp/article/37/3/439/3295587
- Huacan Wu, Shiya Zheng, Xiaolan Yu, et al., Tuning the emission of anti-rigidochromic fluorophore by alternating the polymer matrices for advanced data encryption, J. Lumin., 273, 120706 (2024) https://www.sciencedirect.com/science/article/pii/S0022231324002709
- Benni Du, Weichao Zhang, Hydrolysis of acrolein promoted by water, ammonia or formic acid in the atmosphere: A theoretical investigation, Comput. Theor. Chem. (2024) https://www.sciencedirect.com/science/article/pii/S2210271X24002068
- Mengting Yu, Jia Liu, Xiaoyong Cao, et al., Structures and hydrogen bonds of -SO3H functionalized acid ionic liquids, J. Mol. Liq., 406, 125129 (2024) https://www.sciencedirect.com/science/article/pii/S0167732224011851
- Lie Liu, Yuqi Cui, Qingwen Tang, et al., A Janus membrane with electro-induced multi-affinity interfaces for high-efficiency water purification, Sci. Adv. (2024) https://www.science.org/doi/full/10.1126/sciadv.adn8696
- Dingkai Hu, Qiang Wang, Jing Li, et al., Thermodynamic properties of TPAC-EG deep eutectic solvents: A study based on the IGC method with molecular simulations, J. Mol. Liq. (2024) https://www.sciencedirect.com/science/article/pii/S0167732224011760
- Yue Yang Yang, Jialing Yu, Xiankai Jiang, et al., Insight into the interaction between amino acids and SO2: Detailed bonding modes, ResearchSquare (2024) https://www.researchsquare.com/article/rs-4383234/v1
- Wen Liu, Lulu Huang, Jin Hu, Xiaopeng Xing, Adsorption of Multiple NO Molecules on Anionic Gold Clusters: Electron Transfer and Disproportionation Enhanced by Molecular Aggregation, J. Phys. Chem. A (2024) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.4c02107
- Wenxin Tian, Huimin Zhou, Yetong Liu, et al., A robust polypyrrole nanofibrous membrane for versatile applications: oil-water separation and dye removal, J. Environ. Chem. Eng. (2024) https://www.sciencedirect.com/science/article/pii/S2213343724012399
- Yi-fan Zhang, Xian-wei Hui, Jiang-yu Yu, Zhao-wen Wang, Raman Spectroscopy and Theoretical Calculation on Structure of Na2CO3-K2CO3-NaVO3 Molten Salts, J. Northeastern Univ.(Natural Science), 45, 331 (2024) https://xuebao.neu.edu.cn/natural/EN/10.12068/j.issn.1005-3026.2024.03.004
- Wen-Chao Tu, Yong-Xiang Huang, Yuan-Lin Kong, et al., Biswulfenioidins A–E, dioxygen-bridged abietane-type diterpenoid dimers with anti-Zika virus potential from Orthosiphon wulfenioides, Org. Chem. Front. (2024) https://pubs.rsc.org/en/content/articlelanding/2024/qo/d4qo00619d/
- Chengbin Fu, Kun Du, Jie Xue, et al., Mechanisms of acid generation from ionic photoacid generators for extreme ultraviolet and electron beam lithography,
Phys. Chem. Chem. Phys. (2024) https://pubs.rsc.org/en/content/articlelanding/2024/cp/d4cp01814a/ - Zhonghao Li, Zhou Feng, Hongwei Jin, et al., Absorption characteristics and molecular mechanism of a low viscosity iron-based ionic liquid for removing H2S, J. Environ. Chem. Eng., 12, 113030 (2024) https://www.sciencedirect.com/science/article/pii/S2213343724011606
- Tian Lu, Theoretical Prediction and Comprehensive Characterization of an all-Nitrogenatomic Ring, Cyclo[18]Nitrogen (N18), ChemPhysChem (2024) https://chemistry-europe.onlinelibrary.wiley.com/doi/abs/10.1002/cphc.202400377
- Yajie Zhang, Feng Han, Lei Wang, et al., Influence of the adsorption effect of graphene quantum dot on the photophysical properties and multiple antioxidant processes of gallic acid, Chem. Phys. Lett. (2024) https://www.sciencedirect.com/science/article/pii/S0009261424002628
- Yi Zheng, Yunuo Dai, Yawen Hong, et al., Alkyl chain length affecting the structure and interactions of choline-based amino acid ionic liquids: Insights from a computational study, J. Mol. Liq., 404, 124972 (2024) https://www.sciencedirect.com/science/article/pii/S0167732224010286
- Yajie Zhang, Feng Han, Lei Wang, et al., Influence of the adsorption effect of graphene quantum dot on the photophysical properties and multiple antioxidant processes of gallic acid, Chem. Phys. Lett. (2024) https://www.sciencedirect.com/science/article/pii/S0009261424002628
- Jia-Min Xu, Yi-Cheng Xu, Chen-Xi Cheng, et al., SfuPKS2, a novel type III polyketide synthase from Sargassum fusiforme, shows high substrate specificity, J. Appl. Phycol. (2024) https://link.springer.com/article/10.1007/s10811-024-03262-6
- Hongwei Jin, Xiaohan Li, Kangkang Li, et al., CO2 absorption using two morpholine protic ionic liquids: measurement, model, and quantum chemical calculation, J. Chem. Thermodyn., 196, 107322 (2024) https://www.sciencedirect.com/science/article/pii/S0021961424000752
- Jiaming Mao, Yuhui Ci, Jia Liu, et al., Experimental and theoretical investigation of an ionic liquid-based biphasic solvent for post-combustion CO2 Capture: Breaking through the “Trade-off” effect of viscosity and loading, Chem. Eng. J., 491, 151991 (2024) https://www.sciencedirect.com/science/article/pii/S1385894724034788
- Zhipeng Qiu, Rui Li, Jin Chen, et al., Favored CH-π interaction between enzymatically modified high amylose starch and resveratrol improves digestion resistance, Food Hydrocolloids, 154, 110137 (2024) https://www.sciencedirect.com/science/article/pii/S0268005X24004119 (此文没有按要求引用Molclus!)
- Rongtai Wan, Jiawen Yu, Ziyi Quan, et al., A reusable, healable, and biocompatible PEDOT:PSS hydrogel-based electrical bioadhesive interface for high-resolution electromyography monitoring and time–frequency analysis, Chem. Eng. J., 490, 151454 (2024) https://www.sciencedirect.com/science/article/pii/S1385894724029413 (此文没有按要求引用Molclus!)
- Tianyang Hou, Xiaofeng Yuan, Shuaijie Jiang, et al., Experimental detection of the diamino-pentazolium cation and theoretical exploration of derived high energy materials, Sci. Rep., 14, 10120 (2024) https://www.nature.com/articles/s41598-024-60741-z
- Hua Yang, Jiao Wang, Qiuru Tan, et al., Supramolecular Interaction between Berberine Hydrochloride and Baicalin in Aqueous Solution: Reaction Kinetics, Spectral Analysis and Dynamic Simulation, Heliyon (2024) https://www.cell.com/heliyon/fulltext/S2405-8440(24)06023-7
- Yu Hong, Yuqian Jiang, Chenhuan Yuan, et al., Unraveling temperature-dependent supramolecular polymorphism of naphthalimide-substituted benzene-1,3,5-tricarboxamide derivatives, Chin. Chem. Lett. (2024) https://www.sciencedirect.com/science/article/pii/S1001841724004285
- Anni Sun, Fuchun Nan, Qilin Wei, et al., Color-tunable, multifunctional photochromic composites for wearable UV monitoring and biomechanical energy harvesting, Nano Energy (2024) https://www.sciencedirect.com/science/article/pii/S2211285524004270
- Hanwen Ge, Yuekun Bai, Rui Zhou, et al., Explicable Machine Learning for Predicting High-Efficiency Lignocellulose Pretreatment Solvents Based on Kamlet–Taft and Polarity Parameters, ACS Sustainable Chem. Eng. (2024) https://pubs.acs.org/doi/abs/10.1021/acssuschemeng.4c01563
- Jing Cheng, Rixuan Wang, Yanyu Hu, et al., Fermentation-inspired macroporous and tough gelatin/sodium alginate hydrogel for accelerated infected wound healing, Int. J. Biol. Macromol., 268, 131905 (2024) https://www.sciencedirect.com/science/article/pii/S0141813024027107 (此文没有按要求引用Molclus!)
- Xuyao Qi, Tao Wang, Lanjun Zhang, et al., Mechanism of deep eutectic solvent on coal spontaneous combustion, J. China Coal Soc., 49, 1 (2024) https://doi.org/10.13225/j.cnki.jccs.XR23.1551
- Yanbo Liu, Shuo Wang, Maolin Li, et al., Design and preparation of spherical particles in water via thermal-induced liquid-liquid phase separation and a molecular mechanism study, Chem. Eng. Sci. (2024) https://www.sciencedirect.com/science/article/pii/S0009250924004706
- Qingwen Huang, Xiuping Lou, Dongxia Nie, et al., Accelerated photodegradation of T-2 toxin over magnetic recyclable ZnO/CaFe2O4 nanocomposite with a p-n based Z-scheme heterojunction architecture, Sep. Purif. Technol. (2024) https://www.sciencedirect.com/science/article/pii/S1383586624012577
- Fei Zhao, Qinghua Liu, Minghao Song, et al., Ionic liquid for simultaneous desulfurization and dehydration of flue gas: Data-driven thermodynamics and mechanisms, AIChE J. (2024) https://aiche.onlinelibrary.wiley.com/doi/abs/10.1002/aic.18456
- Chenyang Zhao, Yuan Wang, Yuqian Jiang, et al., Handedness-Inverted and Stimuli-Responsive Circularly Polarized Luminescent Nano/Micro-Materials through Pathway-Dependent Chiral Supramolecular Polymorphism, Adv. Mater. (2024) https://onlinelibrary.wiley.com/doi/abs/10.1002/adma.202403329
- Hongwei Kang, Guoxuan Li, Zhigang Lei, Phase behavior of sodium and potassium salts in their aqueous solutions: Experiment and computational thermodynamics, AIChE J. (2024) https://aiche.onlinelibrary.wiley.com/doi/abs/10.1002/aic.18447
- Xin Jiang, Zhaopeng Hu, Shichao Xu, et al., A low-temperature stretchable XNBR film containing Li-TFSI doped PEDOT:PSS for electromagnetic interference shielding, Chem. Eng. J. (2024) https://www.sciencedirect.com/science/article/pii/S1385894724027396
- Meiyu Zhang, Kang Kang, Lei Li, Chengliang Xiao, Reticular access to triptycene-based cationic metal–organic frameworks for protein-bound uremic toxins sorption, Sep. Purif. Technol. (2024) https://www.sciencedirect.com/science/article/pii/S1383586624012267
- Yiming Zhao, Yongchun Zhang, Qi Liu, et al., The co-promoting mechanism of EMEA/DEEA/PZ ternary water-lean solvent on CO2 capture- a study based on 13C-qNMR and DFT calculations, Sep. Purif. Technol. (2024) https://www.sciencedirect.com/science/article/pii/S1383586624012073
- Ge Zhu, Jinglei Xing, Nafeesa Mushtaq, et al., Synthesis and characterization of high Tg and colorless polyamide-imide films derived from amide-containing diamines with different trifluoromethyl substitution, Polymer (2024) https://www.sciencedirect.com/science/article/pii/S0032386124003689
- Naitian Zhang, Yuehui Li, Wenzhe Shang, et al., Unveiling the key role of excited-state hydrogen bonding in homogeneous photocatalytic CO2 reduction, New J. Chem. (2024) https://pubs.rsc.org/en/content/articlelanding/2024/nj/d3nj05851d/
- Pengcheng Liu, Jia Han, Yan Chen, et al., Binding Strengths and Orientations in CO2 Adsorption on Cationic Scandium Oxides: Governing Factor Revealed by a Combined Infrared Spectroscopy and Theoretical Study, J. Phys. Chem. A (2024) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.4c01562
- Ruiqi Lu, Putian Zhou, Fangfang Ma, et al., Multi-generational oxidation mechanism of M-xylene: Unexpected implications for secondary organic aerosol formation, Atmosph. Environ. (2024) https://www.sciencedirect.com/science/article/pii/S1352231024001869
- Weikang Gao, Chuanlei Liu, Yuxiang Chen, et al., Exploring ionic liquids for the extraction separation of aromatic hydrocarbons from diesel via multiple interaction analyses coupled with experimental evaluation, Fuel, 368, 131634 (2024) https://www.sciencedirect.com/science/article/pii/S0016236124007828
- Heyao Yuan, Xiaolong Li, Jin Li, et al., Detection mechanism of glutathione by off–on fluorescent probe based on excited state intramolecular proton transfer, Chem. Phys. Lett. (2024) https://www.sciencedirect.com/science/article/pii/S0009261424001829
- Zihao Fu, Song Guo, Ying Yu, et al., Oxidation Mechanism and Toxicity Evolution of Linalool, a Typical Indoor Volatile Chemical Product, Environ. Health (2024) https://pubs.acs.org/doi/full/10.1021/envhealth.4c00033
- Yu Cao, Ziyue Zhou, Dongshun Deng, Investigation on mechanisms of ammonia absorption in [Im][SCN]-based eutectic solvents using computational methods, J. Mol. Liq. (2024) https://www.sciencedirect.com/science/article/pii/S0167732224006949
- Xiaoyang Liu, Shivani Kozarekar, Alexander Shaw, et al., Understanding ceiling temperature as a predictive design parameter for circular polymers, Cell Rep. Phys. Sci. (2024) https://www.cell.com/cell-reports-physical-science/fulltext/S2666-3864(24)00146-2
- Jinxin Lin, Liheng Chen, Yanlin Qin, Xueqing Qiu, Understanding Lignin Dissolution with Urea and the Formation of a Lignin Nano-Aggregate: A Multiscale Approach, Nanomaterials, 14, 593 (2024) https://doi.org/10.3390/nano14070593
- Zhijie Shang, Pan Xu, Guoxuan Li, et al., Ionic-Liquid-Assisted Capture of Volatile Low-Carbon Alcohols in Printing Plant Exhaust Gas, ACS Sustainable Chem. Eng. (2024) https://pubs.acs.org/doi/abs/10.1021/acssuschemeng.4c00552 (此文没有按要求引用Molclus!)
- Ming Lin, Jinzhao Guan, Mouhamadou Aziz Diop, et al., Unveiling ionic structure in the LiF-NdF3 molten salt system as determined by Raman spectroscopy and quantum chemical calculations, J. Mol. Liq. (2024) https://www.sciencedirect.com/science/article/pii/S0167732224006585
- Fan Li, Taotao Zhe, Kaixuan Ma, et al., Sustainable Supramolecular Plastics Usable Under High Humidity: Cross-Linking Strategy Enabled by Hydrogen Bonding and Coordination Interactions, ACS Sustainable Chem. Eng. (2024) https://pubs.acs.org/doi/abs/10.1021/acssuschemeng.3c08571
- Jianghe Liu, Sanlue Hu, Hexin Guo, et al., Regulating the inner Helmholtz plane structure at the electrolyte-electrode interface for highly reversible aqueous Zn batteries, J. Energy Chem. (2024) https://www.sciencedirect.com/science/article/pii/S2095495624002365
- Shaopeng Zhang, Jie Ding, Dayong Tian, et al., Synthesis of N,S-containing hyper-cross-linked polymers and their efficient tetracycline and diclofenac sodium adsorption in water, Mater. Sci. Eng. B, 303, 117335 (2024) https://www.sciencedirect.com/science/article/pii/S0921510724001648
- Chao Chen, Siyan Deng, Shuzhou Li, Using small database and energy descriptors to predict molecular thermodynamic energies through mediated learning models, Chem. Eng. J. (2024) https://www.sciencedirect.com/science/article/pii/S1385894724020941
- Honghui Zhao, Hongyuan Shen, Ao You, Yuanqin Yu, Effect of Substitution Group on Intramolecular Hydrogen Bond of Amino Alcohols from Raman spectroscopy, Arxiv (2024) https://arxiv.org/ftp/arxiv/papers/2403/2403.14129.pdf (此文没有按要求引用Molclus!)
- Hangchao Wang, Yali Yang, Chuan Gao, et al., An entanglement association polymer electrolyte for Li-metal batteries, Nat. Commun., 15, 2500 (2024) https://www.nature.com/articles/s41467-024-46883-8
- Ningning Zhu, Jiajun Xu, Qi Su, et al., Site-specific albumin tagging with NIR-II fluorogenic dye for high-performance and super-stable bioimaging, Theranostics, 14, 1860 (2024) https://www.thno.org/v14p1860.htm
- Zhixin Zheng, Xin Huang, Na Wang, et al., Mechanistic Study on the Effect of Solvents on Polymorphic Transformation of Aripiprazole by Solvent Property Parameters and Molecular Dynamics Simulation, Cryst. Growth Des. (2024) https://pubs.acs.org/doi/abs/10.1021/acs.cgd.3c01529
- Xiaoqian Shao, Shanjun Chen, Yan Chen, et al., Probing the geometric and electronic structures of the transition metal oxides RhOn–1/0 (n = 1–4) clusters, Spectro. Chim. Acta, 313, 124146 (2024) https://www.sciencedirect.com/science/article/pii/S1386142524003123
- Haisong Wu, Xiaoyu Li, Qiuyu Chen, et al., Highly efficient separation of phenol with tetraethylammonium chloride-based deep eutectic solvents: Experiments and theoretical calculations, J. Mol. Liq., 399, 124470 (2024) https://www.sciencedirect.com/science/article/pii/S0167732224005269
- Jiaqiang Wang, Da Wu, Han Zhao, et al., Facile synthesis of DES-based functionalized Polyaniline@Fe3O4 magnetic microspheres for continuous and efficient dye removal: Properties and mechanism, Sep. Purif. Technol. (2024) https://www.sciencedirect.com/science/article/pii/S1383586624007238
- Dingkai Hu, Qiang Wang, Xinyue Wang, et al., Experimental and Mechanistic Investigation of Amide-Based Deep Eutectic Solvents for Quinoline Extraction and Separation from Wash Oil, Ind. Eng. Chem. Res. (2024) https://pubs.acs.org/doi/abs/10.1021/acs.iecr.3c04327
- Sheng XIANG, Qilei LIU, Lei ZHANG, Jian DU, Computer-aided design of organic amine solvents for carbon capture based on reaction kinetics, Tsinghua University(Science and Technology), 64, 520 (2024) http://jst.tsinghuajournals.com/CN/abstract/abstract154528.shtml
- Ding Wei, Jialuo Xiang, Qinlan Luo, et al., Kinetic study on CO2 absorption by aqueous secondary amine + tertiary amine blends: Kinetic model and effect of the chain length of tertiary amine, Chem. Eng. Sci. (2024) https://www.sciencedirect.com/science/article/pii/S0009250924002963
- Jing-Wen Wang, Yi-Yi Liu, Guang-Ying Chen, et al., Alkaloid-metabolites from the mangrove-derived fungus Penicillium robsamsonii HNNU0006, Fitoterapia, 175, 105906 (2024) https://www.sciencedirect.com/science/article/pii/S0367326X24000893
- Zhen-Qing Yu, Hao Ren, Xiao-Wei Guo, et al., Targeted isolation, identification, and antioxidant evaluation of aromatic polyketides from a plant-derived fungus Ophiobolus cirsii LZU-1509, Fitoterapia (2024) https://www.sciencedirect.com/science/article/pii/S0367326X24000674
- Ana Aline C. Alcanfor, Leonardo P. da Silva, Raíssa C. de Oliveira, et al., On the role of water in antimony electrodeposition from choline chloride/ethylene glycol/water mixture, J. Mol. Liq. (2024) https://www.sciencedirect.com/science/article/pii/S0167732224004720
- Wenyuan Han, Hao Zhang, Degang Li, et al., Surface engineered carbon quantum dots for efficient photocatalytic hydrogen peroxide production, Appl. Catal. B, 350, 123918 (2024) https://www.sciencedirect.com/science/article/pii/S0926337324002327 (此文没有按要求引用Molclus!)
- Yi-Hao Che, Wen-Ping Ding, Zhi-Hui Xiao, et al., New Phenol Derivatives from the Haima Cold Seep-Derived Fungus Aspergillus subversicolor CYH-17, Mar. Drugs, 22, 117 (2024) https://doi.org/10.3390/md22030117
- Camila Botin Francisco, Cleverton de Souza Fernandes, Fernanda Franco Dourado, et al., Conformational Landscape of α-Halopropiophenones Determined by nJC–H NMR Reveals Unexpected Patterns and Geometric Constraints, J. Phys. Chem. A (2024) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.3c06934
- Yongpan Tian, Zhilou Liu, Zhongshuai Jia, et al., Mechanism for lithium extraction with ether and ester oxygens in armed crown ethers, J. Mol. Liq. (2024) https://www.sciencedirect.com/science/article/pii/S0167732224004069
- Renting Li, Suying Chu, Chuanxin Cui, et al., Study on separation of methanol and dimethyl carbonate azeotropic system with deep eutectic solvents, J. Mol. Liq. (2024) https://www.sciencedirect.com/science/article/pii/S016773222400357X
- Xiaoli Gong, Liyao Zhu, Renyi Zhang, Technical note: A theoretical study on the mechanism of citric aciddriven multi-component nucleation of sulfuric acid-base-water clusters, EGUsphere preprint (2024) https://doi.org/10.5194/egusphere-2023-3113
- Shiyi Zuo, Tian Liu, Lingxiao Li, et al., Tetrasulfide bond boosts the anti-tumor efficacy of dimeric prodrug nanoassemblies, Cell Reports Medicine (2024) https://doi.org/10.1016/j.xcrm.2024.101432
- Yu Wang, Jiuxu Ruan, Ke Xue, et al., Phase Equilibrium of p-Xylene and Isobutanol in Ionic Liquid Separations: Thermodynamic and Mechanistic Analysis, ACS Sustainable Chem. Eng. (2024) https://pubs.acs.org/doi/abs/10.1021/acssuschemeng.3c08507
- Nnamdi M. Ofodum, Qingkai Qi, Richard Chandradat, et al., Advancing Dynamic Polymer Mechanochemistry through Molecular Gears, ChemRxiv (2024) https://chemrxiv.org/engage/chemrxiv/article-details/65c5675e66c13817293ea9e9
- Bo Wang, Jing Chen, Jinyu Wang, et al., Molecular insights into the absorption of SO2 with amine alkyl organosilicon and its optimal absorption temperature, Sep. Purif. Technol. (2024) https://www.sciencedirect.com/science/article/pii/S138358662400399X (此文没有按要求引用Molclus!)
- Qiyang Lin, Jia Yang, Shuai Wang, et al., A Novel Bipolar Surfactant, BETX, for Selective Flotation of Chalcopyrite: Folding-Synergistic Mechanism Based on Intramolecular Weak Interactions, Ind. Eng. Chem. Res. (2024) https://pubs.acs.org/doi/abs/10.1021/acs.iecr.3c04216
- Xiaogai Peng, Wenjin Cao, Zhubin Hu, et al., Observation of a super-tetrahedral cluster of acetonitrile-solvated dodecaborate dianion via dihydrogen bonding, J. Chem. Phys. 160, 054308 (2024) https://pubs.aip.org/aip/jcp/article/160/5/054308/3262360
- Zhubin Hu, Qiaoqiao Shao, Zhipeng Li, et al., Deprotonated sulfamic acid and its homodimers: Does sulfamic acid adopt zwitterion during cluster growth? J. Chem. Phys. 160, 054303 (2024) https://pubs.aip.org/aip/jcp/article/160/5/054303/3261735
- Duomei Xue, Jiaqian Yang, Zeren Chen, et al., Dual-Valence Characteristics of Be11: Tin/Lead-like Superatom, Inorg. Chem. (2024) https://pubs.acs.org/doi/abs/10.1021/acs.inorgchem.3c04200
- Jiangtao Li, Hongye Cheng, Zhiwen Qi, Enhancement of separation performance for ofloxacin enantiomers by a biphasic recognition chiral extraction system, AIChE J. (2024) https://aiche.onlinelibrary.wiley.com/doi/abs/10.1002/aic.18367
- Dong Liu, Yuan Shui, Tao Yang, et al., Geometries, Electronic Structures, Bonding Properties, and Stability Strategy of Endohedral Metallofullerenes TM@C28 (TM = Sc−, Y−, La−, Ti, Zr, Hf, V+, Nb+, Ta+), Inorganics, 12, 40 (2024) https://doi.org/10.3390/inorganics12020040
- Wenfei Ren, Jiguang Li, Baiyi Zu, et al., Design of Highly Efficient Electronic Energy Transfer in Functionalized Quantum Dots Drived Specifically by Ethylenediamine, JACS Au (2024) https://pubs.acs.org/doi/full/10.1021/jacsau.3c00667
- Xueshuang Zhao, Yunzhou Lin, Linbing Huang, Lihang Chen, Theoretical study on formation mechanism of acetic acid associating configurations and their distributions under saturated conditions, J. Mol. Model., 30, 49 (2024) https://link.springer.com/article/10.1007/s00894-024-05850-2
- Yangxia Han, Manchang Kou, Kaijun Quan, et al., Enantioselective Glutamic Acid Discrimination and Nanobiological Imaging by Chiral Fluorescent Silicon Nanoparticles, Anal. Chem. (2024) https://pubs.acs.org/doi/abs/10.1021/acs.analchem.3c05150
- Xuehui Zhang, Hong Liu, Xiaochong Liu, et al., Study of molecular mechanism and extraction performance evaluation for separation of phenolics from alkaline wastewater through synergistic extraction, Energy Sources A, 46, 2180 (2024) https://www.tandfonline.com/doi/abs/10.1080/15567036.2023.2298005
- Lei Miao, Shu-Ting Wang, Quan-Hao Wei, et al., Bioactive monoterpenoids and acetophenones from the aerial parts of Eupatorium fortunei, Phytochem. (2024) https://www.sciencedirect.com/science/article/pii/S0031942224000219
- Xu-Hui Li, Yuan-Yuan Cui, Shi-Lei Ji, et al., Amide and carboxyl dual-functionalized magnetic microporous organic networks for efficient extraction of cephalosporins, Food Chem. (2024) https://www.sciencedirect.com/science/article/pii/S0308814624002073
- Cong Liu, Ying Zou, Manli Zhang, et al., A simple strategy for d/l-carnitine analysis in food samples using ion mobility spectrometry and theoretical calculations, Food Chem. (2024) https://www.sciencedirect.com/science/article/pii/S0308814624001055
- Zhen Yuan, Xingyu Chen, Sisi Fan, et al., Binding Free Energy Calculation Based on the Fragment Molecular Orbital Method and Its Application in Designing Novel SHP-2 Allosteric Inhibitors, Int. J. Mol. Sci., 25, 671 (2024) https://doi.org/10.3390/ijms25010671
- Bo Han, Shuai Li, Di Chang, et al., Efficient phenyl phosphate ester extractant synthesis and solvent extraction performance evaluation for transition metals, Sep. Purif. Technol., 336, 126251 (2024) https://www.sciencedirect.com/science/article/pii/S1383586623031593
- Jingwen Zhou, Xiuliang Sun, Tianpeng Chen, et al., Understanding Structure, Hydrogen Bonding, and Hydrogen Transfer in 1,3-Disubstituted Imidazolium Aggregations: A Density Functional Theory Study, J. Solut. Chem. (2024) https://link.springer.com/article/10.1007/s10953-023-01346-4
- Xiaoyue Xu, Thomas Rades, Holger Grohganz, Molecular interactions of hydrated co-amorphous systems of prilocaine and lidocaine, Int. J. Pharm., 651, 123807 (2024) https://www.sciencedirect.com/science/article/pii/S0378517324000413
- Hao Chen, Xiaofeng Xie, Chaofang Li, et al., Steering electrophilic N center in ionic liquid for efficient oxidative capture of elemental mercury, Chem. Eng. J. (2024) https://www.sciencedirect.com/science/article/pii/S138589472307170X
- Yujia Li, Erping Bi, Influencing mechanisms of tartaric acid on adsorption and degradation of tetracycline on goethite: insight from solid and liquid aspects, Environ. Sci. Pollut. Res. (2023) https://link.springer.com/article/10.1007/s11356-023-31465-w
- Rui Zhang, Dong-Dong Wang, Lian-Yu Tang, et al., Salvirrane A-F, six undescribed nordrimane sesquiterpene derivatives from Salvia castanea Diels f. tomentosa Stib and their cytotoxic activities, Phytochem., 218, 113958 (2024) https://www.sciencedirect.com/science/article/pii/S0031942223003746
- Minghao Song, Fei Zhao, Shuqing Liu, et al., Experimental and Mechanistic Analysis of Chlorinated Volatile Organic Compound Capture by Quaternary Ammonium-Based Deep Eutectic Solvents, Ind. Eng. Chem. Res. (2023) https://pubs.acs.org/doi/abs/10.1021/acs.iecr.3c03838
- Yu Wang, Liwen Zhuang, Yang Wang, et al., Unraveling hydrogen bond interactions between ketoconazole and imidazolium-based ionic liquids: An experimental and theoretical study, J. Mol. Liq. (2023) https://www.sciencedirect.com/science/article/pii/S0167732223026909
- Zhou Kangxi, Wu Li, Lu Xucong, Ni Li, Studies on Quantum Chemical Simulation and Adsorption Properties of Citrinin Molecularly Imprinted Preassembly System, J. Chin. Inst. Food Sci. & Technol., 23, 361 (2023) https://doi.org/10.16429/j.1009-7848.2023.11.034
- Zihao Dong, Yajuan Qu, Yuyang Jiao, et al., Temperature response behavior of poly(1-vinyl-3-methylimidazole dimethyl phosphate-co-N-isopropylacrylamide) in aqueous and ester solutions driven by hydrogen bonding, J. Mol. Liq. (2023) https://www.sciencedirect.com/science/article/pii/S0167732223026557 (此文没有按要求引用Molclus!)
- Xin Su, Yijia Zhou, Lijia Liu, et al., Phytate-modified MOFs grown on carbon nanotubes for efficient adsorption of uranium(Ⅵ) from seawater, Sep. Purif. Technol. (2023) https://www.sciencedirect.com/science/article/pii/S1383586623030101
- Xueru Wang, Liyuan Deng, Enabling cation-exchange-based sorbents for high-capacity and efficient moisture-swing direct air capture of CO2, ResearchSquare (2023) https://www.researchsquare.com/article/rs-3772049/v1
- Wei Wu, Huaifu Ge, Qingqing Zhou, et al., A method to improve dye-uptakes of less soluble disperse dyes for polyester fibers in supercritical CO2 fluid: A theoretical and experimental study, J. Supercrit. Fluids (2023) https://www.sciencedirect.com/science/article/pii/S0896844623003169
- Zhou Feng, Ning Wang, Xiaohan Li, et al., Process optimization, energy consumption analysis, and environmental assessment of total CO2 capture from syngas based on [NEMH][Ac] protic ionic liquid, Int. J. Greenhouse Gas Control, 131, 104037 (2024) https://www.sciencedirect.com/science/article/pii/S1750583623002074
- Feng-Zhi Shi, Yin-Dong Fang, Min Fan, et al., Cytotoxic depsidones and xanthones from Garcinia esculenta Y. H. Li, Fitoterapia (2023) https://www.sciencedirect.com/science/article/abs/pii/S0367326X23003544 (此文没有按要求引用Molclus!)
- Chaoyu Wang, Chaoyang Zhang, Bo You, Xianggui Xue, Exploring the High-Pressure Polymorphs of Flexible Molecules by Crystal Structure Prediction: A Case of 1,3,5-Trinitro-1,3,5-Triazinane, Cryst. Growth Des. (2023) https://pubs.acs.org/doi/abs/10.1021/acs.cgd.3c00709
- Xiaokang Wang, Yuanyuan Cui, Junping Zhang, et al., Enhanced extraction of methanol and dimethyl carbonate by hydrogen bond breaking-reconstruction with deep eutectic solvents, J. Mol. Liq., 394, 123761 (2024) https://www.sciencedirect.com/science/article/abs/pii/S0167732223025680 (此文没有按要求引用Molclus!)
- Qi An, Na Li, Zhehui Zhao, et al., Two Novel Metformin Carboxylate Salts and the Accidental Discovery of Two 1,3,5-Triazine Antihyperglycemic Agent, ACS Omega (2023) https://pubs.acs.org/doi/full/10.1021/acsomega.3c06721
- Wanxiang Zhang, Feifei Lv, Yaozhi Zhang, et al., Experimental and molecular insights on the separation of indole from wash oil by tetraethylammonium-based ionic liquids, AIChE J. (2023) https://aiche.onlinelibrary.wiley.com/doi/abs/10.1002/aic.18311
- Zhihan Tong, Suqing Zeng, Xiaona Li, et al., Glycosidic bond protection of cellulose during solvent dissolution by coordination interaction competition strategy, Carbohydr. Polym. (2023) https://www.sciencedirect.com/science/article/abs/pii/S014486172301130X
- Minghao Song, Fei Zhao, Shuqing Liu, et al., Multi-scale simulation and study of volatile phenols removal from simulated oil by ionic liquids, CIESC J., 74, 3654 (2023) https://hgxb.cip.com.cn/CN/10.11949/0438-1157.20230634
- Kangkang Li, Huanong Cheng, Xiaohan Li, et al., Molecular mechanism-based extractant screening and process design for the separation of methanol/dimethyl carbonate azeotrope, Sep. Purif. Technol., 333, 125916 (2024) https://www.sciencedirect.com/science/article/abs/pii/S1383586623028241
- Yihua Fu, Yan Zhang, Changwei Hu, Zhishan Su, Mechanistic study on coupling of CO2 and epoxide mediated by guanidine/TBAI catalyst, New J. Chem. (2023) https://pubs.rsc.org/en/content/articlelanding/2023/nj/d3nj04395a/
- Hanyu Wu, Jiabin Yin, Xunda Feng, Boosting Phosphoric Acid Retention in Polymer Electrolyte Membranes by Zwitterions: Insights from DFT Calculations and MD Simulations, J. Phys. Chem. B (2023) https://pubs.acs.org/doi/abs/10.1021/acs.jpcb.3c05719
- Wenshuo Zhang, Hairong Zhang, Jiaxin Li, et al., PVP-capped silver nanoparticles for efficient SERS detection of adenine based on the stabilizing and enrichment roles of PVP, Microchim. Acta, 191, 1 (2024) https://link.springer.com/article/10.1007/s00604-023-06047-9 (此文没有按要求引用Molclus!)
- Zhaoyan Zhang, Yingqi Zhao, Ya Zhao, et al., Effects of isoprene on the ozonolysis of Δ3-carene and β-caryophyllene: Mechanisms of secondary organic aerosol formation and cross-dimerization, J. Environ. Sci. (2023) https://www.sciencedirect.com/science/article/abs/pii/S1001074223005259
- Ernest C. Agwamba, Maxwell-Borjor A. Eba, Ibtehaj F. Alshdoukhi, et al., Molecular modeling of Si60 fullerene and Nb-doped Si60 fullerene nanomaterials for SO2, NO2 and CO2 gas sensing, Mater. Sci. Eng. B, 299, 117022 (2024) https://www.sciencedirect.com/science/article/abs/pii/S092151072300764X (此文没有按要求引用Molclus!)
- Pengcheng Liu, Jia Han, Yan Chen, et al., Carbon Dioxide Activation by Discandium Dioxide Cation in the Gas Phase: A Combined Investigation of Infrared Photodissociation Spectroscopy and DFT Calculations, Phys. Chem. Chem. Phys. (2023) https://pubs.rsc.org/en/content/articlelanding/2023/cp/d3cp04995g/
- Peipei Li, Menghang Li, Bing Sun, et al., Integrated Three-Dimensional Microdevice with a Modified Surface for Enhanced DNA Separation from Biological Samples, ACS Appl. Mater. Interfaces (2023) https://pubs.acs.org/doi/abs/10.1021/acsami.3c11681 (此文没有按要求引用Molclus!)
- Qiang Wu, Yubin Yuan, Xuming Wang, et al., Highly Selective Ionic Gel-Based Gas Sensor for Halogenated Volatile Organic Compound Detection: Effect of Dipole–Dipole Interaction, ACS sens. (2023) https://pubs.acs.org/doi/abs/10.1021/acssensors.3c01476
- Jiangli Fan, Yingchao Chen, Tao Xiong, et al., Self-Reporting Photodynamic Nanobody Conjugate for Precise and Sustainable Large-Volume Tumor Treatment, ResearchSquare (2023) https://www.researchsquare.com/article/rs-3598991/v1 (此文没有按要求引用Molclus!)
- Naitian Zhang, Yuehui Li, Wenzhe Shang, et al., Role of excited-state hydrogen bonding in CO2 photoreduction catalyzed by sodium magnesium chlorophyll, Phys. Chem. Chem. Phys. (2023) https://pubs.rsc.org/en/content/articlelanding/2023/cp/d3cp03638c/
- Ling-Feng Qin, Huan-Huan Gao, Xu Zhang, et al., Seventeen undescribed iridoid derivatives with anti-inflammatory effects from Hedyotis diffusa and their structure-activity relationships, Phytochem., 217, 113904 (2024) https://www.sciencedirect.com/science/article/abs/pii/S0031942223003205 (此文没有按要求引用Molclus!)
- Hongwei Jin, Yun Teng, Kangkang Li, et al., Absorption characteristics, model, and molecular mechanism of hydrogen sulfide in morpholine acetate aqueous solution, Chin. J. Chem. Eng. (2023) https://www.sciencedirect.com/science/article/abs/pii/S1004954123002732
- Jun Xiao, Xiaoyu Li, Hailan Lian, et al., Deep eutectic solvents for effective removal of indoor formaldehyde: Theoretical design, experiment, and adsorption mechanism study, J. Mol. Liq., 392, 123473 (2023) https://www.sciencedirect.com/science/article/abs/pii/S0167732223022791
- Eduardo Alejandro Romero Montalvo, Elucidating the “On Water” Mechanism in Accelerated Reactions in Water, PhD thesis of THE UNIVERSITY OF BRITISH COLUMBIA (2023) https://open.library.ubc.ca/media/download/pdf/24/1.0437519/3
- Haina Zhang, Wenjing Guo, Wenqing Du, et al., A Metal-Free Molecular Ferroelectric [4-Me-cyclohexylamine]ClO4 Introduced by Boat and Chair Conformations of Cyclohexylamine, Chem. Eur. J. (2023) https://chemistry-europe.onlinelibrary.wiley.com/doi/abs/10.1002/chem.202302671
- Xiaoyan Feng, Jiachen Li, Yaotian Peng, et al., Zwitterionic polymers as high-performance coatings for hemoperfusion adsorbents and their chemisorption of protein-bound toxins through computational simulations, Coll. Surf.A, 680, 132710 (2024) https://www.sciencedirect.com/science/article/abs/pii/S0927775723017946
- Yunhai Huang, Na Wang, Jingkang Wang, et al., Mechanistic study on metastable and stable liquid–liquid phase separation explored by molecular simulation and thermodynamic analysis, Chem. Eng. J., 284, 119464 (2024) https://www.sciencedirect.com/science/article/abs/pii/S0009250923010205
- Tian Meng, Jingwei Chen, Jiaqiang E, et al., Molecular dynamics study on the dissolution characteristics and cluster formation of Li2CO3 in supercritical water, J. Mol. Liq., 392, 123454 (2023) https://www.sciencedirect.com/science/article/abs/pii/S0167732223022602
- Yawen Yuan, Shasha Liu, Shideng Yuan, et al., Novel insights into the effects of asphaltenes on oil phase properties: Crude oil, oil-water interface, and graphene oxide synergistic demulsification, J. Mol. Liq., 391, 123405 (2023) https://www.sciencedirect.com/science/article/abs/pii/S0167732223022110 (此文没有按要求引用Molclus!)
- Tomsmith O. Unimuke, Idongesit J. Mbonu, Hitler Louis, et al., Surface engineering of non-platinum-based electrocatalysts for sustainable hydrogen production: Encapsulation, doping, and decoration approach, Int. J. Hyd. Energy (2023) https://www.sciencedirect.com/science/article/abs/pii/S036031992305245X
- Xingguo Wei, Lingrui Cui, Jun Xu, et al., Effective electrostatic potential surface and aromatic affinity as quantitative guide for deep extraction denitrification from aromatic-rich oils, Sep. Purif. Technol. 330, 125476 (2024) https://www.sciencedirect.com/science/article/abs/pii/S1383586623023845
- Yuhui Li, Xinlang Yang, Yuanqin Yu, et al., Dependence of Intramolecular Hydrogen Bond on Conformational Flexibility in Linear Aminoalcohols, J. Phys. Chem. A (2023) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.3c04674
- Feng Lin, Zhaoyang Ju, Yao Li, et al., Hydroxyl-groups engineering of triazole-based polymers for efficient photocatalytic hydrogen peroxide production, J. Clean. Prod., 428, 139371 (2023) https://www.sciencedirect.com/science/article/abs/pii/S0959652623035291
- Na Liu, Zijian Han, Yunxiang Lu, et al., Dimeric cation-cation aggregates stabilized by 2Ch-2N chalcogen bonds: crystallographic and theoretical evidences, Chem. Phys. (2023) https://www.sciencedirect.com/science/article/abs/pii/S0301010423002951
- Jiangang Li, Guohui Xing, Jilong Wu, et al., Photoirradiation-Gated Excitation-Dependent Room-Temperature Phosphorescence in Through-Space Charge Transfer Molecules, Laser Photonics Rev. (2023) https://onlinelibrary.wiley.com/doi/abs/10.1002/lpor.202300704
- Qing-Ru Liu, Xian-Li Lin, Zhen-Ming Lu, et al., Influence on the volatilization of ethyl esters: Nonnegligible role of long-chain fatty acids on Baijiu flavor via intermolecular interaction, Food Chem. (2023) https://www.sciencedirect.com/science/article/abs/pii/S030881462302349X
- Haiyang Gao, Mo Xiong, Chuncai Kong, et al., A theoretical study on hydrated sodium ion-phenylalanine clusters Na+(Phe)(H2O)n (n = 0-6; Phe = phenylalanine), Phys. Chem. Chem. Phys. (2023) https://pubs.rsc.org/en/content/articlelanding/2023/cp/d3cp03144f/
- Xiaocong Yao, Ji Mu, Yi Zheng, et al., Tailoring the adsorption behaviors of flucytosine on BnNn (n = 12, 16, 20, and 24) nanocage scaffolds: A computational insight on drug delivery applications, Coll. Surf. A (2023) https://www.sciencedirect.com/science/article/abs/pii/S0927775723015650
- Yongjie Dan, Huan Luo, Pengjian Gong, et al., Structural, Energetic and Dynamic Investigation of Poly(ethylene oxide) in Imidazolium-Based Ionic Liquids with Different Cationic Structures, Phys. Chem. Chem. Phys. (2023) https://pubs.rsc.org/en/content/articlelanding/2023/cp/d3cp01946b/
- Wanxiang Zhang, Guoxuan Li, Zhengrun Chen, et al., Efficient capture of benzene and its homologues volatile organic compounds with protic [MIM][NTF2] and aprotic [EMIM][NTF2] ionic liquids: Experimental and computational thermodynamics, J. Environ. Chem. Eng., 11, 111124 (2023) https://www.sciencedirect.com/science/article/abs/pii/S2213343723018638
- Sijin Zhou, Yameng Hou, Xianglei Kong, Structural Diversity of Protonated Citric Acid-Ammonia Clusters and Its Atmospheric Implication, J. Phys. Chem. A (2023) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.3c05160
- Lincai Peng, Weipeng Song, Junhua Zhang, et al., Solvent-promoted selective hydroxymethylation of biomass-derived furan compounds, ResearchSquare (2023) https://doi.org/10.21203/rs.3.rs-3266097/v1 (此文没有按要求引用Molclus!)
- Beiqian Tian, Hongxun Hao, Xin Huang, et al., Solvent Effect on Molecular Conformational Evolution and Polymorphic Manipulation of Cimetidine, Cryst. Growth Des. (2023) https://pubs.acs.org/doi/abs/10.1021/acs.cgd.3c00661
- Suocheng Chi, Yingzhe Yu, Minhua Zhang, Theoretical Insights into Midchain Radicals and Branching Characteristics in Solution Copolymerization of Ethylene and Vinyl Acetate, J. Phys. Chem. B (2023) https://pubs.acs.org/doi/abs/10.1021/acs.jpcb.3c04570
- Zu-Jia Lu, Yong Hu, Wen-Shuai Dong, et al., From Concept to Synthesis: Developing Heat-Resistant High Explosives through Automated High-Throughput Virtual Screening, J. Phys. Chem. C (2023) https://pubs.acs.org/doi/abs/10.1021/acs.jpcc.3c04039
- Qilei Liu, Sheng Xiang, Jian Du, et al., Improved Prediction of Reaction Kinetics for Amine Absorbent-based Carbon Capture using Reactive Site-based Transition State Conformer Search Method, ChemRxiv (2023) https://chemrxiv.org/engage/chemrxiv/article-details/64fe6bd899918fe537bad226
- Jingang He, Yunxiang Dai, Jinfeng Zhong, et al., Difference in the complexation of cholesterol with β-cyclodextrin derivatives: A combined theoretical and experimental study, Food Chem. (2023) https://www.sciencedirect.com/science/article/abs/pii/S0308814623020770
- Junfeng Teng, Fang Zong, Zhe Zhang, et al., Measurement, Correlation, and Analysis of the Solubility of Triethylamine Hydrochloride in ten Pure Solvents, J. Mol. Liq. (2023) https://www.sciencedirect.com/science/article/abs/pii/S0167732223018469
- Yanrong Jiang, Zhubin Hu, Cheng Zhong, et al., Locking water molecules via ternary O-H···O intramolecular hydrogen bonds in perhydroxylated closo-dodecaborate, Phys. Chem. Chem. Phys. (2023) https://pubs.rsc.org/en/content/articlelanding/2023/cp/d3cp03555g/
- Qiaolong Zhai, Shaoqi Yang, Shuheng Zhao, et al., Fractionation of poplar wood with different acid hydrotropes: Lignin dissolution behavior and mechanism evaluation, Int. J. Biol. Macromol. (2023) https://www.sciencedirect.com/science/article/abs/pii/S0141813023035936 (此文没有按要求引用Molclus!)
- Kaicheng Du, Xingyao Li, Changwei Zheng, et al., Monoterpenoid indole alkaloid dimers from the Melodinus axillaris induce G2/M phase arrest and apoptosis via p38 MAPK activation in HCT116 cells, Bioorg. Chem., 140, 106841 (2023) https://www.sciencedirect.com/science/article/abs/pii/S0045206823005023
- Yuhui Ci, Tianying Chen, Feiyun Li, et al., Cellulose dissolution and regeneration behavior via DBU-levulinic acid solvents, Int. J. Biol. Macromol. (2023) https://www.sciencedirect.com/science/article/abs/pii/S014181302303444X
- Bi-Bo Li, Shu-Hai Zhang, Rui-Jun Gou, et al., DFT study on the intermolecular interaction between two energetic ionic salts: a case of TKX-50 and ADN, Chem. Papers (2023) https://link.springer.com/article/10.1007/s11696-023-03022-9
- Qixuan Lin, Qiwen Zhan, Yue Wu, et al., Molecular scale behavior of xylan during solvent-controlled extraction and precipitation, Phys. Chem. Chem. Phys. (2023) https://pubs.rsc.org/en/content/articlelanding/2023/cp/d3cp01385e/
- Haiming Hua, Xueying Yang, Peng Zhang, Jinbao Zhao, Understanding the Influence of Functional Groups on the PEO Composite Electrolytes through a Theoretical, Spectroscopic, and Conduction Behavior Study of Model Systems, J. Phys. Chem. C (2023) https://pubs.acs.org/doi/abs/10.1021/acs.jpcc.3c02823
- Yanjing Li, Shilong Dong, Lili Wang, et al., The extraction of aromatics using n-methylpyrrolidone: Liquid-liquid equilibrium determination and mechanism exploration, Chin. J. Chem. Eng. (2023) https://www.sciencedirect.com/science/article/abs/pii/S1004954123002100
- Zhihan Zheng, Yicheng Xu, Hang Qu, et al., Enhancement of anti-diabetic activity of pomelo peel by the fermentation of Aspergillus oryzae CGMCC23295: In vitro and in silico docking studies, Food Chem., 432, 137195 (2024) https://www.sciencedirect.com/science/article/abs/pii/S0308814623018137
- Jian Gao, Zhengning Xu, Runlong Cai, et al., Molecular identification of organic acid molecules from α-pinene ozonolysis, Atmosph. Environ. (2023) https://www.sciencedirect.com/science/article/abs/pii/S1352231023004788
- Design, Synthesis and Characterization of Ferrocene Containing Aromatic Foldamers & Synthesis and Study of Novel Indole Based Chalcones for Anti-cancer Activities, dissertation of Université catholique de Louvain (2023) https://dial.uclouvain.be/pr/boreal/object/boreal%3A263406/datastream/PDF_01/view
- Guoao Li, Zhenxing Li, Liuzhou Gao, et al., Combined Molecular Dynamics and Coordinate Driving Method for Automatically Searching Complicated Reaction Pathways, Phys. Chem. Chem. Phys. (2023) https://pubs.rsc.org/en/content/articlehtml/2023/cp/d3cp02443a
- Xiaomeng Zhang, Yongjian Lian, Shendong Tan, Shi Yin, et al., Organosulfate Produced from Consumption of SO3 Speeds up Sulfuric Acid-Dimethylamine Atmospheric Nucleation, EGUsphere Preprint (2023) https://egusphere.copernicus.org/preprints/2023/egusphere-2023-1649/egusphere-2023-1649.pdf
- Ge Gao, Bin Xu, Xiaoyi Gao, et al., New insights into the structure-activity relationship for CO2 capture by tertiary amines from the experimental and quantum chemical calculation perspectives, Chem. Eng. J. (2023) https://www.sciencedirect.com/science/article/abs/pii/S1385894723040081
- Xiaohu Zhou, Ende Huang, Licheng Zhong, et al., Vibrational spectroscopy of neutral and cationic 2-Methoxyethanol, J. Mol. Struct., 1294, 136389 (2023) https://www.sciencedirect.com/science/article/abs/pii/S0022286023014795
- Chao Sun, Yajuan Qu, Yasen Dai, et al., Comparative study on ionic liquids and deep eutectic solvents in the separation of fuel additive isopropyl alcohol and ethyl acetate by the experimental study and molecular simulation, Fuel, 354, 129397 (2023) https://www.sciencedirect.com/science/article/abs/pii/S0016236123020112
- Mengjin Zhou, Yanli Zhang, Ke Xue, et al., Separation of fuel additives based on mechanism analysis and thermodynamic phase behavior, Chin. J. Chem. Eng. (2023) https://doi.org/10.1016/j.cjche.2023.05.015
- Luis A. Desales-Guzmán, Elizabeth Vazquez-Rivas, Juan H. Pacheco Sánchez, Ab Initio Study on the Potential of Metal-Decorated Pristine and Activated Carbyne for Hydrogen Storage Applications, Energy Fuels (2023) https://pubs.acs.org/doi/abs/10.1021/acs.energyfuels.3c01559
- Chun-Hong Hu, Yueqian Sang, Ya-Wei Yang, et al., Organic charge-transfer complex induces chemoselective decarboxylation to aryl radicals for general functionalization, Chem (2023) https://www.cell.com/chem/pdf/S2451-9294(23)00325-X.pdf
- Kaiming Kang, Xiaoqiao Jia, Keyang Zheng, et al., Physical properties of natural deep eutectic solvent and its application in remediation of heavy metal lead in soil, J. Contam. Hydrol., 258, 104222 (2023) https://www.sciencedirect.com/science/article/abs/pii/S016977222300092X
- Wenjin Cao, Qinqin Yuan, Hanhui Zhang, et al., How generic is iodide-tagging photoelectron spectroscopy: An extended investigation on the Gly·X− (Gly = glycine, X = Cl or Br) complexes, J. Chem. Phys., 159, 034305 (2023) https://pubs.aip.org/aip/jcp/article/159/3/034305/2903069
- Zhiye Wang, Bingling Dai, Yuming Su, et al., Why Ligand Doping Increases the Fluorescence of an Anthracene-Based Metal–Organic Framework, Inorg. Chem. (2023) https://pubs.acs.org/doi/abs/10.1021/acs.inorgchem.3c01036
- Yanbo Liu, Jinbo Ouyang, Xuemei Wei, et al., Prediction of the Phase Diagram of Liquid-liquid Phase Separation during Crystallization via a Molecular Mechanism study, J. Mol. Liq. (2023) https://www.sciencedirect.com/science/article/abs/pii/S0167732223014381
- Yingqi Tang, Nannan Li, Hao Li, et al., Probing the Water Mediated Proton Transfer in Histidine Tautomerization, J. Mol. Liq. (2023) https://www.sciencedirect.com/science/article/abs/pii/S0167732223014435
- Zujin Bai, Jun Deng, Caiping Wang, et al., Mechanism of [BMIM][BF4] inhibiting coal groups activity from experiments and DFT calculations, Fuel, 353, 129175 (2023) https://www.sciencedirect.com/science/article/abs/pii/S0016236123017891
- Jia Han, Lei Wang, Wenjin Cao, et al., Photogeneration of Singlet Oxygen Catalyzed by Hexafluoroisopropanol for Selective Degradation of Dyes, ISCIENCE (2023) https://doi.org/10.1016/j.isci.2023.107306
- Yuru Zhang, Jia Wen, Yichen Zhou, et al., Novel efficient capture of hexavalent chromium by polyethyleneimine/amyloid fibrils/polyvinyl alcohol aerogel beads: Functional design, applicability, and mechanisms, J. Hazard. Mater., 458, 132017 (2023) https://www.sciencedirect.com/science/article/abs/pii/S0304389423013006 (此文没有按要求引用Molclus!)
- Weiwei Han, Jiabao Fan, Taotao Qiang, et al., A novel salt and condensate–resistant foam co-stabilized by mixtures of surfactants and citric acid for gas well deliquification, J. Mol. Liq., 385, 122426 (2023) https://www.sciencedirect.com/science/article/abs/pii/S0167732223012308
- Xinyan Li, Wei Cao, Evaluating the effects of oxidation and the deagglomerating potential of selected additives on asphaltene aggregation via computational modeling, Constr. Build. Mater., 394, 132291 (2023) https://www.sciencedirect.com/science/article/abs/pii/S095006182302007X
- Qiyang Lin, Shuai Wang, Xin Ma, et al., O-Isobutyl-N-hydroxyethyl Thionocarbamate: Molecular Behavior and Flotation Mechanism to Chalcopyrite, Ind. Eng. Chem. Res. (2023) https://pubs.acs.org/doi/abs/10.1021/acs.iecr.3c01029
- Kexin Yin, Yanan Shang, Dongdong Chen, et al., Redox potentials of pollutants determining the dominate oxidation pathways in manganese single-atom catalyst (Mn-SAC)/peroxymonosulfate system: selective catalytic mechanisms for versatile pollutants, Appl. Catal. B (2023) https://www.sciencedirect.com/science/article/abs/pii/S0926337323006720
- Qing-Wei Meng, Shaochun Wu, Mingjie Liu, et al., Guanidinium-based covalent organic framework membrane for single-acid recovery, Sci. Adv. (2023) https://www.science.org/doi/epdf/10.1126/sciadv.adh0207
- Rong Zhang, Ning Duan, Linhua Jiang, et al., Electronic Excitation Characteristics and Spectral Behavior of Phosphate Anions, Langmuir (2023) https://pubs.acs.org/doi/abs/10.1021/acs.langmuir.3c00847
- Guochun Lv, Jiaoxue Yang, Xiaomin Sun, et al., Understanding the atmospheric transformation mechanism of an emerging fluorinated alcohol (FESOH), Atmosph. Environ., 309, 119903 (2023) https://www.sciencedirect.com/science/article/abs/pii/S1352231023003291
- Baoping Zhu, Yang Xu, Hanwen Ge, et al., Theoretical study of lactic acid-based deep eutectic solvents dissociation of hemicellulose with different hydrogen bonding acceptors, Int. J. Biol. Macromol., 244, 125342 (2023) https://www.sciencedirect.com/science/article/abs/pii/S0141813023022365 (此文没有按要求引用Molclus!)
- Huachao Yang, Yiheng Qi, Zifan Wang, et al., Sodium Nitrate/Formamide Deep Eutectic Solvent as Flame-Retardant and Anticorrosive Electrolyte Enabling 2.6 V Safe Supercapacitors with Long Cyclic Stability, Energy Environ. Mater. (2023) https://onlinelibrary.wiley.com/doi/10.1002/eem2.12641 (此文没有按要求引用Molclus!)
- Yifan Zhang, Xianwei Hu, Wendi Zhang, et al., Study on Decomposition Mechanism of K3AlF6 by Quantum Chemical Calculations, Russ. J. Phys. Chem. A, 97, 997 (2023) https://link.springer.com/article/10.1134/S0036024423050308
- Wanxiang Zhang, Zhigang Lei, Yan Wang, et al., Molecular mechanism and process of efficient separation of benzene and cyclohexane using a low-cost deep eutectic solvent, Chem. Eng. Sci., 279, 118963 (2023) https://www.sciencedirect.com/science/article/abs/pii/S0009250923005195 (此文没有按要求引用Molclus!)
- Chao Sun, Jiafu Xing, Yajuan Qu, et al., Liquid–Liquid Equilibrium Measurements and Interaction Explorations for Separation of Dimethyl Carbonate and Isopropyl Alcohol Using ChCl-Based Deep Eutectic Solvents, Ind. Eng. Chem. Res. (2023) https://pubs.acs.org/doi/abs/10.1021/acs.iecr.3c01045
- Xionghui Ma, Jinsheng Kang, Yuwei Wu, et al., A bifunctional polycentric-affinity MOF/MXene heterojunction-based molecularly imprinted photoelectrochemical organophosphorus-sensing platform, Chem. Eng. J. (2023) https://www.sciencedirect.com/science/article/abs/pii/S1385894723026190
- Benjamin List, Jung Tae Han, Hui Zhou, Brønsted Acid Catalyzed Asymmetric Silylation of Biaryl Diols, Synlett (2023) https://www.thieme-connect.com/products/ejournals/pdf/10.1055/a-2100-1575.pdf
- Jinyun Zheng, Xinxin Liu, Wenbin Li, et al., Green Synthesis of Novel Conjugated Poly (perylene diimide) as Cathode with Stable Sodium Storage, NanoResearch (2023) https://www.sciopen.com/article_pdf/1661634992397860865.pdf
- Wei Liu, Zexin Wang, Nannan Chen, et al., A novel insight into the dual state emission mechanism of a new rofecoxib derivative and its multi-functional applications, J. Photochem. Photobiol. A, 443, 114868 (2023) https://www.sciencedirect.com/science/article/abs/pii/S1010603023003337
- Chen Yu, Degang Li, Wenyuan Han, et al., Improvement of photostability and thermal stability of PVC by carbon quantum dots loaded on TiO2, Vinyl Add. Technol. (2023) https://4spepublications.onlinelibrary.wiley.com/doi/abs/10.1002/vnl.22012
- Norberto Monteiro, Richele Machado, Modeling the adsorption mechanism of 3-tertiary- butyl-4-hydroxyanisole (3BHA) on polyethylene and polypropylene microplastics., ResearchSquare (2023) https://doi.org/10.21203/rs.3.rs-2943457/v1
- Xukang Jin, Qi Duan, Xiuting Li, et al., Dissolution of Curcumin by Deep Eutectic Solvent of Fatty Acids, J. East China Univ. Sci. and Technol. (2023) https://journal.ecust.edu.cn/cn/article/doi/10.14135/j.cnki.1006-3080.20230228001
- Yuling Zhang, Hongwei Ren, Baochai Li, et al., Mechanistic insights into the lignin dissolution behavior in amino acid based deep eutectic solvents, Int. J. Biol. Macromol. (2023) https://www.sciencedirect.com/science/article/abs/pii/S0141813023017233
- Da Sheng, Lingjun Bu, Shumin Zhu, et al., Pre-oxidation coupled with charged covalent organic framework membranes for highly efficient removal of organic chloramines precursors in algae-containing water treatment, Chemosphere, 333, 138982 (2023) https://www.sciencedirect.com/science/article/abs/pii/S0045653523012493
- Fawze Alnadari, Sam Al-Dalali, Fei Pan, et al., Physicochemical Characterization, Molecular Modeling, and Applications of Carboxymethyl Chitosan-Based Multifunctional Films Combined with Gum Arabic and Anthocyanins, Food Bioproc. Technol. (2023) https://link.springer.com/article/10.1007/s11947-023-03122-0
- Qing Sun, Hang Chen, Jianguo Yu, A DFT investigation of the lithium extraction process under different diluent environments, Chem. Eng. Sci., 277, 118857 (2023) https://www.sciencedirect.com/science/article/abs/pii/S000925092300413X
- Huikun Zhang, Weiqiang Tang, Peng Xie, Shuangliang Zhao, Demystifying Solvent Effects on Diels–Alder Reactions in Pure and Mixed Solvents: A Combined Electronic DFT and QM/MM Study, Ind. Eng. Chem. Res. (2023) https://pubs.acs.org/doi/abs/10.1021/acs.iecr.3c01033
- Pan Xu, Zhijie Shang , Guoxuan Li, et al., Efficient purification of biogas using ionic liquid as absorbent: Molecular thermodynamics, dynamics and experiment, J. Environ. Chem. Eng., 11, 110083 (2023) https://www.sciencedirect.com/science/article/abs/pii/S2213343723008229 (此文没有按要求引用Molclus!)
- Ikechukwu C. Nwobodo, Hitler Louis, Tomsmith O. Unimuke, et al., Molecular Simulation of the Interaction of Diclofenac with Halogen (F, Cl, Br)-Encapsulated Ga12As12 Nanoclusters, ACS Omega (2023) https://pubs.acs.org/doi/full/10.1021/acsomega.2c06097 (此文没有按要求引用Molclus!)
- Sanbao Dong, Jiabao Fan, Chenwei Liu, et al., Experimental and computational investigations on foaming properties of anionic-nonionic-zwitterionic surfactants and amino acid compounds to address the liquid loading of natural gas wells, J. Mol. Liq., 382, 122016 (2023) https://www.sciencedirect.com/science/article/abs/pii/S016773222300819X
- Xiaolan Qin, Yue Xu, Boxuan Lou, et al., Enhanced Direct Crystallization Resolution of Racemates Mediated in Chiral Ionic Liquid Cosolvent Mixtures: Experimental, Statistical, and QM Simulation Studies, Cryst. Growth Des. (2023) https://pubs.acs.org/doi/abs/10.1021/acs.cgd.3c00364
- Yu Gao, Zhi-Bin Lu, Theoretical insight into the influence of different molecular design strategies on photovoltaic properties for a series of POM-based dyes applied in dye-sensitized solar cells, New J. Chem. (2023) https://pubs.rsc.org/en/content/articlelanding/2023/nj/d3nj00823a/
- Pan Xu, Zhijie Shang, Guoxuan Li, et al., Biomethane purification with quaternary ammonium salts-based deep eutectic solvents: Experiment and computational thermodynamics, Sep. Purif. Technol., 318, 123980 (2023) https://www.sciencedirect.com/science/article/abs/pii/S1383586623008882
- Qinghua Liu, Chengmin Gui, Guoxuan Li, Zhigang Lei, Investigation of Ionic Liquids as Extraction Solvents for Separating Bicyclic Aromatic S/N-Compounds from FCC Diesel, ACS Sustainable Chem. Eng. (2023) https://pubs.acs.org/doi/abs/10.1021/acssuschemeng.3c01103
- Wen-yu Zheng, Xin-xin Pang, Yue-tong Huang, et al., Molecular Transformation and Characterizations of Super-deamidated Wheat Gluten by a Tunable Hydrated Ternary Natural Deep Eutectic Solvent, ACS Sustainable Chem. Eng. (2023) https://pubs.acs.org/doi/abs/10.1021/acssuschemeng.3c00655 (此文没有按要求引用Molclus!)
- Ziyi Wang, Weigen Chen, Tianyi Sang, et al., Fullerenol-based Toxic Fluoride Gas Sensing: A Promising Way to Monitoring Li-ion Battery Status, Surf. Interf. (2023) https://www.sciencedirect.com/science/article/abs/pii/S2468023023002638
- Zhichao Gong, Xuejiao Rong, Guomin Sui, et al., Intriguing Optical Properties due to the Intermolecular Interaction between AIE and ACQ Materials, Adv. Opt. Mater. (2023) https://onlinelibrary.wiley.com/doi/abs/10.1002/adom.202203075
- Pingping Shao, Ruoya Wang, Tianxiang Zhao, Chun Zhu, Protic amino-2-propanol hydrochloride-based deep eutectic solvents with multiple hydrogen bond sites for efficient capture of NH3, Int. J. Hyd. Energy (2023) https://www.sciencedirect.com/science/article/abs/pii/S0360319923018475 (此文没有按要求引用Molclus!)
- Yuting Li, Cheng Cao, Meng Cai, et al., Green Hydrophobic Deep Eutectic Solvents as Low-Viscosity and Efficient Lubricants, Tribol. Int. (2023) https://www.sciencedirect.com/science/article/abs/pii/S0301679X23003183 (此文没有按要求引用Molclus!)
- Shilong Dong, Xiaoyan Sun, Lili Wang, et al., Prediction, Application, and Mechanism Exploration of Liquid–Liquid Equilibrium Data in the Extraction of Aromatics Using Sulfolane, Processes, 11, 1228 (2023) https://doi.org/10.3390/pr11041228
- Yongpan Tian, Fan Zhang, Juan Xie, et al., Electrophilic substitution reactivity prediction of crown ether derivatives, J. Phys. Org. Chem. (2023) https://onlinelibrary.wiley.com/doi/abs/10.1002/poc.4509
- Shengyu Yu, Jiahao Liu, Zhe Wang, et al., Design of Y-Shaped Trimers of Chiral Phase-Transfer Catalysts for the Asymmetric Alkylation of Amino Acid Derivatives, Synlett (2023) https://www.thieme-connect.com/products/ejournals/abstract/10.1055/a-2065-3962
- Yimeng Sun, Yuqian Jiang, Jian Jiang, et al., Keto-form directed hierarchical chiral self-assembly of Schiff base derivatives with amplified circularly polarized luminescence, Chin. Chem. Lett. (2023) https://www.sciencedirect.com/science/article/abs/pii/S1001841723002632
- Dan Li, Cuiping Li, Efficient and Reversible Capture of CO2 in CO2-Binding Organic Liquids Formed by 1,1,3,3-Tetramethylguanidine and Glycerol Derivatives, ACS Sustainable Chem. Eng., 11, 5335 (2023) https://pubs.acs.org/doi/abs/10.1021/acssuschemeng.2c05109
- Biao Zhao, Huanyu Ma, Haoran Jia, et al., Triphenylamine-Derived Solid-State Emissive Carbon Dots for Multicolor High-Efficiency Electroluminescent Light-Emitting Diodes, Angew. Chem. (2023) https://onlinelibrary.wiley.com/doi/abs/10.1002/anie.202301651
- Zongwei Hao, Huajian Xu, Yiyang Yu, et al., Preparation of the starch-lipid complexes by ultrasound treatment: Exploring the interactions using molecular docking, Int. J. Biol. Macromol., 237, 124187 (2023) https://www.sciencedirect.com/science/article/abs/pii/S0141813023010814 (此文没有按要求引用Molclus!)
- Long Han, Wen Zheng, Sheng-Yan Qian, et al., New Guaiane-Type Sesquiterpenoids Biscogniauxiaols A–G with Anti-Fungal and Anti-Inflammatory Activities from the Endophytic Fungus Biscogniauxia Petrensis, J. Fungi, 9, 393 (2023) https://doi.org/10.3390/jof9040393
- Benni Du, Weichao Zhang, Kinetics, Products, and Mechanisms Study of the Atmospheric Degradation of (E)-4-Methoxy-3-buten-2-one with Hydroxyl Radicals, ACS Earth Space Chem. (2023) https://pubs.acs.org/doi/abs/10.1021/acsearthspacechem.2c00388
- Zhaoyan Zhang, Chong Wang, Yingqi Zhao, et al., Autoxidation Mechanism and Kinetics of Methacrolein in the Atmosphere, J. Phys. Chem. A (2023) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.3c00128
- Jie Han, Siyuan Liu, Huanan Wang, et al., Pd/Xu-Phos-catalyzed asymmetric elimination of fully substituted enol triflates into axially chiral trisubstituted allenes, Sci. Adv. (2023) https://www.science.org/doi/full/10.1126/sciadv.adg1002
- Zhen-Chao Long, Zhiyou Wei, Kai-Wen Liu, et al., Structures and Bonding Properties of Lithium Polysulfide Clusters LiSn−/0(n = 3-5) and Li2S4−/0: Size-Selected Anion Photoelectron Spectroscopy and Theoretical Calculations, Phys. Chem. Chem. Phys. (2023) https://pubs.rsc.org/en/content/articlelanding/2023/cp/d2cp06061b/
- Su Wang, Qifang Sun, Qing Zhang, et al., Li-Ion Transfer Mechanism of Ambient-Temperature Solid Polymer Electrolyte toward Lithium Metal Battery, Adv. Energy Mater. (2023) https://onlinelibrary.wiley.com/doi/abs/10.1002/aenm.202204036 (此文没有按要求引用Molclus!)
- Chao Li, Yun Teng, Huanong Cheng, et al., Density, viscosity, and H2S solubility of N-butylmorpholine bromide iron-based ionic liquids, J. Mol. Liq. (2023) https://www.sciencedirect.com/science/article/abs/pii/S0167732223003951 (此文没有按要求引用Molclus!)
- Zicheng Cai, Chun Zhu, Guoqing Chen, et al., Study on intermolecular hydrogen bond of uric acid water-clusters, Chem. Phys. Lett. (2023) https://www.sciencedirect.com/science/article/abs/pii/S000926142300129X
- Bumaliya Abulimiti, Huan An, Zhenfei Gu, et al., Femtosecond Time-Resolved Observation of Relaxation and Wave Packet Dynamics of the S1 State in Electronically Excited o-Fluoroaniline, Molecules, 28, 1999 (2023) https://doi.org/10.3390/molecules28041999
- Wei-Han Rao, Lin Yu, Jian-Dong Ding, Stride Strategy to Enable a Quasi-ergodic Search of Reaction Pathways Demonstrated by Ring-opening Polymerization of Cyclic Esters, Chin. J. Polym. Sci. (2023) https://link.springer.com/article/10.1007/s10118-023-2930-6
- Hongjuan Zhu, Danyang Zhang, Eryin Feng, Xiaowei Sheng, Effects of aggregation on the structures and excited-state absorption for zinc phthalocyanine, Phys. Chem. Chem. Phys. (2023) https://pubs.rsc.org/en/content/articlelanding/2023/cp/d2cp04372f/
- Qinpei Lu, Gang Wu, Xiaomeng Hao, et al., Isolation, Structure Elucidation, and First Total Synthesis of Quinomycins K and L, Two New Octadepsipeptides from the Maowei Sea Mangrove-Derived Streptomyces sp. B475, Mar. Drugs, 21, 143 (2023) https://www.mdpi.com/1660-3397/21/3/143/
- ZHUANG LYUCHAO, DEVELOPMENT OF METAL HALIDE PEROVSKITE FILMS FOR LIGHT EMITTING DIODE APPLICATIONS, PhD Dissertation of The Hong Kong Polytechnic University (2022) https://theses.lib.polyu.edu.hk/bitstream/200/12182/3/6630.pdf (此文没有按要求引用Molclus!)
- Jiajing Bao, Xingchi Zhang, Hongfei Bie, et al., A quantum chemical computation and model investigation for autoignition kinetic of a long chain oxygenate: Tri-propylene glycol methyl ether, Fuel, 343, 127933 (2023) https://www.sciencedirect.com/science/article/abs/pii/S001623612300546X (此文没有按要求引用Molclus!)
- Ling Pei, Weak Interactions between Epinephrine and Thymine, Lett. Org. Chem., 20, 230 (2023) https://www.ingentaconnect.com/content/ben/loc/2023/00000020/00000003/art00005
- Xipeng Yang, Yongjie Dong, Song Ma, et al., Humidity-resistant organic room-temperature phosphorescence materials synthesized using catalyst-free click reaction, Chem. Eng. J., 462, 142198 (2023) https://www.sciencedirect.com/science/article/abs/pii/S1385894723009294
- Huanfei Xu, Chenyang Dong, Weixian Wang, et al., Machine learning prediction of deep eutectic solvents pretreatment of lignocellulosic biomass, Ind. Crops Prod., 196 (2023) https://www.sciencedirect.com/science/article/abs/pii/S0926669023001954 (此文没有按要求引用Molclus!)
- Hong-Ying Yang, Weidong Yao, Pei-Zhi Huang, et al., Euphohelides A–C, ent-Abietane-Type Norditerpene Lactones from Euphorbia helioscopia and Their Anti-Inflammatory Activities, J. Nat. Prod. (2023) https://pubs.acs.org/doi/abs/10.1021/acs.jnatprod.3c00041 (此文没有按要求引用Molclus!)
- Xu Yang, Bao Zhang, Yao Tian, et al., Electrolyte design principles for developing quasi-solid-state rechargeable halide-ion batteries, Nat. Commun., 14, 925 (2023) https://www.nature.com/articles/s41467-023-36622-w
- Huajian Xu, Zongwei Hao, Junwei Gao, et al., Complexation between rice starch and cellulose nanocrystal from black tea residues: Gelatinization properties and digestibility in vitro, Int. J. Biol. Macromol., 234, 123695 (2023) https://www.sciencedirect.com/science/article/abs/pii/S0141813023005883 (此文没有按要求引用Molclus!)
- Yifan Zhang, Xianwei Hu, Fengguo Liu, et al., Experimental and quantum chemical investigations on the generation mechanism of Al-V intermediate alloy by aluminothermic reduction of NaVO3, J. Alloys Compd. (2023) https://www.sciencedirect.com/science/article/abs/pii/S0925838823005558
- Xin Liu, Mengke Bu, Changgong Meng, Superior Thermostability of Poly-Silicic Acid Analogues of Zeolite Composite/Secondary Building Units: A Theoretical Investigation, J. Phys. Chem. C (2023) https://pubs.acs.org/doi/abs/10.1021/acs.jpcc.2c08147
- Jia Gao, Li Chen, Wendong Xing, et al., “Nanomagnet-inspired” design on molecularly imprinted nanofiber membrane: Mechanisms for improved transport selectivity of sufficient specific sites, J. Membrane Sci. (2023) https://www.sciencedirect.com/science/article/abs/pii/S0376738823001230
- Aikebaier Jumai, Guo-An Zou, Ge-yu Liu, Haji Akber Aisa, Pyrrolizidine alkaloids from Jacobaea vulgaris Gaertn and theoretical studies on intramolecular interactions, Nat. Prod. Res. (2023) https://www.tandfonline.com/doi/abs/10.1080/14786419.2023.2174533
- Xuan Zheng, Nairong Yang, Yanjun Hou, Kaicong Cai, Dissecting amide-I vibrations in histidine dipeptide, Spectrochim. Acta A (2023) https://www.sciencedirect.com/science/article/pii/S1386142523001099
- Shiyao Niu, Xiaoqing Wu, Qifeng Hou, et al., Theoretical Kinetic Studies on Thermal Decomposition of Glycerol Trinitrate and Trimethylolethane Trinitrate in the Gas and Liquid Phases, J. Phys. Chem. A (2023) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.2c07282
- Shuai-Ting Yan, Zhen-Chao Long, Xi-Ling Xu, et al., Anion photoelectron spectroscopy and quantum chemical calculations of bimetallic niobium-aluminum clusters NbAln‾/0 (n = 3-12): Identification of half-encapsulated symmetric structure for NbAl12‾, Phys. Chem. Chem. Phys. (2023) https://pubs.rsc.org/en/content/articlehtml/2023/cp/d2cp04978c
- Huan Niu, Guanghao Qu, Mingru Li, et al., Improved surface flashover voltage of epoxy following polythiourea-assisted coating with high gas adsorption ability, Appl. Surf. Sci. (2023) https://www.sciencedirect.com/science/article/abs/pii/S0169433223002222
- Haonan Wu, Lu Shen, Li Tian, et al., Excitation of quantum-sized (CuS)n clusters for NIR-II photothermal application: Insights from experiment and DFT calculation, J. Alloys Comp., 941, 169005 (2023) https://www.sciencedirect.com/science/article/abs/pii/S0925838823003080
- Honggang Shi, Zeping Lu, Huajin Xu, et al., Selective and Efficient Synthesis of Pine Sterol Esters Catalyzed by Deep Eutectic Solvent, Molecules, 28, 993 (2023) https://doi.org/10.3390/molecules28030993
- Wen Yu, Yiyang Bo, Yiling Luo, et al., Enhancing effect of choline chloride-based deep eutectic solvents with polyols on the aqueous solubility of curcumin–insight from experiment and theoretical calculation, Chin. J. Chem. Eng. (2022) https://www.sciencedirect.com/science/article/abs/pii/S1004954123000150
- Yuling Zhang, Hongwei Ren, Hasmerya Maarof, et al., The Effect of Water Content on Lignin Solubilization in Deep Eutectic Solvents, J. Mol. Liq. (2023) https://www.sciencedirect.com/science/article/abs/pii/S0167732223000740
- Wei Liu, Xinli Wang, Renfu Li, et al., The location of benzothiazole on the skeleton of rofecoxib defines an AIEgen and its potential application as multifunctional materials, J. Mol. Struct. (2023) https://www.sciencedirect.com/science/article/abs/pii/S0022286023000844
- Shi Quan Wu, Li Li, Qi Qi Liang, et al., Density Functional Study of transition metal (Fe,Co,Ni) doped C60 fullerenes as 6-thioguanine delivery system, Appl. Organomet. Chem. (2022) https://onlinelibrary.wiley.com/doi/abs/10.1002/aoc.7018
- Xiao-Wei Guo, Zhen-Qing Yu, Junmin Xi, et al., Isolation and Identification of Novel Antioxidant Polyketides from an Endophytic Fungus Ophiobolus cirsii LZU-1509, J. Agric. Food Chem. (2023) https://pubs.acs.org/doi/abs/10.1021/acs.jafc.2c07386
- Ling Zheng, Jinfeng Zhong, Xiong Liu, et al., Physicochemical properties and intermolecular interactions of a novel diacylglycerol oil oleogel made with ethyl cellulose as affected by γ-oryzanol, Food Hydrocolloids (2023) https://www.sciencedirect.com/science/article/abs/pii/S0268005X23000309
- Yixin Ma, Yajuan Qu, Yaojin ji, et al., Structure-activity relationship and experimental study of organic solvents and deep eutectic solvents in separation of cyclohexane-ethyl acetate, J. Mol. Liq. (2023) https://www.sciencedirect.com/science/article/abs/pii/S0167732223000661 (此文没有按要求引用Molclus!)
- Imma Carbo-Bague, Cen Li, Brooke L. McNeil, et al., Comparative Study of a Decadentate Acyclic Chelate, HOPO-O10, and Its Octadentate Analogue, HOPO-O8, for Radiopharmaceutical Applications, Inorg. Chem. (2023) https://pubs.acs.org/doi/abs/10.1021/acs.inorgchem.2c03671
- Yaping Tao, Hengwei Zhang, Yi Wang, Revealing and predicting the relationship between the molecular structure and antioxidant activity of flavonoids, LWT (2023) https://www.sciencedirect.com/science/article/pii/S0023643823000117
- Shengya Zhang, Wenhui Fang, Bo Zhao, et al., Investigating the H-Bond network of Ternary (Xylitol-Water-Acetic acid) Aqueous Solutions by Raman Spectroscopy and DFT, J. Mol. Liq. (2022) https://www.sciencedirect.com/science/article/abs/pii/S0167732222026988
- Yong Liu, Long Wang, Chang Liu, et al., Enhanced cadmium removal by biochar and iron oxides composite: Material interactions and pore structure, J. Environ. Manag., 330, 117136 (2023) https://www.sciencedirect.com/science/article/pii/S0301479722027098
- Bo Li, Lei Lou, Peng Jin, Locating the hydrogen atoms in endohedral clusterfullerenes by density functional theory, Phys. Chem. Chem. Phys. (2022) https://pubs.rsc.org/en/content/articlelanding/2022/cp/d2cp05050a/
- Siyuan Chen, Haiming Cai, Xiaodong Du, et al., Adsorption behavior of hierarchical porous biochar from shrimp shell for tris(2-chloroethyl) phosphate (TCEP): Sorption experiments and DFT calculations, Environ. Res., 219, 115128 (2023) https://www.sciencedirect.com/science/article/abs/pii/S0013935122024550
- Meijie Sun, Suqian Gu, Xiangyang Liu, et al., Adsorption mechanism of ammonia nitrogen and phenol on lignite surface: Molecular dynamics simulations and quantum chemical calculations, Fuel, 337, 127157 (2023) https://www.sciencedirect.com/science/article/abs/pii/S0016236122039813 (此文没有按要求引用Molclus!)
- Jiafu Xing, Yajuan Qu, Zihao Su, et al., Phase Equilibria and Mechanism Insights into the Separation of Isopropyl Acetate and Methanol by Deep Eutectic Solvents, Ind. Eng. Chem. Res. (2022) https://pubs.acs.org/doi/abs/10.1021/acs.iecr.2c03488
- Yanrong Jiang, Zhaojie Cai, Qinqin Yuan, et al., Highly Structured Water Networks in Microhydrated Dodecaborate Clusters, J. Phys. Chem. Lett., 13, 11787 (2022) https://pubs.acs.org/doi/abs/10.1021/acs.jpclett.2c03537
- Yong-Qiang Xie, Ming-Ming Han, You-Ming Zhang, et al., A novel fluorescent probe with high sensitivity for sequential detection of CN− and Al3+ in highly aqueous medium and its applications in living cell bioimaging, J. Photochem. Photobiol. A (2022) https://www.sciencedirect.com/science/article/abs/pii/S1010603022007110
- Qian Yao, Yansong Zhu, Cheng Zheng, et al., Intermolecular Cross-Linking Reinforces Polymer Binders for Durable Alloy-Type Anode Materials of Sodium-Ion Batteries, Adv. Energy Mater. (2022) https://onlinelibrary.wiley.com/doi/10.1002/aenm.202202939
- Yumiao Huo, Jichen Lv, Yanchao Xie, et al., Structurally Regulated Carbazole–Pyridine Derivatives Based on Space-Crowded Theory for Efficient Narrowband Ultraviolet Nondoped Organic Light-Emitting Diodes from the High-Lying Reverse Intersystem Crossing Process, ACS Appl. Mater. Interfaces (2022) https://pubs.acs.org/doi/abs/10.1021/acsami.2c20806
- Hongxin Qiu, Xiaohao Sun, Bozeng Wu, et al., Evaluation of the difference in adsorption of sodium alginate as an efficient and non-toxic arsenopyrite inhibitor on the surface of arsenopyrite and chalcopyrite, Appl. Surf. Sci. (2022) https://www.sciencedirect.com/science/article/abs/pii/S0169433222035449
- Qi Liang Lu, Xiao Dong Liu, Qi Quan Luo, Chen Ri Wang, Quasi-planar Co atom-doped boron cluster: CoB192−, J. Mol. Model., 29, 7 (2023) https://link.springer.com/article/10.1007/s00894-022-05404-4
- Heyao Yuan, Peng Zhang, Hongbin Zhan, et al., Theoretical investigation of turn off–on mechanism of a new fluorescence probe L, Chem. Phys. Lett. (2022) https://www.sciencedirect.com/science/article/abs/pii/S0009261422009137
- Wenjin Liu, Huan Tang, Beichen Yang, et al., Molecular level insight into the different interaction intensity between microplastics and aromatic hydrocarbon in pure water and seawater, Sci. Total Environ., 862, 160786 (2023) https://www.sciencedirect.com/science/article/abs/pii/S0048969722078895
- Study on spectral properties of butyl hydroxytoluene: experiment and theoretical calculation, Inorg. Chem. Commun. (2022) https://www.sciencedirect.com/science/article/abs/pii/S1387700322010917
- Lipeng Su, Jiankun Zhuo, Hao Liu, et al., Fragmentation modeling of gas-phase ionic liquid clusters in high-voltage electric field, Fuel, 335, 126919 (2023) https://www.sciencedirect.com/science/article/abs/pii/S0016236122037437
- Surajit Metya, Aloke Das, S–H···O Hydrogen Bond Can Win over O–H···S Hydrogen Bond: Gas-Phase Spectroscopy of 2-Fluorothiophenol···H2O Complex, J. Phys. Chem. A (2022) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.2c06083
- Weidong Qiu, Denghui Liu, Mengke Li, et al., Confining donor conformation distributions for efficient thermally activated delayed fluorescence with fast spin-flipping, ResearchSquare (2022) https://www.researchsquare.com/article/rs-2314933/v1
- Xiaoyu Li, Jun Xiao, Wanqing Wei, et al., Preparation mechanism and performance evaluation of deep eutectic solvent-lignin/ZnO composites by one-pot, Int. J. Biol. Macromol. (2022) https://www.sciencedirect.com/science/article/abs/pii/S014181302202757X (此文没有按要求引用Molclus!)
- Tian Liu, Lingxiao Li, Shuo Wang, et al., Hybrid chalcogen bonds in prodrug nanoassemblies provides dual redox-responsivity in the tumor microenvironment, Nat. Commun., 13, 7228 (2022) https://www.nature.com/articles/s41467-022-35033-7
- Shuping He, Ying Lv, Xiaohang Wu, et al., Mechanistic Insights into Oxidation-Induced Size Conversion of [Au6(dppp)4]2+ to [Au8(dppp)4Cl2]2+, Inorg. Chem. (2022) https://pubs.acs.org/doi/abs/10.1021/acs.inorgchem.2c02885
- Jiguang Li, Da Lei, Zhiwei Ma, et al., A General Twisted Intramolecular Charge Transfer Triggering Strategy by Protonation for Zero-Background Fluorescent Turn-On Sensing, J. Phys. Chem. Lett., 13, 10871 (2022) https://pubs.acs.org/doi/abs/10.1021/acs.jpclett.2c02847
- Lin Chen, Li Ge, Qinghui Liang, et al., Eutectic solvents formed by (+)-DTTA and L-menthol as novel chiral recognition and separation media for enantioselective extraction of valsartan enantiomers, J. Mol. Liq., 368, 120818 (2022) https://www.sciencedirect.com/science/article/abs/pii/S0167732222023571
- Jiabin Huang, Can Huang, Qifeng Hou, et al., A theoretical study on dissociative photoionization and photoionization cross-sections of a typical ketohydroperoxide in n-butane low-temperature oxidation, Combust. Flame (2022) https://www.sciencedirect.com/science/article/abs/pii/S0010218022004990
- Ding Wei, Qinlan Luo, Tong Ouyang, et al., An experimental and theoretical study on the effects of amine chain length on CO2 absorption performance, AIChE J. (2022) https://aiche.onlinelibrary.wiley.com/doi/abs/10.1002/aic.17960
- Tong Deng, Yifu Chen, Yanbo Liu, et al., Constructing Janus Microsphere Membranes for Particulate Matter Filtration, Directional Water Vapor Transfer, and High-Efficiency Broad-Spectrum Sterilization, Small (2022) https://onlinelibrary.wiley.com/doi/abs/10.1002/smll.202205010 (此文没有按要求引用Molclus!)
- Zhengshan Luo, Bowen Cao, Tianhang Song, et al., Visible-Light Organophotoredox-Mediated [3 + 2] Cycloaddition of Arylcyclopropylamine with Structurally Diverse Olefins for the Construction of Cyclopentylamines and Spiro[4.n] Skeletons, J. Org. Chem. (2022) https://pubs.acs.org/doi/abs/10.1021/acs.joc.2c02061
- Xiaodi Dong, Baoquan Wan, Yang Feng, et al., Ultra-low-permittivity, high hydrophobic, and excellent thermally stable fluoroelastomer/polyimide composite films employing dielectric reduction, Eur. Polym. J., 181, 111667 (2022) https://www.sciencedirect.com/science/article/abs/pii/S0014305722006711
- Jianguang Qi, Yajuan Qu, Mengjin Zhou, et al., Phase behavior and molecular insights on the separation of dimethyl carbonate and methanol azeotrope by extractive distillation using deep eutectic solvents, Sep. Purif. Technol., 305, 122489 (2023) https://www.sciencedirect.com/science/article/abs/pii/S1383586622020457
- Meichen Li, Yunqiang Zhang, Tingting Han, et al., Verniciflavanol A, a profisetinidin-type-4-arylflavan-3-ol from toxicodendron vernicifluum protects SH-SY5Y cells against H2O2-Induced oxidative stress, Phytochem., 205, 113487 (2023) https://www.sciencedirect.com/science/article/abs/pii/S0031942222004034
- Zongwei Hao, Shengjun Han, Huajian Xu, et al., Insights into the rheological properties, multi-scale structure and in vitro digestibility changes of starch-β-glucan complex prepared by ball milling, Int. J. Biol. Macromol. (2022) https://www.sciencedirect.com/science/article/abs/pii/S014181302202459X (此文没有按要求引用Molclus!)
- Aamir Hassan Shah, Zisheng Zhang, Zhihong Huang, et al., The role of alkali metal cations and platinum-surface hydroxyl in the alkaline hydrogen evolution reaction, Nat. Catal., 5, 923 (2022) https://www.nature.com/articles/s41929-022-00851-x
- Zhengrun Chen, Yanli Zhang, Mengjin Zhou, et al., Mechanism analysis and process optimization of acetone–methanol azeotrope separation using 1-ethyl-3-methylimidazolium acetate based mixed extractants, J. Clean. Prod. (2022) https://www.sciencedirect.com/science/article/abs/pii/S0959652622042597
- Cheng Xie, Qiang Xu, Weiye Chen, et al., Evaluating the role of hydroxyl keto-hydroperoxide in the low temperature oxidation of alkenes, Combust. Flame, 246, 112414 (2022) https://www.sciencedirect.com/science/article/abs/pii/S001021802200431X
- Yuhao Liu, Ying Lin, Bin Cao, et al., Enhancement of polysiloxane/epoxy resin compatibility through an electrostatic and van der Waals potential design strategy, Polym. Test., 117, 107820 (2023) https://www.sciencedirect.com/science/article/pii/S0142941822003415
- Zengjiang Zhang, Bo Zheng, Anlin Du, Insight into the retardation of retrogradation of chestnut starch by heat-moisture treatment with flavonoids, Food Chem. (2022) https://www.sciencedirect.com/science/article/abs/pii/S0308814622025493
- Hui Maa, Bin-Bin Yanga, Zhen Wang, et al., A three dimensional graphdiyne-like porous triptycene network for gas adsorption and separation, RSC Adv., 12, 28299 (2022) https://pubs.rsc.org/en/content/articlehtml/2022/ra/d2ra04031j
- Xu Zhao, Ning Zhang, Guang-Feng Wu, et al., Intermolecular copigmentation between anthocyanidin-3,5-O-diglucosides and three phenolic compounds: Insights from experimental and theoretical studies, Food Chem. Adv. (2022) https://www.sciencedirect.com/science/article/pii/S2772753X22000995
- Jingsi Qiu, Yue Liu, Quantum chemistry calculation and experimental research on the component proportion of black disperse dye, Textile Research Journal (2022) https://journals.sagepub.com/doi/abs/10.1177/00405175221125255
- Wenjin Cao, Zhubin Hu, Xiaogai Peng, et al., Annihilating Actinic Photochemistry of the Pyruvate Anion by One and Two Water Molecules, J. Am. Chem. Soc. (2022) https://pubs.acs.org/doi/abs/10.1021/jacs.2c06319
- Wei Lei, Yiming Luo, Bingbo Li, et al., Theoretical study on the interaction between 3,4-dinitropyrazole and cyclotetramethylene tetranitramine, Monatshefte für Chemie - Chemical Monthly (2022) https://link.springer.com/article/10.1007/s00706-022-02979-9 (此文没有按要求引用Molclus!)
- Binbin Nian, Yi Hu, Anionic H-bonds improve the disorganization of starch in metal chloride hydrate-natural deep eutectic solvents, Carbohyd. Polym., 298, 120150 (2022) https://www.sciencedirect.com/science/article/abs/pii/S0144861722010554
- Qian-Jin Guo, Shu-Hai Zhang, Rui-Jun Gou, et al., A theoretical investigation on the interaction mechanism between 1-methyl-3,4,5-trinitropyrazole and 1,3,5,7-Tetranitro-1,3,5,7-tetrazocane, Chem. Paper (2022) https://link.springer.com/article/10.1007/s11696-022-02500-w
- Shuai Tang, Xianwei Hu, Jiangyu Yu, et al., Dissolution reaction of TiO2 in molten 6.58NaF-AlF3:A Raman spectroscopy and computational simulation study, J. Mol. Liq. (2022) https://www.sciencedirect.com/science/article/abs/pii/S0167732222019705
- Xiaofan Li, Ning Wang, Guochun Lv, et al., Experimental and theoretical insight into the transformation behaviors and risk assessment of Flutamide in UV/O3/PMS system, J. Cleaner Prod., 375, 134167 (2022) https://www.sciencedirect.com/science/article/abs/pii/S0959652622037398
- He Bian, Fang Xu, Shengchao Wei, et al., A conversion strategy to disaggregate asphaltenes via mild hydrotreatment: Theoretical and experimental investigation, Chem. Eng. J. (2022) https://www.sciencedirect.com/science/article/pii/S000925092200690X
- Haihui Wang, Xiaohui Zhu, Han Xiao, et al., Self-assembled Immunostimulatory Nanosphere Combined with PD-L1 to Enhanced Cancer Chemoimmunotherapy, Mater. Design (2022) https://www.sciencedirect.com/science/article/pii/S026412752200781X (此文没有按要求引用Molclus!)
- Chun-Lai Ren, Yue Shan, Pengfei Zhang, et al., Uncovering the molecular mechanism for dual effect of ATP on phase separation in FUS solution, Sci. Adv. (2022) https://www.science.org/doi/full/10.1126/sciadv.abo7885
- Tinglei Chen, Guo Ye, Haowei Wu, et al., Highly Conductive and Underwater Stable Ionic Skin for All-Day Epidermal Biopotential Monitoring, Adv. Funct. Mater. (2022) https://onlinelibrary.wiley.com/doi/abs/10.1002/adfm.202206424
- Hui Wang, Guangzhao Zhang, Yukun Chen, et al., Highly Conductive and Underwater Stable Ionic Skin for All-Day Epidermal Biopotential Monitoring, Chem. Eng. J. (2022) https://www.sciencedirect.com/science/article/abs/pii/S1385894722046071
- Ge Zhu, Haoji Lao, Feng Feng, et al., Synthesis and characterization of poly(amide-imide)s with high Tg and low CTE derived from isomeric amide-containing diamines, Eur. Polym. J. (2022) https://www.sciencedirect.com/science/article/abs/pii/S0014305722005626
- Shuo Wang, Yanbo Liu, Shilin Guo, et al., Design of Single and Multicomponent Drug Spherical Particles with Improved Powder Properties in Water Instead of Organic Solvents: A Case Study of Gemfibrozil, ACS Sustainable Chem. Eng. (2022) https://pubs.acs.org/doi/abs/10.1021/acssuschemeng.2c03982
- Chun-Xiao Jiang, Bo Yu, Ya-Mei Miao, et al., Correction to Indole Alkaloids from a Soil-Derived Clonostachys rosea, J. Nat. Prod. (2022) https://pubs.acs.org/doi/full/10.1021/acs.jnatprod.2c00778 (此文没有按要求引用Molclus!)
- Yao Ma, Haoliang Li, Chunsheng Xie, et al., Treatment of PBDEs from Soil-Washing Effluent by Granular-Activated Carbon: Adsorption Behavior, Influencing Factors and Density Functional Theory Calculation, Processes, 10, 1815 (2022) https://www.mdpi.com/2227-9717/10/9/1815
- Qi Li, Songtao Liu, Nyv Mondele Mbola, Responsive hydrogel-based three-dimensional photonic crystal sensor for lactic acid detection, Anal. Bioanal. Chem. (2022) https://link.springer.com/article/10.1007/s00216-022-04300-3
- Yao Ma, Jinfan Chen, Xiaodong Du, et al., Efficient removal of polybrominated diphenyl ethers from soil washing effluent by dummy molecular imprinted adsorbents: Selectivity and mechanisms, J. Environ. Sci. (2022) https://www.sciencedirect.com/science/article/abs/pii/S1001074222004284
- Shuai-Ting Yan, Xi-Ling Xu, Hong-Guang Xu, Wei-Jun Zheng, Anion Photoelectron Spectroscopy and Quantum Chemical Calculations of Bimetallic Oxide Clusters YCu2On–/0 (n = 2–5), J. Phys. Chem. A (2022) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.2c03968
- Yan Zhang, Cidan Lv, Changwei Hu, Zhishan Su, Mechanistic Study of Asymmetric Alkynylation of Isatin-Derived Ketimine Mediated by a Copper/Guanidine Catalyst, J. Org. Chem. (2022) https://pubs.acs.org/doi/abs/10.1021/acs.joc.2c01321
- Ablajan Turak, Haji Akber Aisa, Oxygen heterocyclic Diels-Alder-Type sesquiterpenoid dimers from Vernonia anthelmintica, Phytochem. (2022) https://www.sciencedirect.com/science/article/abs/pii/S0031942222003028
- Hai-Wei Yan, Ya-Nan Yang, Xu Zhang, et al., Eight New Arnebinol B-Based Meroterpenoids with Planar Chirality in the Constrained 6/10/5 Tricyclic Skeleton from Arnebia euchroma and Their Cytotoxicities, Bioorg. Chem. (2022) https://www.sciencedirect.com/science/article/abs/pii/S0045206822004977 (此文没有按要求引用Molclus!)
- Yan Wen, Li Rao, Fan Xu, et al., Six pairs of phenylpropanoid enantiomers from Cinnamomum mollifolium, Phytochem. (2022) https://www.sciencedirect.com/science/article/abs/pii/S0031942222002643
- Long Cheng, Yuanhui Ji, Photocatalytic activation of sulfite by N-doped porous biochar/MnFe2O4 interface-driven catalyst for efficient degradation of tetracycline, Green Energy Environ. (2022) https://www.sciencedirect.com/science/article/pii/S2468025722001224
- Xiaorui Lv, Jianlou Mu, Wenxiu Wang, et al., Effects and mechanism of natural phenolic acids/fatty acids on copigmentation of purple sweet potato anthocyanins, Curr. Res. Food Sci., 5, 1243 (2022) https://www.sciencedirect.com/science/article/pii/S2665927122001204
- Xin Ai, Fei Pan, Zichen Yang, et al., Computational design of a chitosan derivative for improving the color stability of anthocyanins: Theoretical calculation and experimental verification, Int. J. Biol. Macromol., 219, 721 (2022) https://www.sciencedirect.com/science/article/abs/pii/S0141813022017457
- Hongxin Qiu, Bozeng Wu, Jiushuai Deng, et al., The effect of collectors on froth stability of frother: Atomic-scale study by experiments and molecular dynamics simulations, J. Mol. Liq., 364, 120035 (2022) https://www.sciencedirect.com/science/article/abs/pii/S0167732222015732
- Suocheng Chi, Yingzhe Yu, Minhua Zhang, An investigation on chain transfer to monomers and initiators, termination of radical chains and primary radicals in EVA copolymerization process based on DFT calculation and microkinetic simulation, Polymer (2022) https://www.sciencedirect.com/science/article/abs/pii/S0032386122006693
- Xitong Zhao, Jizhen Li, Shuxin Quan, et al., Study on the effect of solvent on cocrystallization of CL-20 and HMX through theoretical calculations and experiments, RSC Adv., 12, 21255 (2022) https://pubs.rsc.org/en/content/articlehtml/2022/ra/d2ra03730k
- Xiaoyang Liu, C. Turner, Electronic structure calculations of the fundamental interactions in solvent extraction desalination, J. Mol. Liq., 364, 119986 (2022) https://www.sciencedirect.com/science/article/abs/pii/S0167732222015240
- Quan-Chang Gu, Xiao-Li Wei, Qing Ji, et al., Two dimers generated by lithospermic decarboxylation coupling from Danshen, Bioorg. Chem., 128, 106065 (2022) https://www.sciencedirect.com/science/article/abs/pii/S0045206822004710
- Yong Liu, Jie Ma, Jiaoli Gao, et al., Stability and interaction of biochar and iron mineral nanoparticles: effect of pH, ionic strength, and dissolved organic matter, Biochar, 4, 47 (2022) https://link.springer.com/article/10.1007/s42773-022-00172-z
- Jianglian She, Yi Chen, Yuxiu Ye, et al., New Carboxamides and a New Polyketide from the Sponge-Derived Fungus Arthrinium sp. SCSIO 41421, Mar. Drugs, 20, 475 (2022) https://www.mdpi.com/1660-3397/20/8/475
- Xinyu Wang, Keyang Zheng, Zhixiao Peng, et al., Exploiting Proton Masking to Protect Amino Achieve Efficient Capture CO2 by Amino-acids Deep eutectic solvents, Sep. Purif. Technol. (2022) https://www.sciencedirect.com/science/article/abs/pii/S1383586622013430
- Mohsen Doust Mohammadi, Faheem Abbas, Hitler Louis, et al., Trapping of CO, CO2, H2S, NH3, NO, NO2, and SO2 by Polyoxometalate compound, Comput. Theor. Chem. (2022) https://www.sciencedirect.com/science/article/abs/pii/S2210271X22002390
- Mingli Mu, Xinfeng Zhang, Gangqiang Yu, et al., Effective absorption of dichloromethane using deep eutectic solvents, J. Hazard. Mater. (2022) https://www.sciencedirect.com/science/article/abs/pii/S0304389422014595
- Shenghao Wang, Qing Huang, A spectroscopic approach to identifying the out-of-plane conformations of Ni(II) meso-tetraphenylporphyrin in solution, J. Mol. Struct. (2022) https://www.sciencedirect.com/science/article/abs/pii/S0022286022014326
- Minhua Zhang, Suocheng Chi, Yingzhe Yu, DFT Investigation of Polyethylene-co-vinyl Acetate: Kinetics of Initiation and Propagation, Copolymer Composition, and Unit Sequence Distribution, Ind. Eng. Chem. Res. (2022) https://pubs.acs.org/doi/abs/10.1021/acs.iecr.2c01401
- Xiaorui Lv, Liling Li, Xiaomin Lu, et al., Effects of organic acids on color intensification, thermodynamics, and copigmentation interactions with anthocyanins, Food Chem., 396, 133691 (2022) https://www.sciencedirect.com/science/article/abs/pii/S0308814622016533 (此文没有按要求引用Molclus!)
- Liping Jiang, Xiaolong Fu, Jun Jiang, et al., Catalytic Effect of Ferrocenyl Energetic Catalysts for N-Guanylurea Dinitramide (GUDN or FOX-12) Decomposition, ACS Omega (2022) https://pubs.acs.org/doi/full/10.1021/acsomega.2c03026
- Yuanwei Zhu, Nan Qiao, Shuqi Dong, et al., Side-Chain Engineering of Polystyrene Dielectrics Toward High-Performance Photon Memories and Artificial Synapses, Chem. Mater. (2022) https://pubs.acs.org/doi/abs/10.1021/acs.chemmater.2c01252
- Xin Deng, Jing Xia, Bo Hu, et al., Hyjapones A−D, trimethylated acyphloroglucinol meroterpenoids from Hypericum japonicum thunb. With anti-inflammatory activity, Phytochem., 202, 113308 (2022) https://www.sciencedirect.com/science/article/abs/pii/S0031942222002242
- Jin Chen, XiXi Zeng, Ling Chen, Evolution of microstructures and hydrogen bond interactions within choline amino acid ionic liquid and water mixtures, Phys. Chem. Chem. Phys. (2022) https://pubs.rsc.org/en/content/articlehtml/2022/cp/d2cp01990f
- Danyang Zhang, Hongjuan Zhu, Chunrui Wang, et al., Three-Photon-Induced Singlet Excited-State Absorption for the Tunable Ultrafast Optical-Limiting in Distyrylbenzene: A First-Principles Study, Phys. Chem. Chem. Phys. (2022) https://pubs.rsc.org/en/content/articlelanding/2022/cp/d2cp01753a/
- Xin DENG, Jing XIA, Meng-Yu QIAN, et al., Ascyrones A–E, type B bicyclic ployprenylated acylphloroglucinol derivatives from Hypericum ascyron, Chin. J. Nat. Med., 20, 473 (2022) https://www.sciencedirect.com/science/article/pii/S1875536422601675
- Dule Hao, Zheng Zhang, Yu Cheng, et al., Rational design of ibuprofen-based redox-responsive anti-cancer polymeric drug delivery systems, J. Mater. Sci. (2022) https://link.springer.com/article/10.1007/s10853-022-07347-w (此文没有按要求引用Molclus!)
- Aobulikasimu Hasan, Meng Zhang, Zhan-Peng Shang, et al., Bioactive prenylated phenolic compounds from the aerial parts of Glycyrrhiza uralensis, Phytochem., 201, 113284 (2022) https://www.sciencedirect.com/science/article/abs/pii/S003194222200200X
- Bowei Yuan, Shi-Ya Tang, Shaodong Zhou, Striking Size and Doping Effects of Ti-Si-O Clusters on Methane Conversion Reactions, Chem. Eur. J. (2022) https://chemistry-europe.onlinelibrary.wiley.com/doi/abs/10.1002/chem.202201136
- Fangjian Shang, Runze Liu, Meiheng Lv, et al., Unraveling the Key Role of the Benzyl Group in the Synthesis of CL-20 Precursor HBIW, ACS Omega (2022) https://pubs.acs.org/doi/full/10.1021/acsomega.2c02161
- Yangyu Zhou, Hao Liu, Xiaoyang Jin, et al., Significant π Bonding in Coinage Metal Complexes OCTMCCO- from Infrared Photodissociation Spectroscopy and Theoretical Calculations, J. Chem. Phys. (2022) https://aip.scitation.org/doi/abs/10.1063/5.0099789
- Jianping Li, Christos T. Maravelias, Reid C. Van Lehn, Adaptive Conformer Sampling for Property Prediction Using the Conductor-like Screening Model for Real Solvents, Ind. Eng. Chem. Res. (2022) https://pubs.acs.org/doi/full/10.1021/acs.iecr.2c01163
- Jie Zhu, Dejian Shen, Baosheng Jin, Shengxing Wu, Theoretical investigation on the formation mechanism of carbonate ion in microbial self-healing concrete: Combined QC calculation and MD simulation, Constr. Build. Mater., 342, 128000 (2022) https://www.sciencedirect.com/science/article/abs/pii/S0950061822016695
- Yaya Wang, Kairui Zhang, Ruyi Li, et al., Dynamic hydrogen bonds promote C-H functionalization driven by Cl- anion, Tetrahedron Chem., 2, 100016 (2022) https://www.sciencedirect.com/science/article/pii/S2666951X22000122
- Jing Su, Liuyang Wang, Qixin Lu, et al., Alkali Resistance Mechanism of Cyano-containing Heterocyclic Disperse Dyes, J. Mol. Struct. (2022) https://www.sciencedirect.com/science/article/abs/pii/S0022286022010948
- Bo Zheng, Zipeng Liu, Ling Chen, et al., Effect of starch-catechin interaction on regulation of starch digestibility during hot-extrusion 3D printing: Structural analysis and simulation study, Food Chem., 393, 133394 (2022) https://www.sciencedirect.com/science/article/abs/pii/S0308814622013565 (此文没有按要求引用Molclus!)
- Yan Li, Kun Hu, Chao Huang, et al., Improvement of solubility, stability and antioxidant activity of carotenoids using deep eutectic solvent-based microemulsions, Coll. Surf. B, 217, 112591 (2022) https://www.sciencedirect.com/science/article/abs/pii/S0927776522002740 (此文没有按要求引用Molclus!)
- Yongyi Li, Shuai Zhao, Yunpeng Sun, et al., Automatic MS/MS Data Mining Strategy for Discovering Target Natural Products: A Case of Lindenane Sesquiterpenoids, Anal. Chem. (2022) https://pubs.acs.org/doi/full/10.1021/acs.analchem.2c01559
- Bo Tang, Mingxue Li, Xueyu Wang, et al., Formation of persistent free radicals and reactive chlorine species during photochemical processes of Polychlorophenols: Effect of temperature, humidity and particles, Chem. Eng. J., 446, 137149 (2022) https://www.sciencedirect.com/science/article/abs/pii/S1385894722026390
- Chunhui Li, Hui He, Chenxi Hou, et al., A quantum-chemistry and molecular-dynamics study of non-covalent interactions between tri-n-butyl phosphate and 1-butyl-3-methylimidazolium bis(trifluoromethylsulfonyl)imide, J. Mol. Liq., 360, 119430 (2022) https://www.sciencedirect.com/science/article/abs/pii/S0167732222009680
- Jinyun Zheng, Wenjie Li, Jiawei Zhang, et al., Effects of Flexible Group Length of Phosphonate Monomers on the Performance of Gel Polymer Electrolytes for Sodium-Ion Batteries with Ultralong Cycling Life, ACS Sustainable Chem. Eng. (2022) https://pubs.acs.org/doi/abs/10.1021/acssuschemeng.2c01531
- Wei-Lu Ding, Tao Zhang, Yanlei Wang, et al., Machine Learning Screening of Efficient Ionic Liquids for Targeted Cleavage of the β–O–4 Bond of Lignin, J. Phys. Chem. B (2022) https://pubs.acs.org/doi/abs/10.1021/acs.jpcb.1c10684
- Mengmeng He, Wenke Li, Xiaoqiang Liu, et al., CCSD(T)/CBS法标定CO2和CH4转化为CH3COOH的活化能, J. Sichuan Univ. Sci. Eng., 35, 22 (2022) http://xb.suse.edu.cn/xbzk/files/2022/2/20220507115405_8552.pdf (此文没有按要求引用Molclus!)
- Hui Zhou, Yu Zhou, Han Yong Bae, et al., Organocatalytic stereoselective cyanosilylation of small ketones, Nature, 605, 84 (2022) https://www.nature.com/articles/s41586-022-04531-5
- Wen Liu, Lulu Huang, Jin Hu, Xiaopeng Xing, Various Bond Interactions between NO and Anionic Gold Clusters: A Theoretical Calculation, Phys. Chem. Chem. Phys. (2022) https://pubs.rsc.org/en/content/articlelanding/2022/cp/d1cp05213f/
- Long Chen, Yu Huang, Yonggang Xue, et al., Kinetic and Mechanistic Investigations of OH-Initiated Atmospheric Degradation of Methyl Butyl Ketone, J. Phys. Chem. A (2022) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.2c01126
- Sheng Fan, Xinru Lu, Haoliang Li, et al., Efficient removal of organophosphate esters by ligand functionalized MIL-101 (Fe): Modulated adsorption and DFT calculations, Chemosphere (2022) https://www.sciencedirect.com/science/article/abs/pii/S0045653522013741
- Yaya Wang, Kairui Zhang, Ruyi Li, et al., Dynamic hydrogen bonds promote C–H functionalization driven by Cl− anion, Tetrahedron Chem (2022) https://www.sciencedirect.com/science/article/pii/S2666951X22000122
- Jiguang Li, Zhiwei Ma, Da Lei, et al., Precisely modulated electrostatic attraction to the recognition site for on-site ultrafast visualization of trace hydrazine, Cell. Rep. Phys. Sci. (2022) https://www.sciencedirect.com/science/article/pii/S2666386422001527
- Ruoxi Liu, Leyao Wang, Suneng Liu, et al., Quantum Chemistry Research on an Inorganic Chemistry Problem: Structure Search and Properties of P42- Clusters, Univ. Chem. (2022) http://www.dxhx.pku.edu.cn/CN/10.3866/PKU.DXHX202112011
- Yumeng Zhang, Zhouming Zhao, Kai Wang, et al., Molecular docking assisted exploration on solubilization of poorly soluble drug remdesivir in sulfobutyl ether-tycyclodextrin, AAPS Open, 8, 9 (2022) https://aapsopen.springeropen.com/articles/10.1186/s41120-022-00054-5
- M.J. Bezbaruah, P. Barukial, B. Ahmed, et al., Physicochemical Properties and Structural Analysis of Some Organic Solvent (OS)-Water Systems: A Quantum Mechanical Study, Phys. Chem. Res., 10, 559 (2022) https://www.physchemres.org/article_148721_0c9636f4e27ada732d216d095a58e37e.pdf
- Haijun Long, De Guo, Liyuan Wang, et al., Aggregation induced emission enhancement and chromism properties of a vinyl bridged naphthalene diimide dimer, Dyes Pigments, 203, 110330 (2022) https://www.sciencedirect.com/science/article/abs/pii/S0143720822002522 (此文没有按要求引用Molclus!)
- Jun-Yao Zhang, Bei-Bei Yang, Ya-Dong Yang, et al., Specific chiroptical sensing of cysteine via ultrasound-assisted formation of disulfide bonds in aqueous solution, Ultrason. Sonochem., 86, 106007 (2022) https://www.sciencedirect.com/science/article/pii/S1350417722001006
- Huimeng Wang, Yi Fan, Yaqi Hou, et al., Host-guest liquid gating mechanism with specific recognition interface behavior for universal quantitative chemical detection, Nat. Commun., 13, 1906 (2022) https://www.nature.com/articles/s41467-022-29549-1
- Mengna Luo, Shanshan Chang, Yihong Li, et al., Molecular Networking-Based Screening Led to the Discovery of a Cyclic Heptadepsipeptide from an Endolichenic Xylaria sp., J. Nat. Prod. (2022) https://pubs.acs.org/doi/abs/10.1021/acs.jnatprod.1c01108
- Yunxiang Dai, Jinfeng Zhong, Jiaqi Li, et al., Interaction mechanism of cholesterol/β-cyclodextrin complexation by combined experimental and computational approaches, Food Hydrocolloids (2022) https://www.sciencedirect.com/science/article/abs/pii/S0268005X22002454
- Limin Lu, Bin Shi, Tianyu Tang, et al., Spectral Analysis of Epinephrine Molecule Based on Density Functional Theory, Spectro. Spect. Anal., 42, 248 (2022) https://www.opticsjournal.net/Articles/OJ1a843728da7ffe72/FullText
- Hao Wang, Peng Wu, Changyong Li, Reversible and Efficient Absorption of SO2 with Natural Amino Acid Aqueous Solutions: Performance and Mechanism, ACS Sustainable Chem. Eng. (2022) https://pubs.acs.org/doi/abs/10.1021/acssuschemeng.1c08186
- Junchao Ma, Chao Qin, Michael Gatheru Waigi, et al., Functional group substitutions influence the binding of benzophenone-type UV filters with DNA, Chemosphere, 299, 134490 (2022) https://www.sciencedirect.com/science/article/abs/pii/S0045653522009833
- RuiLiu, Xiaoli Qin, Xiong Liu, et al., Host-guest interactions between oleic acid and β-cyclodextrin: A combined experimental and theoretical study, Food Chem. (2022) https://www.sciencedirect.com/science/article/abs/pii/S030881462200872X (此文没有按要求引用Molclus!)
- Dong Liu, Song Xu, Gerui Pei, et al., Geometries, electronic structures, and bonding properties of endohedral Group-14 Zintl clusters TM@E10 (TM = Fe, Co, Ni; E = Ge, Sn, Pb), J. Comput. Chem. (2022) https://onlinelibrary.wiley.com/doi/abs/10.1002/jcc.26838
- Yuzhi Song, Ming Qin, Songsong Liu, et al., Theoretical perspective of relationship between molecular structure and luminescence properties for circularly polarized thermally activated delayed fluorescence, Spectrochim. Acta A (2022) https://www.sciencedirect.com/science/article/abs/pii/S1386142522003134
- Hechen Wu, Xiao-Nan Wu, Xuegang Liu, et al., Iridium Dimer Anion-Mediated C≡C Triple Bond Cleavage and Successive Dehydrogenation of Acetylene in the Gas Phase, J. Phys. Chem. A (2022) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.2c00664
- Da Zhang, DaWei Zhu, WenYao Guo, et al., The Fluorine-Rich Electrolyte as an Interface Modifier to Stabilize Lithium Metal Battery at Ultra-Low Temperature, Adv. Funct. Mater. (2022) https://onlinelibrary.wiley.com/doi/abs/10.1002/adfm.202112764 (此文没有按要求引用Molclus!)
- Jie Ma, Yan Qiu, Junying Zhao, et al., Effect of Agricultural Organic Inputs on Nanoplastics Transport in Saturated Goethite-Coated Porous Media: Particle Size Selectivity and Role of Dissolved Organic Matter, Environ. Sci. Technol. (2022) https://pubs.acs.org/doi/abs/10.1021/acs.est.1c07574
- Yuntao Zhang, GAS-PHASE ACID-BASE PROPERTIES AND CONFORMATIONS OF OLIGOPEPTIDES THROUGH MASS SPECTROMETRY AND OLIGOPEPTIDES THROUGH MASS SPECTROMETRY AND COMPUTATIONAL STUDIES COMPUTATIONAL STUDIES, Dissertation of University of the Pacific (2022) https://scholarlycommons.pacific.edu/cgi/viewcontent.cgi?article=4785&context=uop_etds
- Yaya Wang, Kairui Zhang, Ruyi Li, et al., Hydrogen bonds promote site-selective phenol functionalization enabled by Cl-anion, ChemRxiv (2022) https://chemrxiv.org/engage/chemrxiv/article-details/6219c89d011b5818eeb54519
- Simei Sun, Song Zhang, Jiao Song, et al., Ultrafast proton transfer dynamics of 2-(2'-hydroxyphenyl)benzoxazole dye in different solvents, Chin. Phys. B (2021) https://iopscience.iop.org/article/10.1088/1674-1056/ac3734/meta
- Mengdi Guo, Qiuhao Yi, Chaonan Cui, et al., Gas-Phase Synthesis of Metal Olefins: Plasma-Assisted Methane Dehydrogenation and C═C Bond Formation, J. Phys. Chem. A (2022) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.1c10012
- Ziyi Wang , Ruimin Song, Weigen Chen, et al., Vibrational Spectra and Molecular Vibrational Behaviors of Dibenzyl Disulfide, Dibenzyl Sulphide and Bibenzyl, Int. J. Mol. Sci., 23, 1958 (2022) https://www.mdpi.com/1422-0067/23/4/1958/htm
- Ni Shen, Runhan Li, Can Liu, et al., Photocatalytic Cross-Couplings of Aryl Halides Enabled by o-Phosphinophenolate and o-Phosphinothiophenolate, ACS Catal., 12, 2788 (2022) https://pubs.acs.org/doi/abs/10.1021/acscatal.1c05941
- Yan-rong Zhao, Guo-an Zou, Haji Akber Aisa, Guaianolides and unusual 3-oxa-guaianolides from Artemisia macrocephala, Phytochem., 197, 113108 (2022) https://www.sciencedirect.com/science/article/abs/pii/S0031942222000243
- Ying Tian, Jiayi Li, Han Hu, et al., Acid-Triggered, degradable and high strength-toughness copolyesters: Comprehensive experimental and theoretical study, J. Hazard. Mater. (2022) https://www.sciencedirect.com/science/article/abs/pii/S0304389422001807
- Eduardo Romero-Montalvo, Gino A. DiLabio, Computational Study of Hydrogen Bond Interactions in Water Cluster–Organic Molecule Complexes, J. Phys. Chem. A, 125, 13369 (2021) https://pubs.acs.org/doi/10.1021/acs.jpca.1c01377
- Jianan Chen, Zhipeng Li, Yanrong Jiang, et al., Study of electronic structures and the micro-solvation effect of SO3– and HSO3– in atmospheric aerosols, J. East China Normal Univ., 2022, 31 https://xblk.ecnu.edu.cn/CN/10.3969/j.issn.1000-5641.2022.01.005
- Hongru Zhang, Bingjie Huo, Mengya Sun, et al., The mechanism explosion of separating binary azeotropic system with intermediate-boiling-point solvent based on vapor–liquid equilibrium experiment, quantum chemical calculation and molecular dynamics simulation, J. Chem. Thermodyn., 168, 106730 (2022) https://www.sciencedirect.com/science/article/abs/pii/S002196142200009X (此文没有按要求引用Molclus!)
- Yu-ling Liu, He Huang, Yong-jin Peng, Fluorescent Probe for Simultaneous Detection of Human Serum Albumin and Sulfite: A Theoretical Analysis, J. Mol. Struct. (2022) https://www.sciencedirect.com/science/article/abs/pii/S0022286022001144
- Senqi Guo, Chun Zhu, Guoqing Chen, et al., A theoretical study on intermolecular hydrogen bonds of isopropanol-water clusters, Theor. Chem. Acc., 141, 6 (2022) https://link.springer.com/article/10.1007/s00214-022-02865-x
- Shuyi Zong, Jingkang Wang, Xin Huang, et al., Formation and stabilization mechanism of mesoscale clusters in solution, IUCrJ (2022) https://journals.iucr.org/m/issues/2022/02/00/lt5045/index.html (此文没有按要求引用Molclus!)
- Wen-jun Hu, Rui-jun Gou, Shu-hai Zhang, et al., Theoretical investigation on the intermolecular interactions between 3-nitro-1,2,4-triazol-5-one and 2,6-diamino-3,5-dinitropyrazine-1-oxide using DFT methods, Chem. Papers (2022) https://link.springer.com/article/10.1007/s11696-021-02059-y
- Xuecheng Zhou, Shasha Yang, Huan Yang, et al., Mechanism of heteroatom-doped Cu5 catalysis for hydrogen evolution reaction, Int. J. Hyd. Energy (2022) https://www.sciencedirect.com/science/article/abs/pii/S0360319921048321
- Xinyu Wang, Baoyou Liu, Huilong Yang, Jie Tian, Properties of binary mixtures of a novel Natural deep eutectic solvent (glycolic acid + xylitol) and water at several temperatures, Fluid Phase Equilibr. (2022) https://www.sciencedirect.com/science/article/abs/pii/S0378381222000152
- Xuwei Liu, Jiayi Li, Agnès Rolland-Sabaté, et al., Experimental and theoretical investigation on interactions between xylose-containing hemicelluloses and procyanidins, Carbohyd. Polym. (2022) https://www.sciencedirect.com/science/article/abs/pii/S0144861721014739
- Zisheng Zhang, Amanda L. Kummeth, Jenny Y. Yang, Anastassia N. Alexandrova, Inverse Molecular Design of Alkoxides and Phenoxides for Aqueous Direct Air Capture of CO2, ChemRxiv (2022) https://chemrxiv.org/engage/chemrxiv/article-details/61cca090a53f1b0b86ac318b
- Jing-jing DENG, Ze-jun LUO, Chu WANG, Xi-feng ZHU, Extraction of phenols from bio-oil aqueous fraction by hydrophobic ionic liquids, J. Fuel Chem. Technol., 49, 1832 (2021) https://www.sciencedirect.com/science/article/abs/pii/S1872581321601081
- Wen-yu Zheng, Xiao-mei Wu, Ming-xuan Li, et al., Synergistic strongly coupled super-deamidation of wheat gluten by glucose-organic acid natural deep eutectic solvent and the efficaciousness of structure and functionality, Food Hydrocolloid. (2021) https://www.sciencedirect.com/science/article/abs/pii/S0268005X21008535(此文没有按要求引用Molclus!)
- Yanran Chen, Faqiong Zhao, Baizhao Zeng, Fabrication of surface molecularly imprinted electrochemical sensor for the sensitive quantification of chlortetracycline with ionic liquid and MWCNT improving performance, Talanta, 239, 123130 (2022) https://www.sciencedirect.com/science/article/abs/pii/S0039914021010523 (此文没有按要求引用Molclus!)
- Liheng Chen, Si-Man Luo, Cong-Min Huo, et al., New insight into lignin aggregation guiding efficient synthesis and functionalization of a lignin nanosphere with excellent performance, Green Chem. (2021) https://pubs.rsc.org/en/content/articlelanding/2021/gc/d1gc03651c/
- Da Zhang, Rong Gu, Wenyao Guo, et al., Long-Life and High-Rate-Charging Lithium Metal Batteries Enabled by a Flexible Active Solid Electrolyte Interphase Layer, ACS Appl. Mater. Interfaces (2021) https://pubs.acs.org/doi/abs/10.1021/acsami.1c19952 (此文没有按要求引用Molclus!)
- Wen-Shuai Dai, Bin Yang, Shuai-Ting Yan, et al., Structural and Electronic Properties of LaSin–/0 (n = 2–6) Clusters: Anion Photoelectron Spectroscopy and Density Functional Calculations, J. Phys. Chem. A (2021) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.1c08487
- Han Hua, Jiayi Li, Shenggan Luo, et al., Design of 2,5-Furandicarboxylic Based Polyesters Degraded in Different Environmental Conditions: Comprehensive Experimental and Theoretical Study, J. Hazard. Mater. (2021) https://www.sciencedirect.com/science/article/abs/pii/S0304389421027217 (此文没有按要求引用Molclus!)
- Chengmin Gui, Zhiwei Li, Zhigang Lei, Yichun Dong, Combinatorial screening of ionic liquid extractant for removal of methanol from methylal, Chem. Eng. Sci., 249, 117317 (2022) https://www.sciencedirect.com/science/article/pii/S0009250921008824
- Leiqiang Han, Shuang Liang, Weiwei Mu, et al., Amphiphilic small molecular mates match hydrophobic drugs to form nanoassemblies based on drug-mate strategy, Asian J. Pharm. Sci. (2021) https://www.sciencedirect.com/science/article/pii/S1818087621001033 (此文没有按要求引用Molclus!)
- Menghang Li, High throughput nucleic acid solid phase separation process based on microscale carrier, Msc dissertation of Dalian University of Technology (2021) http://doi.org/10.26991/d.cnki.gdllu.2021.001919 (此文没有按要求引用Molclus!)
- Shuai Qian, Xiaoyang Liu, C. Heath Turner, Jason E. Bara, Glycerol-derived Solvents Containing Two or Three Distinct Functional Groups Enabled by Trifluoroethyl Glycidyl Ether, AIChE J. (2021) https://aiche.onlinelibrary.wiley.com/doi/abs/10.1002/aic.17533
- Yi-Cong Lou, Pu Jing, DFT study of the stabilization effect on anthocyanins via secondary interactions, Food Chem.: Mol. Sci., 3, 100057 (2021) https://www.sciencedirect.com/science/article/pii/S2666566221000484 (此文没有按要求引用Molclus!)
- Qiang Guo, Jincheng Zhang, Xiaoyu Li, et al., Photophysical and electrochemical properties of alternating copolymers consisting of 3,4-ethylenedioxythiophene and 4,4′-diphenylamine/3,6-carbazole, Polym.-Plast. Tech. Mat. (2021) https://www.tandfonline.com/doi/abs/10.1080/25740881.2021.2005095
- Tianyun Jin, Pinglin Li, Cili Wang, et al., Jellynolide A, pokepola esters, and sponalisolides from the aquaculture sponge Spongia officinalis L, Phytochem., 194, 113006 (2022) https://www.sciencedirect.com/science/article/pii/S0031942221003551
- Shiliang Chen, Zhijing Wu, Qiujin Fan, Jiachi Huang, Graphene-oxide-bacterial-cellulose nanohybrid gives a “substrate-driven enhancement” effect to catalytic activity of phthalocyanine, Cellulose (2021) https://link.springer.com/article/10.1007/s10570-021-04336-1
- Hui Liu, Hao Chen, Xiaofeng Xie, et al., Low-temperature Hg0 abatement by ionic liquid based on weak interaction, J. Hazard. Mater. (2021) https://www.sciencedirect.com/science/article/pii/S0304389421028053
- Xiaomin Qiu, Yajuan Qu, Mengjing Zhou, et al., Comparison of Deep Eutectic Solvents and Organic Solvent Effects on the Separation of Ternary Azeotropes by the Experimental Study and Molecular Simulation, ACS Sustainable Chem. Eng. (2021) https://pubs.acs.org/doi/abs/10.1021/acssuschemeng.1c06379 (此文没有按要求引用Molclus!)
- Zihao Fu, Hong-Bin Xie, Jonas Elm, et al., Atmospheric Autoxidation of Organophosphate Esters, Environ. Sci. Technol. (2021) https://pubs.acs.org/doi/abs/10.1021/acs.est.1c04817
- Jinlong Fan, Gang Wang, Shuo Xu, et al., Removal of impurities from bismuth pickling solution using solvent extraction with TBP, Hydrometallurgy, 207, 105779 (2022) https://www.sciencedirect.com/science/article/abs/pii/S0304386X21002280
- Xiaoyang Liu, C.Heath Turner, Understanding Gas Absorption in Multivalent Ionic Liquids via Solute-Solvent Interaction Analyses, Chem. Phys. Lett. (2021) https://www.sciencedirect.com/science/article/pii/S0009261421008873
- Simei Sun, Song Zhang, Jiao Song, et al., Ultrafast proton transfer dynamics of 2-(2'-hydroxyphenyl)benzoxazole dye in different solvents, Chin. Phys. B (2021) https://iopscience.iop.org/article/10.1088/1674-1056/ac3734/meta
- Chen Liu, Haiyong Zhang, Yonggang Wang, et al., Extraction separation of aromatic homologues from n-decane using DMSO: Influence of the alky side chain length, J. Chem. Thermodyn. (2021) https://www.sciencedirect.com/science/article/abs/pii/S0021961421002871
- Eman Nabil, Ahmed A. Hasanein, Rua B. Alnoman, Mohamed Zakaria, Optimizing the Cosensitization Effect of SQ02 Dye on BP-2 Dye-Sensitized Solar Cells: A Computational Quantum Chemical Study, J. Chem. Inf. Model. (2021) https://pubs.acs.org/doi/abs/10.1021/acs.jcim.1c00739
- Shideng Yuan, Shasha Liu, Yingzhe Du, et al., Atomistic insights into heterogeneous reaction of formic acid on mineral oxide particles, Chemosphere, 287, 132430 (2022) https://www.sciencedirect.com/science/article/pii/S0045653521029027
- Chen Liu, Di Zhao, Haiyong Zhang, et al., Cumene extraction separation from alkanes using DMSO: Influence of the alkane structure, Fluid Phase Equilibr. (2021) https://www.sciencedirect.com/science/article/abs/pii/S0378381221002995
- Yanli Zhang, Xiaomin Qiu, Hongru Zhang, et al., Quantum chemistry of solvent selectivity and economic, exergy and environment analysis of extractive distillation process for separating bi nary azeotropic mixture, J. Taiwan Inst. Chem. E (2021) https://www.sciencedirect.com/science/article/pii/S1876107021005423
- Chen Liu, Haiyong Zhang, Yonggang Wang, et al., The physical nature of the interaction in DMSO extraction separation of C8H10 isomers/n-decane systems, Phys. Chem. Chem. Phys. (2021) https://pubs.rsc.org/en/content/articlelanding/2021/cp/d1cp03128g/
- Han Hu, Jiayi Li, Ying Tian, et al., Design of High-Barrier and Environmentally Degradable FDCA-Based Copolyesters: Experimental and Theoretical Investigation, ACS Sustainable Chem. Eng. (2021) https://pubs.acs.org/doi/abs/10.1021/acssuschemeng.1c04687
- Jia Zhang, Ya-Nan Yang, Jian-Shuang Jiang, et al., New dimeric phloroglucinol derivatives from Agrimonia pilosa and their hepatoprotective activities, Bioorg. Chem., 116, 105341 (2021) https://www.sciencedirect.com/science/article/abs/pii/S0045206821007185
- Hewei Song, Amit Kumar, Yuling Zhang, A novel approach for the removal of Pb2+ and Cd2+ from wastewater by sulfur-ferromagnetic nanoparticles (SFMNs), Chemosphere (2021) https://www.sciencedirect.com/science/article/abs/pii/S004565352102628X (此文没有按要求引用Molclus!)
- Hechen Wu, Xiao-Nan Wu, Xiaoyang Jin, et al., Quadruple C–H Bond Activations of Methane by Dinuclear Rhodium Carbide Cation [Rh2C3]+, JACS Au (2021) https://pubs.acs.org/doi/full/10.1021/jacsau.1c00265
- Yongpan Tian, Wenwen Chen, Chengcheng Wang, et al., Computational study for the electrophilic reactivity prediction of crown ethers, J. Mol. Liq., 341, 117418 (2021) https://www.sciencedirect.com/science/article/abs/pii/S0167732221021425
- Zilin Meng, Zikai Liu, Jiaxuan Fan, et al., Perfluoro octanoic acid-modified magnetic hyperbranched polyamideamine as a sorbent for the extraction of fluorine-containing pesticides from water samples, J. Sep. Sci. (2021) https://analyticalsciencejournals.onlinelibrary.wiley.com/doi/abs/10.1002/jssc.202100502
- Yao Lu, Xiao fan Hu, Won Hee Lee, et al., Effects of bulky 2,2′-substituents in dianhydrides on the microstructures and gas transport properties of thermally rearranged polybenzoxazoles, J. Membrane Sci., 639, 119777 (2021) https://www.sciencedirect.com/science/article/pii/S0376738821007213 (此文没有按要求引用Molclus!)
- Chun-Xiao Jiang, Bo Yu, Ya-Mei Miao, et al., Indole Alkaloids from a Soil-Derived Clonostachys rosea, J. Nat. Prod. (2021) https://pubs.acs.org/doi/abs/10.1021/acs.jnatprod.1c00457
- Xuexian Gu, Computer simulation of molecularly imprinted material based on tetrachlorodibenzo-p-dioxin, New Chem. Mater., 49, 171 (2021) https://www.hgxx.org/CN/10.19817/j.cnki.issn%25201006-3536.2021.07.039
- Arafat Md Yasir, Jie Ma, Xiaoxue Ouyang, et al., Effects of selected functional groups on nanoplastics transport in saturated media under diethylhexyl phthalate co-contamination conditions, Chemosphere (2021) https://www.sciencedirect.com/science/article/pii/S0045653521024371
- Zhihao Zhou, Qingquan Qiu, Yanxing Zhao, et al., Calculations and analyses of molecular features and properties of nitrogen / carbon tetrafluoride mixture, Comput. Theor. Chem. (2021) https://www.sciencedirect.com/science/article/pii/S2210271X21002693
- Yan Huang, Le-Wu Zhan, Qian Zhang, et al., Theoretical research on cage-like furazan-based energetic compounds and its derivatives, J. Mol. Model., 27, 243 (2021) https://link.springer.com/article/10.1007/s00894-021-04849-3
- Xiaoyang Liu, C.Heath Turner, Computational Study of the Electrostatic Potential and Charges of Multivalent Ionic Liquid Molecules, J. Mol. Liq. (2021) https://www.sciencedirect.com/science/article/pii/S0167732221019140
- Tianyun Jin, Pinglin Li, Cili Wang, et al., Two new spongian diterpene derivatives from the aquaculture sponge Spongia officinalis Linnaeus, 1759, Nat. Prod. Res. (2021) https://www.tandfonline.com/doi/abs/10.1080/14786419.2021.1961137
- Xiaoyan Ding, Xin Song, Xing Chen, et al., Degradation and mechanism of hexafluoropropylene oxide dimer acid by thermally activated persulfate in aqueous solutions, Chemosphere, 286, 131720 (2022) https://www.sciencedirect.com/science/article/abs/pii/S0045653521021925
- Baoyou Liu, Jie Tian, Investigation of Glycolic Acid Natural Deep Eutectic Solvents with Strong Proton Donors for Ammonia Capture and Separation, Ind. Eng. Chem. Res. (2021) https://pubs.acs.org/doi/abs/10.1021/acs.iecr.1c01456
- Li-Li Tian, Hao Ren, Jun-Min Xi, et al., Diverse anti-inflammation and anti-cancer polyketides isolated from the endophytic fungi Alternaria sp. MG1, Fitoterapia (2021) https://www.sciencedirect.com/science/article/abs/pii/S0367326X21001751
- Jin Chen, Xixi Zeng, Ling Chen, Regulation nature of water-choline amino acid ionic liquid mixtures on the disaggregation behavior of starch, Carbohyd. Polym. (2021) https://www.sciencedirect.com/science/article/abs/pii/S0144861721008614
- Biyu Jin, Dongyun Wang, Juan Zhu, et al., A Self-Healable Polyelectrolyte Binder for Highly Stabilized Sulfur, Silicon, and Silicon Oxides Electrodes, Adv. Funct. Mater. (2021) https://onlinelibrary.wiley.com/doi/abs/10.1002/adfm.202104433 (此文没有按要求引用Molclus!)
- Cui-Mei Li, Di Wu, Xiao Tian, et al., Probing the effect of carbon doping on structures, properties, and stability of magnesium clusters, Theor. Chem. Acc., 140, 111 (2021) https://link.springer.com/article/10.1007/s00214-021-02810-4
- Mengdi Guo, Ben Huang, Qiuhao Yi, Zhixun Luo, Gas-Phase Synthesis and Deposition of Metal-bipyridine Complex [M-bpy1-2]+ (M=Ag, Cu) with Conformation Transition, Phys. Chem. Chem. Phys. (2021) https://pubs.rsc.org/en/content/articlehtml/2021/cp/d1cp02399c
- Aikebaier Jumai, Ruxianguli Rouzimaimaiti, Guo-An Zou, Haji Akber Aisa, Pyrrolizidine alkaloids and unusual millingtojanine A-B from Jacobaea vulgaris (syn. Senecio jacobaea L.), Phytochemistry, 190, 112862 (2021) https://www.sciencedirect.com/science/article/abs/pii/S0031942221002119
- Jiake Fan, Baoyun Ye, Chongwei An, Jingyu Wang, Theoretical calculation on the interaction mechanism between 2,6‐diamino‐3,5‐dinitropyrazine‐1‐oxide and ammonium perchlorate, J. Energ. Mater. (2021) https://www.tandfonline.com/doi/abs/10.1080/07370652.2021.1948150
- Xu Zhao, Xinke Zhang, Xiaoming He, et al., Acetylation of Malvidin-3-O-glucoside Impedes Intermolecular Copigmentation: Experimental and Theoretical Investigations, J. Agric. Food Chem. (2021) https://pubs.acs.org/doi/10.1021/acs.jafc.1c02378
- Shideng Yuan, Hua Zhang, Yuxi Wang, et al., Atomistic insights into heterogeneous reaction of hydrogen peroxide on alumina particles: Combining DFT calculation and ReaxFF molecular dynamics simulations, Coll. Surf. A (2021) https://www.sciencedirect.com/science/article/abs/pii/S092777572100933X
- Congyi Peng, Lei Ma, Yiming He, et al., Theoretical Study on the Structure and Properties of AlnPn (n = 2~9) Clusters, Modern Physics, 11, 41, (2021) https://www.hanspub.org/journal/PaperInformation.aspx?paperID=42466(此文没有按要求引用Molclus!)
- Wen Su, Yaling Zhu, Chenggang Qi, Teaching Geometries of Coordination Compounds Using Computational Chemistry: Examples of Two Kinds of Four-coordinate Complexes, Front. Edu. Res., 4, 45 (2021) https://francis-press.com/uploads/papers/KGCHeNldgXUH7JjW0s00jT3LVIQ9xlAa2pyYMAeR.pdf
- Zikai Liu, Zilin Meng, Jiaxuan Fan, et al., Use of 1-octyl-3-methylimidazole hexafluorophosphate modified magnetic hyperbranched polyamideamine as sorbent for the extraction of pyrethroid insecticides from tea infusion, J. Sep. Sci. (2021) https://analyticalsciencejournals.onlinelibrary.wiley.com/doi/abs/10.1002/jssc.202100230
- Shucheng Qin, Lei Meng, Yongfang Li, Molecular Properties and Aggregation Behavior of Small-Molecule Acceptors Calculated by Molecular Simulation, ACS Omega (2021) https://pubs.acs.org/doi/full/10.1021/acsomega.1c01394
- Tian‐Yun Jin, Ping‐Lin Li, Ci‐Li Wang, Racemic Bisindole Alkaloids: Structure, Bioactivity, and Computational Study, Chin. J. Chem. (2021) https://onlinelibrary.wiley.com/doi/abs/10.1002/cjoc.202100255
- Shasha Lv, Xiyu Li, Li Yang, et al., Computational Design of Photoswitchable Anion Receptors: Red-Shifted and Bistable Di-ortho-fluoro Di-ortho-chloro Azobenzene Derivatives, Chem. Phys. (2021) https://www.sciencedirect.com/science/article/abs/pii/S0301010421001579
- Cong-Min Huo, Liheng Chen, Hai-Yang Wang, et al., Density Functional Theory-guided Drug Loading Strategy for Sensitized Tumor-homing Thermotherapy, Chem. Eng. J. (2021) https://www.sciencedirect.com/science/article/abs/pii/S1385894721017319(此文没有按要求引用Molclus!)
- Hailong Wang, Qiuping Guan, Xueye Wang, Theoretical research of covalent and controllable molecular brake based on 9-triptycene, Theor. Chem. Acc. (2021) https://link.springer.com/article/10.1007/s00214-021-02762-9
- Wei Du, Yue Gong, Yunfei Xu, et al., Can the Crystal of Conformational Polymorph Nucleate Directly from Its Conformer? The Case of Flufenamic Acid, Cryst. Growth Des. (2021) https://pubs.acs.org/doi/abs/10.1021/acs.cgd.1c00362
- Haoran Sun, Jiajia Wang, Bo Zhen, et al., Polycyclic polyprenylated acylphloroglucinol derivatives from Hypericum pseudohenryi, Phytochem., 187, 112761 (2021) https://www.sciencedirect.com/science/article/abs/pii/S0031942221001102
- Yanrong Jiang, Qinqin Yuan, Wenjin Cao, et al., Gaseous Cyclodextrin-closo-Dodecaborate Complexes ΧCD·[B12X12]2- (Χ = α, β, γ; X = F, Cl, Br, I): Electronic Structures and Intramolecular Interactions, Phys. Chem. Chem. Phys. (2021) https://pubs.rsc.org/en/content/articlehtml/2021/cp/d1cp01131f
- Yu-Jiao Dong, Bo Zhu, Yun Geng, et al., Radical Mechanism of IrIII/NiII-Metallaphotoredox Catalyzed C(sp3)−H Functionalization Triggered by Proton-Coupled Electron Transfer: Theoretical Insight, CCS Chem. (2021) https://www.chinesechemsoc.org/doi/pdf/10.31635/ccschem.021.202100802
- Shideng Yuan, Shasha Liu, Xueyu Wang, et al., Atomistic Insights into Heterogeneous Reaction of Hydrogen Peroxide on Mineral Oxide Particles, Appl. Surf. Sci. (2021) https://www.sciencedirect.com/science/article/abs/pii/S0169433221007832
- BailiLi, Xuan Ji, Lixian Tian, et al., Sequence modulation of tunneling barrier and charge transport across histidine doped oligo-alanine molecular junctions, Chin. Chem. Lett. (2021) https://www.sciencedirect.com/science/article/abs/pii/S1001841721002540
- Yanbo Li, Weiye Chen, Long Zhu, et al., Intramolecular CH3-Migration-Controlled Cation Reactions in the VUV Photochemistry of 2-Methyl-3-Buten-2-ol Investigated by Synchrotron Photoionization Mass Spectrometry and Theoretical Calculations, Phys. Chem. Chem. Phys. (2021) https://pubs.rsc.org/en/content/articlelanding/2021/cp/d1cp00490e/
- Limin Man, Yu Yang, Hui Wang, et al., In Situ-Cross-linked Supramolecular Eco-Binders for Improved Capacity and Stability of Lithium–Sulfur Batteries, ACS Appl. Energy Mater. (2021) https://pubs.acs.org/doi/abs/10.1021/acsaem.1c00207(此文没有按要求引用Molclus!)
- Jiguang Du, Gang Jiang, An aromatic Ca2B8 complex for reversible hydrogen storage, Int. J. Hyd. Energy (2021) https://www.sciencedirect.com/science/article/abs/pii/S036031992100906X
- Hao-Ran Sun, Jia-Jia Wang, Bo Zhen, et al., Hypseudohenrins I − K: three new polycyclic polyprenylated acylphloroglucinol derivatives from Hypericum pseudohenryi, J. Asian Nat. Prod. Res. (2021) https://www.tandfonline.com/doi/full/10.1080/10286020.2021.1906232
- Liu He, Li Zhang, Ying Wan, Nong Li, Yiding Ren, Structures and Energetics of Elemental Sulfur in Hydrogen Sulfide, J. Clust. Sci. (2021) https://link.springer.com/article/10.1007/s10876-021-02046-z
- Yuan-Fei Zhou, Kun Hu, Fang Wang, et al., 3-Hydroxy-4-methyldecanoic Acid-Containing Cyclotetradepsipeptides from an Endolichenic Beauveria sp., J. Nat. Prod. (2021) https://pubs.acs.org/doi/abs/10.1021/acs.jnatprod.0c01305
- Cong Liu, Yanhui Li, Xu Cui, et al., Construction of a recyclable dual-responsive TiO2-based photocatalyst modified with ZnIn2S4 nanosheets and zinc phthalocyanine for Cr(VI) reduction under visible light, Chem. Eng. J., 417, 129332 (2021) https://www.sciencedirect.com/science/article/abs/pii/S1385894721009207(此文没有按要求引用Molclus!)
- Chang Wang, Jianbao Wu, Xinxin Zhao, et al., Lithium Bond-Enhanced Capacity of Dipyridyl Polysulfides for LSBs, ACS Appl. Energy Mater. (2021) https://pubs.acs.org/doi/abs/10.1021/acsaem.0c03236
- Liu Sifan, Xu Zanxin, Zhang Guangjie, Dong Junxing, Lignan derivatives and a jasmonic acid derivative from the seeds of Orychophragmus violaceus, Nat. Prod. Res. (2021) https://www.tandfonline.com/doi/abs/10.1080/14786419.2021.1886100(此文没有按要求引用Molclus!)
- Hongru Zhang, Fei Zhao, Zhaoyuan Ma, et al., Design and optimization for the separation of cyclohexane-isopropanol-water using mixed extractants with thermal integration based on molecular mechanism, Sep. Purif. Technol. (2021) https://www.sciencedirect.com/science/article/abs/pii/S1383586621002422
- Sibo Lin, Jenna C. Fromer, Yagnaseni Ghosh, et al., Computer-assisted catalyst development via automated modelling of conformationally complex molecules: application to diphosphinoamine ligands, Sci. Rep., 11, 4534 (2021) https://www.nature.com/articles/s41598-021-82816-x
- Dezhi Yang, Junzi Cao, Tianyu Heng, et al., Theoretical Calculation and Structural Analysis of the Cocrystals of Three Flavonols with Praziquantel, Cryst. Growth Des. (2021) https://pubs.acs.org/doi/abs/10.1021/acs.cgd.0c01706
- Qi Yu, Jingyi Feng, Jie Li, et al., Mechanisms of aromatic molecule - oxygen-containing functional group interactions on carbonaceous material surfaces, Chemosphere (2021) https://www.sciencedirect.com/science/article/abs/pii/S0045653521004902
- Lei Zhao, Fei Pan, Arshad Mehmood, et al., Improved color stability of anthocyanins in the presence of ascorbic acid with the combination of rosmarinic acid and xanthan gum, Food Chem. (2021) https://www.sciencedirect.com/science/article/abs/pii/S030881462100323X
- Lili Zhang, Yan Li, Jing Gao, Phase behavior and physicochemical properties of thermoreversible aqueous biphasic systems composed of ionic liquids and deep eutectic solvents, CIESC Journal (2021) http://www.hgxb.com.cn/CN/10.11949/0438-1157.20201412 (此文没有恰当引用Molclus)
- Duomei Xue, Di Wu, Zeren Chen, et al., On Close Parallels between the Zintl-Based Superatom Ge9Be and Chalcogen Elements, Inorg. Chem. (2021) https://pubs.acs.org/doi/abs/10.1021/acs.inorgchem.0c03531
- Weilu Ding, Xing-Liang Peng, Zhu-Zhu Sun, et al., Ionic liquid decoration for hole transport improvement of PEDOT, Mater. Adv. (2021) https://pubs.rsc.org/en/Content/ArticleLanding/2021/MA/D1MA00040C#
- Zhengyan Zhao, Wei Liu, Yantao Shi, et al., An insight into the reaction mechanism of CO2 photoreduction catalyzed by atomically dispersed Fe atoms supported on graphitic carbon nitride, Phys. Chem. Chem. Phys. (2021) https://pubs.rsc.org/en/content/articlelanding/2021/cp/d0cp05570k/
- Chen Yang, Zitong Liu, Yanwei Li, et al., Electric field–catalyzed single-molecule Diels-Alder reaction dynamics, Sci. Adv., 7, eabf0689 (2021) https://advances.sciencemag.org/content/7/4/eabf0689
- Chenzhe Xu, Zheng Zhang, Suicai Zhang, et al., Manipulation of Perovskite Crystallization Kinetics via Lewis Base Additives, Adv. Funct. Mater. (2021) https://onlinelibrary.wiley.com/doi/abs/10.1002/adfm.202009425 (此文没有恰当引用molclus)
- Shuangyu Wang, Jiaqi Liu, Peng Cheng, et al., Application of an immobilized ionic liquid for the preparation of hydroxylamine via hydrolysis of cyclohexanone oxime, Z. anorg. allg. Chem. (2021) https://onlinelibrary.wiley.com/doi/abs/10.1002/zaac.202000370 (此文没有恰当引用molclus)
- Peipei Li, Menghang Li, Fan Zhang, et al., High-efficient nucleic acid separation from animal tissue samples via surface modified magnetic nanoparticles, Sep. Purif. Technol., 262, 118348 (2021) https://www.sciencedirect.com/science/article/abs/pii/S1383586621000502 (此文没有恰当引用molclus)
- Guoqing Wu, Ying Liu, Guangliang Liu, et al., Characterizing the electronic structure of ionic liquid/benzene catalysts for the isobutane alkylation, J. Mol. Liq. (2021) https://www.sciencedirect.com/science/article/abs/pii/S0167732221001379 (此文没有恰当引用molclus)
- Ming Lin, Xianwei Hu, Jiangyu Yu, et al., Raman spectroscopy and quantum theory calculations on complexes in the KF-AlF3-Al2O3 system, J. Mol. Liq. (2021) https://www.sciencedirect.com/science/article/abs/pii/S0167732220375097
- Tingting Zhu, Ping Ning, Zezhi Chen, Structures and spectroscopic properties of low-energy candidate structures for toluene-(H2O)n (n=1–10) clusters, J. Mol. Liq. (2021) https://www.sciencedirect.com/science/article/abs/pii/S0167732220374559
- Qi Liang Lu, Qi Quan Luo, Structures and Properties of CoB19+/0/– Clusters, ACS Omega (2020) https://pubs.acs.org/doi/full/10.1021/acsomega.0c05542
- Wen-Yu Zu, Jian-Wei Tang, Kun Hu, et al., Chaetolactam A, an Azaphilone Derivative from the Endophytic Fungus Chaetomium sp. g1, J. Org. Chem. (2020) https://pubs.acs.org/doi/abs/10.1021/acs.joc.0c02214
- Bin Shi, Jiancheng Yu, Tianyu Tang, et al., Calculation of the UV Spectrum and Electrophilic Reactive Sites of Fentanyl Molecule Based on the Density Functional Theory, Russ. J. Phys. Chem. A, 94, 2586 (2020) https://link.springer.com/article/10.1134/S0036024420120055
- Jia Han, Lei Wang, Hanhui Zhang, et al., Determinant Factor for Thermodynamic Stability of Sulfuric Acid–Amine Complexes, J. Phys. Chem. A (2020) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.0c07908
- Shasha Yang, Dewei Rao, Jingjing Ye, et al., Mechanism of transition metal cluster catalysts for hydrogen evolution reaction, Int. J. Hydrogen Energy (2020) https://www.sciencedirect.com/science/article/abs/pii/S0360319920341756
- Shasha Lv, Xiyu Li, Li Yang, et al., Azopyrazole-Based Photoswitchable Anion Receptor for Dihydrogen Phosphate Transport, J. Phys. Chem. A, 124, 9692 (2020) https://pubs.acs.org/doi/abs/10.1021/acs.jpca.0c08108
- Yingying Liu, Changyan Zhu, Chaoxia Wen, et al., The Al≡Al triple bond in Al2X5+ and Al2X62+(X=Li, Na) clusters with multiple alkali metals coordination, New J. Chem. (2020) https://pubs.rsc.org/en/content/articlelanding/2020/nj/d0nj04819d/
- Junwu Chen, Kun Dong, Lei Liu, Anti-electrostatic hydrogen bonding between anions of ionic liquids:A density functional theory study, Arxiv (2020) https://arxiv.org/ftp/arxiv/papers/2011/2011.07678.pdf
- Dan Yu, Di Wu, Jing-yao Liu, et al., Unveiling the potential of superalkali cation Li3+ for capturing nitrogen, Phys. Chem. Chem. Phys. (2020) https://pubs.rsc.org/en/content/articlelanding/2020/cp/d0cp03769a/
- Tianwei Mu, Yu Xi, Manhong Huang, Gang Chen, Search for optimal monomers for fabricating active layers in thin-film composite osmosis membranes by conceptual density functional theory, J. Mol. Model., 26, 334 (2020) https://link.springer.com/article/10.1007/s00894-020-04578-z
- Shuai Hou, Congcong Zhang, Bin Jiang, et al., Investigation of Highly Efficient and Reversible Absorption of SO2 Using Ternary Functional Deep Eutectic Solvents, ACS Sustainable Chem. Eng. (2020) https://pubs.acs.org/doi/abs/10.1021/acssuschemeng.0c05480
- Long Chen, Yu Huang, Yonggang Xue, et al., Atmospheric oxidation of 1-butene initiated by OH radical: Implications for ozone and nitrous acid formations, Atmos Environ., 244, 118010 (2021) https://www.sciencedirect.com/science/article/pii/S1352231020307433
- Yan Huang, Qian Zhang, Le-Wu Zhan, et al., Theoretical studies on oxadiazole-based layer stacking nitrogen-rich high-performance insensitive energetic materials, J. Mol. Model., 26, 298 (2020) https://link.springer.com/article/10.1007/s00894-020-04555-6
- Yifan Jiang, Zhenhang Wang, Zhigang Lei, Gangqiang Yu, Structural effects on thermodynamic behavior and hydrogen bond interactions of water–ionic liquid systems, Chem. Eng. Sci. (2020) https://www.sciencedirect.com/science/article/pii/S0009250920307181
- Huanfei Xu, Yi Kong, Jianjun Peng, et al., Comprehensive analysis of important parameters of choline chloride-based deep eutectic solvent pretreatment of lignocellulosic biomass, Bioresource. Technol. (2020) https://www.sciencedirect.com/science/article/abs/pii/S0960852420314838
- Dan Li, Yong Kang, Significantly promoted SO2 uptake by the mixture of N-methylated ethylene imine polymer and 1-ethyl-3-methylimidazolium tetrazolate, J. Hazard. Mater. (2020) https://www.sciencedirect.com/science/article/abs/pii/S0304389420320914
- Hongxin Qiu, Zherui Chen, Guanghui Wang, Effect of Dodecyl Trimethyl Ammonium Bromide on the Migration of Water Molecules in the Pores of Lignite: An Experimental and Molecular Simulation Study, ACS Omega (2020) https://pubs.acs.org/doi/full/10.1021/acsomega.0c04012
- Yuyang Liu, Chuanqun Liu, Jianjun Li, Comparison of Vitamin C and Its Derivative Antioxidant Activity: Evaluated by Using Density Functional Theory, ACS Omega (2020) https://pubs.acs.org/doi/full/10.1021/acsomega.0c04318
- Zeyu Liu, Tian Lu, Qinxue Chen, Intermolecular interaction characteristics of the all-carboatomic ring, cyclo[18]carbon: Focusing on molecular adsorption and stacking, Carbon (2020) https://www.sciencedirect.com/science/article/abs/pii/S0008622320309076
- Xinghua Liu, Wenhao Yuan, Jingsong Zhang, et al., Thermal decomposition of 1-hexene by flash pyrolysis: A study of initial decomposition mechanism, P. Combust. Inst. (2020) https://www.sciencedirect.com/science/article/abs/pii/S1540748920304922
- Hui Li, Qiuping Guan, Zishang Jia, Xueye Wang, Theoretical study of switching characteristics of molecular tweezers based on bis(Zn-salphen), J. Mol. Model., 26, 265 (2020) https://link.springer.com/article/10.1007/s00894-020-04527-w
- Qiujun Lu, Ting Huang, Jieqiong Zhou, Limitation-induced fluorescence enhancement of carbon nanoparticles and their application for glucose detection, Spectrochim. Acta A (2020) https://www.sciencedirect.com/science/article/abs/pii/S1386142520308726
- Bin Jiang, Shuai Hou, Luhong Zhang, et al., Ether-linked diamine carboxylate ionic liquid
aqueous solution for efficient absorption of SO2, Ind. Eng. Chem. Res. (2020) DOI: 10.1021/acs.iecr.0c02877 - Shizhuo Hu, Yi Wang, Wenjun Zhou, et al., Dielectric Properties of CF3SO2F/N2 and CF3SO2F/CO2 Mixtures as a Substitute to SF6, Ind. Eng. Chem. Res. (2020) DOI: 10.1021/acs.iecr.0c03401
- Yifan Jiang, Shuai Huang, Zhigang Lei, Ruinian Xu, Removal of Methyl Ethyl Ketone and Sec-butanol from Hydrogen by Absorption with Ionic Liquids, Ind. Eng. Chem. Res. (2020) DOI: 10.1021/acs.iecr.0c02208
- Haodan Yu, Bing Yang, Michael Gatheru Waigi, et al., The effects of functional groups on the sorption of naphthalene on microplastics, Chemosphere, 261, 127592 (2020) DOI: 10.1016/j.chemosphere.2020.127592
- J. Mol. Liq. (2020) DOI: 10.1016/j.molliq.2020.113747
- Org. Chem. Front. (2020) DOI: 10.1039/D0QO00458H
- ACS Omega (2020) DOI: 10.1021/acsomega.0c01375
- Beilstein Archives (2020) DOI: 10.3762/bxiv.2020.74.v1
- Sci. China Chem., 63, 361 (2020) DOI: 10.1007/s11426-019-9670-2
- J. Energy Chem., 52, 269 (2021) DOI: 10.1016/j.jechem.2020.04.065
- Energ. Fuel. (2020) DOI: 10.1021/acs.energyfuels.0c01030
- J. Phys. Org. Chem., 33, e4036 (2020) DOI: 10.1002/poc.4036
- ACS Appl. Mater. Interfaces, 12, 4002 (2020) DOI: 10.1021/acsami.9b20323
- Mater. Chem. Front., 4, 1507 (2020) DOI: 10.1039/D0QM00016G
- Phys. Chem. Chem. Phys. (2020) DOI: 10.1039/D0CP02449J
- Theor. Chem. Acc., 139, 39 (2020) DOI: 10.1007/s00214-020-2553-8
- Corrosion Science, 164, 108334 (2020) DOI: 10.1016/j.corsci.2019.108334
- J. Mater. Chem. C, 8, 219 (2020) DOI: 10.1039/C9TC04025K
- SAE Technical Paper 2020-01-0357 (2020) DOI: 10.4271/2020-01-0357
- J. Mol. Graph. Model., 92, 32 (2020) DOI: 10.1016/j.jmgm.2019.06.013
- J. Phys. Chem. A, 124, 2628 (2020) DOI: 10.1021/acs.jpca.0c00371
- Mater. Today Commun., 24, 101020 (2020) DOI: 10.1016/j.mtcomm.2020.101020
- J. Energy Chem., 44, 19 (2020) DOI: 10.1016/j.jechem.2019.09.009
- New J. Chem., 44, 8996 (2020) DOI: 10.1039/D0NJ01150A
- Phys. Chem. Chem. Phys., 22, 7193 (2020) DOI: 10.1039/D0CP00700E
- Environ. Sci.: Processes Impacts, 21, 1560 (2019) DOI: 10.1039/C9EM00254E
- J. Phys. Chem. A, 123, 10739 (2019) DOI: 10.1021/acs.jpca.9b08833
- J. Phys. Chem. C, 123, 29543 (2019) DOI: 10.1021/acs.jpcc.9b07695
- ACS Sustainable Chem. Eng., 7, 18174 (2019) DOI: 10.1021/acssuschemeng.9b05691
- Chem. Eur. J., 25, 9568 (2019) DOI: 10.1002/chem.201901460
- J. Chem. Phys., 150, 244305 (2019) DOI: 10.1063/1.5100733
- Org. Chem. Front., 6, 3917 (2019) DOI: 10.1039/C9QO01008D
- J. Mater. Chem. C, 7, 11005 (2019) DOI: 10.1039/C9TC03334C
- Org. Chem. Front., 6, 1619 (2019) DOI: 10.1039/C9QO00281B
- Int. J. Quantum Chem., 119, e25961 (2019) DOI: 10.1002/qua.25961
- Phys. Chem. Chem. Phys., 21, 18250 (2019) DOI: 10.1039/C9CP03081F
- ACS Sustainable Chem. Eng., 7, 8347 (2019) DOI: 10.1021/acssuschemeng.8b06822
- Inorg. Chim. Acta, 495, 118950 (2019) DOI: 10.1016/j.ica.2019.06.001
- J. Mol. Liq., 255, 176 (2018) DOI: 10.1016/j.molliq.2018.01.121
- Phys. Chem. Chem. Phys., 20, 23898 (2018) DOI: 10.1039/C8CP04600J
- Struct. Chem., 29, 1663 (2018) DOI: 10.1007/s11224-018-1143-z
- J. Mol. Liq., 269, 839 (2018) DOI: 10.1016/j.molliq.2018.08.109
- Int. J. Mod. Phys. B, 32, 1850073 (2018) DOI: 10.1142/S021797921850073X
- Eur. Phys. J. D, 72, 103 (2018) DOI: 10.1140/epjd/e2018-80776-8
- Mol. Phys., 116, 1871 (2018) DOI: 10.1080/00268976.2018.1459919
- Phys. Chem. Chem. Phys., 19, 28434 (2017) DOI: 10.1039/C7CP05610A
- J. Theor. Comput. Chem., 16, 1750058 (2017) DOI: 10.1142/S0219633617500584
- Chem. Phys. Lett., 663, 128 (2016) DOI: 10.1016/j.cplett.2016.10.006
- RSC Adv., 6, 55007 (2016) DOI: 10.1039/c6ra07597e
- J. Phys. Chem. A, 120, 4560 (2016) DOI: 10.1021/acs.jpca.6b05529
- 化工进展, 35, 2670 (2016) http://d.wanfangdata.com.cn/periodical/hgjz201609008